科学家开发出变形式碱基编辑新系统
来源:营养与健康所 2021-05-14 12:58
Nature Cell Biology在线发表了中国科学院上海营养与健康研究所研究员杨力课题组与合作者在碱基编辑研究领域发布的最新进展——Eliminating base-editor-induced genome-wide and transcriptome-wide off-target mutations。碱基编辑器(base
Nature Cell Biology在线发表了中国科学院上海营养与健康研究所研究员杨力课题组与合作者在碱基编辑研究领域发布的最新进展——Eliminating base-editor-induced genome-wide and transcriptome-wide off-target mutations。
碱基编辑器(base editor)主要是由两个主要的功能结构域组分组成,分别为识别基因组特定DNA序列定位的定位器(locator)和可对该特定基因组DNA序列(或者位点)进行编辑的效应器(effector)。其中,胞嘧啶碱基编辑系统主要以nCas9/sgRNA作为locater执行对编辑的识别和定位功能,以胞嘧啶脱氨酶APOBEC/AID作为effector执行胞嘧啶脱氨的功能并最终实现C-to-T的编辑。随着碱基编辑技术的不断开发,基因编辑效率和靶向范围显着提高,但在全基因组和/或全转录组水平仍存在大规模的脱靶效应,这限制了其在生物学和临床上的应用。
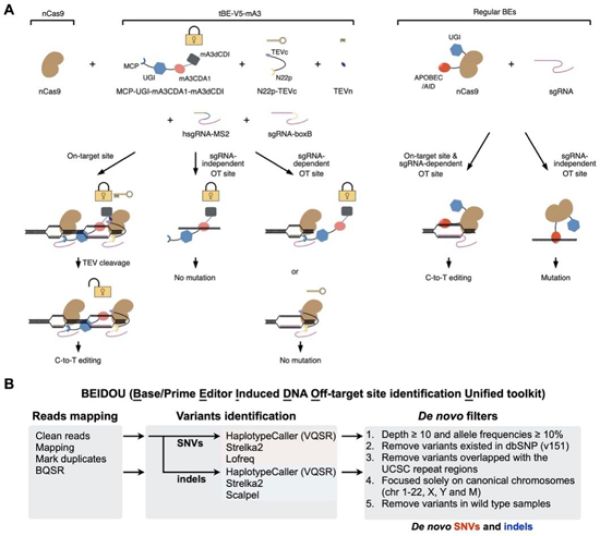
该研究中,研究人员首先发现了在胞嘧啶脱氨酶中天然存在的抑制序列结构域(deoxycytidine deaminase inhibitor,dCDI),并利用其开发出一系列可高效催化靶位点C-to-T编辑、抑制脱靶位点C-to-T编辑的新型碱基编辑系统,并依据其特性,命名为变形式碱基编辑系统(transformer base editors,简称tBEs)(图A)。结合高通量测序技术和计算生物学的研究方法,研究人员评估了tBE的编辑效率、脱靶效应和编辑产物纯度,结果显示,tBE碱基编辑系统可以在执行高效C-to-T编辑的同时,最大程度地减少由locator产生的Cas9/sgRNA依赖性脱靶效应(sgRNA-dependent off-target,OTsg)和effector产生的Cas9/sgRNA非依赖性单链DNA脱靶效应(sgRNA-independent single-stranded off-target,OTss)。此外,该研究还构建了在全基因组检测脱靶的计算分析流程BEIDOU(Base/Prime Editor Induced DNA Off-target site identification Unified toolkit,https://github.com/YangLab/BEIDOU)(图B),并结合全转录组RNA脱靶鉴定工具RADAR(RNA-editing Analysis-pipeline to Decode All twelve-types of RNA-editing events,https://github.com/YangLab/RADAR)(Wang et al. Cell Rep 2020),证明了tBE具有与本底水平类似的全基因组和全转录组突变效应,提示tBE可能不具有早期BE3的脱靶效应。另外,该研究成功地将tBE用于小鼠Pcsk9的编辑,通过引入终止密码子的方法降低了小鼠体内PCSK9的表达和LDL-C的水平。对编辑后小鼠进行全基因组和全转录组测序表明,tBE在in vivo水平同样仅产生与背景水平相一致的突变率。相关研究证明了tBE碱基编辑系统具有很好的特异性和安全性,提高了碱基编辑系统的应用价值,拓展了碱基编辑系统在点突变引起的疾病治疗中的应用前景。(生物谷Bioon.com)
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


