研究人员开发出基于单分子测序平台的scNanoHi-C技术,可精准检测单细胞高阶染色质互作
来源:网络 2023-09-18 10:45
真核生物基因的表达受到基因组中顺式作用元件的复杂调控。哺乳动物基因组中存在大量的顺式作用元件,例如:启动子、增强子、沉默子、绝缘子等等,其数量远远超过蛋白编码基因。
真核生物基因的表达受到基因组中顺式作用元件的复杂调控。哺乳动物基因组中存在大量的顺式作用元件,例如:启动子、增强子、沉默子、绝缘子等等,其数量远远超过蛋白编码基因。目前人类基因组中已知的顺式调控元件就有一百多万个,而蛋白编码基因只有大约两万个。遗传学研究也表明基因调控不仅仅是单个基因之间一对一的简单调控事件,而是以调控网络的形式发挥作用,不同的调控元件以及靶基因之间存在着复杂的相互作用。例如,一个基因的启动子可以整合来自多个增强子或者沉默子的调控作用,一个增强子元件也能够同时影响多个基因的表达1-3。随着三维基因组技术的发展,人们对基因表达调控相关的染色质构象已经有了一定的理解,但由于技术的限制,大部分研究都是集中在成对的相互作用(pair-wiseinteraction)上,而对于多个顺式调控元件同时与一个基因启动子之间的高阶相互作用(high-orderinteraction)的研究仍然比较有限。此外,多个基因组元件是如何通过三维基因组构象的变化同时参与基因表达调控的机制目前也尚不清楚。
近年来,为了探究更精准和全面的染色质互作情况,检测高阶染色质互作的技术也相继出现。然而,这些技术往往局限于基因组的特定位点,或是需要特殊的仪器设备。得益于三代测序平台(单分子测序平台)的日渐成熟,最近开发的基于牛津纳米孔技术(Oxford Nanopore Technology, ONT)的Pore-C方法4在检测染色质高阶相互作用方面表现出优异的性能,可以通过应用新的统计方法有效地分析全基因组中多个染色质位点之间高阶相互作用的协同性。
尽管上述这些基于大量细胞的研究方法能够有效地检测染色质的高阶相互作用,但它们无法解决细胞间的异质性问题,阻碍了它们在复杂组织器官样品中的应用。而现有的单细胞Hi-C(single-cell Hi-C,scHiC)技术受限于二代测序较短的读长(通常是双端总共300bp)也难以对染色质高阶相互作用进行检测。目前除了单细胞超分辨率成像以外,2022年开发的scSPRITE5是唯一一种可以在单细胞水平检测染色质高阶相互作用的测序方法。但是该方法更适用于远距离的间接染色质高阶相互作用,而对于与基因调控更相关的直接染色质高阶相互作用的检测能力很有限。此外,scHi-C的另一个挑战是很难平衡捕获细胞群体异质性所需的高通量(每次实验能够检测大量单细胞)与探索高分辨率3D基因组结构所需的高深度(每个单细胞中捕获大量染色质相互作用)之间的矛盾。因此,需要一种可扩展的scHi-C方法来剖析高阶染色质三维结构,并在单细胞水平上研究这些染色质高阶相互作用在不同生物过程中的协同调控机制。
为了应对这些挑战,北京大学生物医学前沿创新中心汤富酬课题组在Nature Methods上发表题为“scNanoHi-C: a single-cell long-read concatemer sequencing method toreveal high-order chromatin structures within individual cells”的文章。该研究在国际上率先使用单分子测序平台开发了一种基于邻近连接的单细胞染色质构象捕获方法,称为scNanoHi-C。该方法实现了在单细胞水平的高阶染色质相互作用检测,并且在通量上具有很好的灵活性,能够满足不同的实验需求。
在实验上,scNanoHi-C依次使用1%甲醛(FA)和1.5mM戊二酸二琥珀酰亚胺酯(DSG)孵育进行交联,以降低连接反应的随机噪音并兼顾对短程和长程染色质相互作用的高灵敏度检测。为了尽可能完整地保留单细胞中固定连接后的染色质三维结构信息,该研究设计了一种灵活的单细胞基因组长片段扩增方法。该方法使用两端具有相同接头的低浓度Tn5转座酶以提高DNA片段扩增长度和基因组覆盖度,并通过设计24种带有不同条码标签的Tn5酶结合后续PCR扩增中引入的条码标签共同控制测序的通量。通过这种方式,scNanoHi-C 能够在一次PromethION测序中对少至几个单细胞进行低通量、高覆盖度测序或者对数千个单细胞(最高可达24×96=2304个细胞)进行高通量、低覆盖度测序,可以根据实验需求灵活进行选择(图1)。
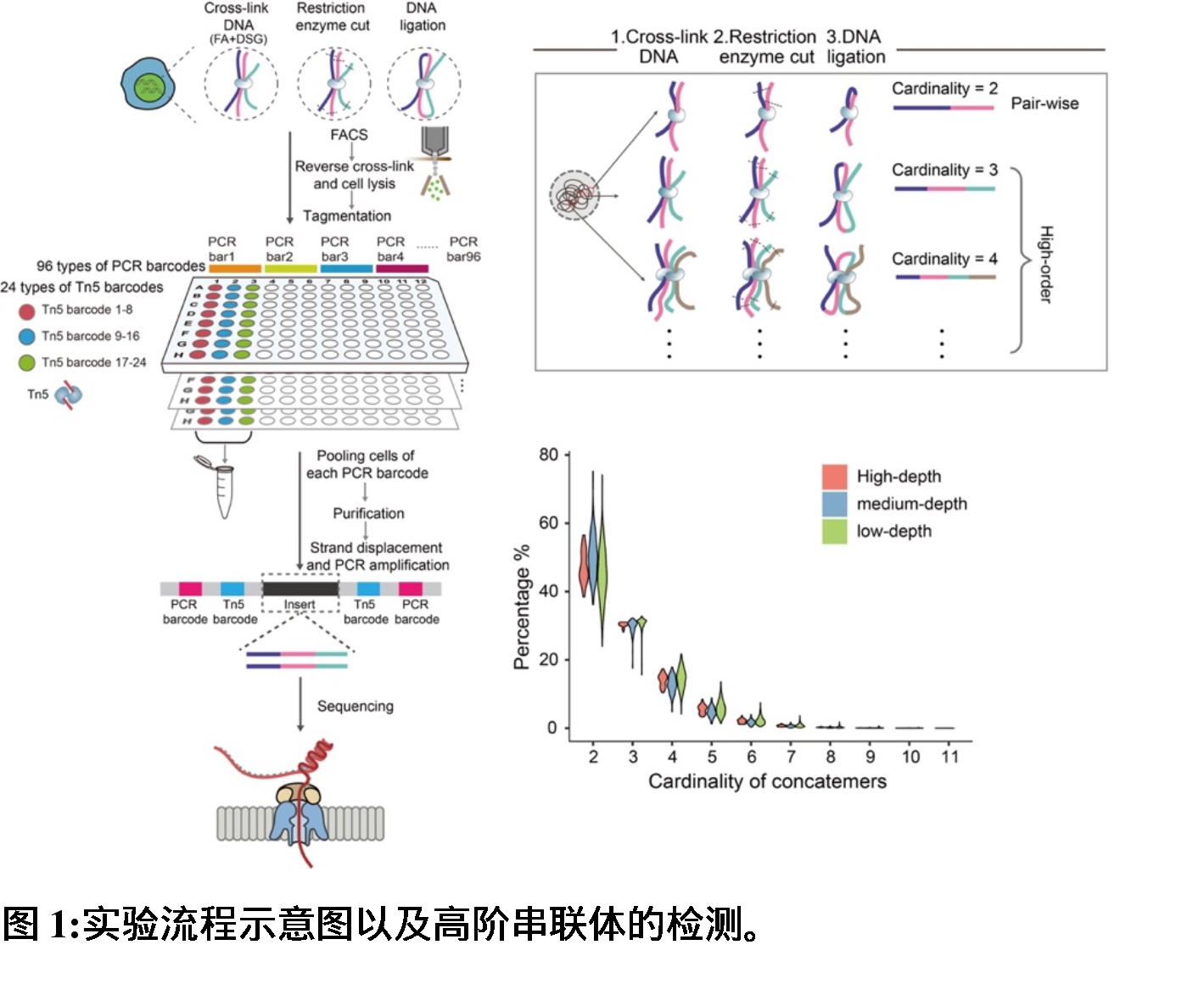
为了评估scNanoHi-C技术的可靠性,该研究首先将scNanoHi-C应用于正常二倍体的GM12878细胞系,并分别使用低深度(~0.2Gb/cell)、中等深度(~1Gb/cell)、高深度(~4Gb/cell)三种策略进行测序,并与基于二代测序平台的大量细胞原位Hi-C标准数据集进行比较,结果显示出很高的一致性。同时每个策略检测到的串联体(含有有效染色质相互作用的测序读段)中大约一半为高阶串联体(包含三个以上不同调控元件间的相互作用)。在这些高阶串联体中,大约58%是三联体,26%是四联体,其余为五联体以上的多联体(基数从5到11不等)。
接着该研究在多个方面对scNanoHi-C的应用进行了探索:
scNanoHi-C可以在单细胞水平上精准捕获染色质三维结构的异质性。
scNanoHi-C能够在单细胞水平检测各层级染色质结构特征,包括染色体领域(整条染色体,50Mb-200Mb尺度的结构特征)、A/B区室(常染色质区域与异染色质区域,5Mb-20Mb尺度的结构特征),以及拓扑关联结构域样结构(TAD-like,0.5Mb-5Mb尺度的结构特征)。同时,scNanoHi-C的单个染色质片段长度(单体长度,平均610bp)相较于传统基于二代测序平台的scHi-C(测序不超过150bp)显著提高,这大大增加了其在染色质相互作用对中捕获到单核苷酸多态性(SNP)位点的机会,能够在二倍体细胞中直接判定单倍型的单体比例由原来二代测序平台的大约9%提高到了25%。因此,scNanoHi-C也可用于有效地重建单个二倍体细胞的基因组三维构象。同时,利用单细胞A/B 区室化值(single-cell A/B compartment value, scA/B value),scNanoHi-C对GM12878、HG002 和K562三种人类细胞系进行了聚类分析,能够在单细胞精度准确将三种细胞分开,并识别了细胞类型间的染色质差异区室化区域。此外,scNanoHi-C也能够准确地检测每个单细胞的基因组拷贝数变异(CNV)特征。分析结果表明,scNanoHi-C准确地捕获了GM12878细胞培养过程中产生的非整倍体亚克隆以及K562细胞的拷贝数变异。同时,scNanoHi-C也可应用于结构变异的检测,如准确检测出了K562细胞中BCR-ABL1和NUP214-XKR3的基因融合事件(染色体易位事件)。

scNanoHi-C能够在单个细胞中准确鉴定高阶染色质相互作用。
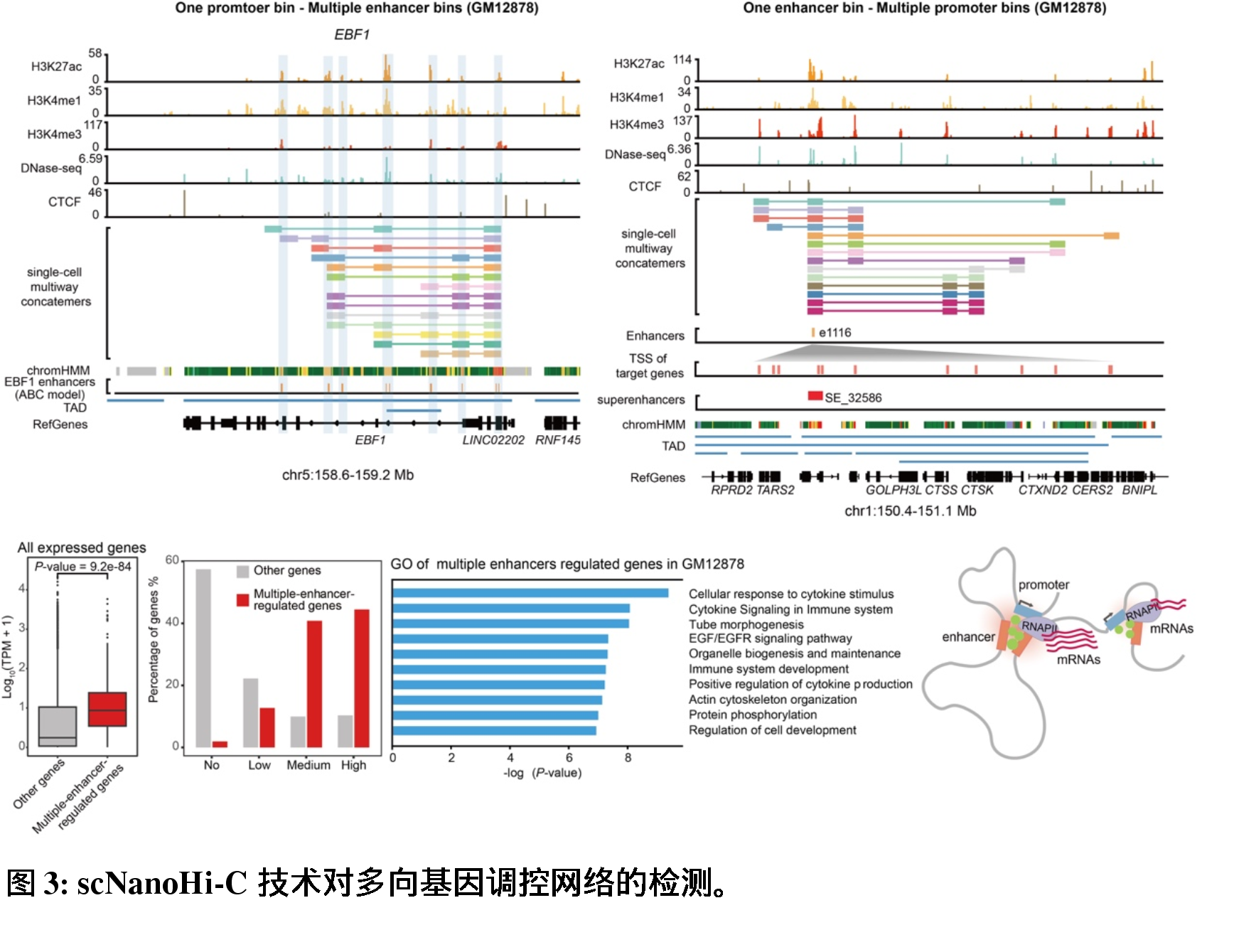
该研究在GM12878 细胞数据集中,使用scNanoHi-C得到的单细胞高阶串联体信息结合ABC模型(Activity-by-contacts model)6预测的增强子-启动子(E-P) 相互作用关系共同鉴定了增强子-启动子高阶相互作用。通过这种方式,该研究首次在单个细胞中以20kb的分辨率直接观察到1097个基因的单个启动子能够与多个增强子同时发生相互作用,表明这些基因可能同时受到多个增强子的调控。这些受到高阶调控的基因主要富集在与GM12878这种B淋巴细胞的功能相关的免疫信号通路上,并且通常表现出更高的表达水平。特别地,这些基因中还包括一些B细胞谱系特异性转录因子如EBF1以及EBV超级增强子相关基因如MIR155HG、IKZF3和ETS1等。这些结果表明,多个增强子的协同调控可能是确保关键基因高水平稳健表达的一种潜在机制。通过类似的方法,该研究还在单个细胞中鉴定出了1422个能够与多个启动子同时发生相互作用的增强子。此外,该研究发现部分高阶基因调控作用能够在多个单细胞中被检测到,这可能与细胞中频繁使用的关键转录程序有关,后续可以通过发展基于富集策略的具有更高分辨率的Hi-C技术进行进一步的深入研究。
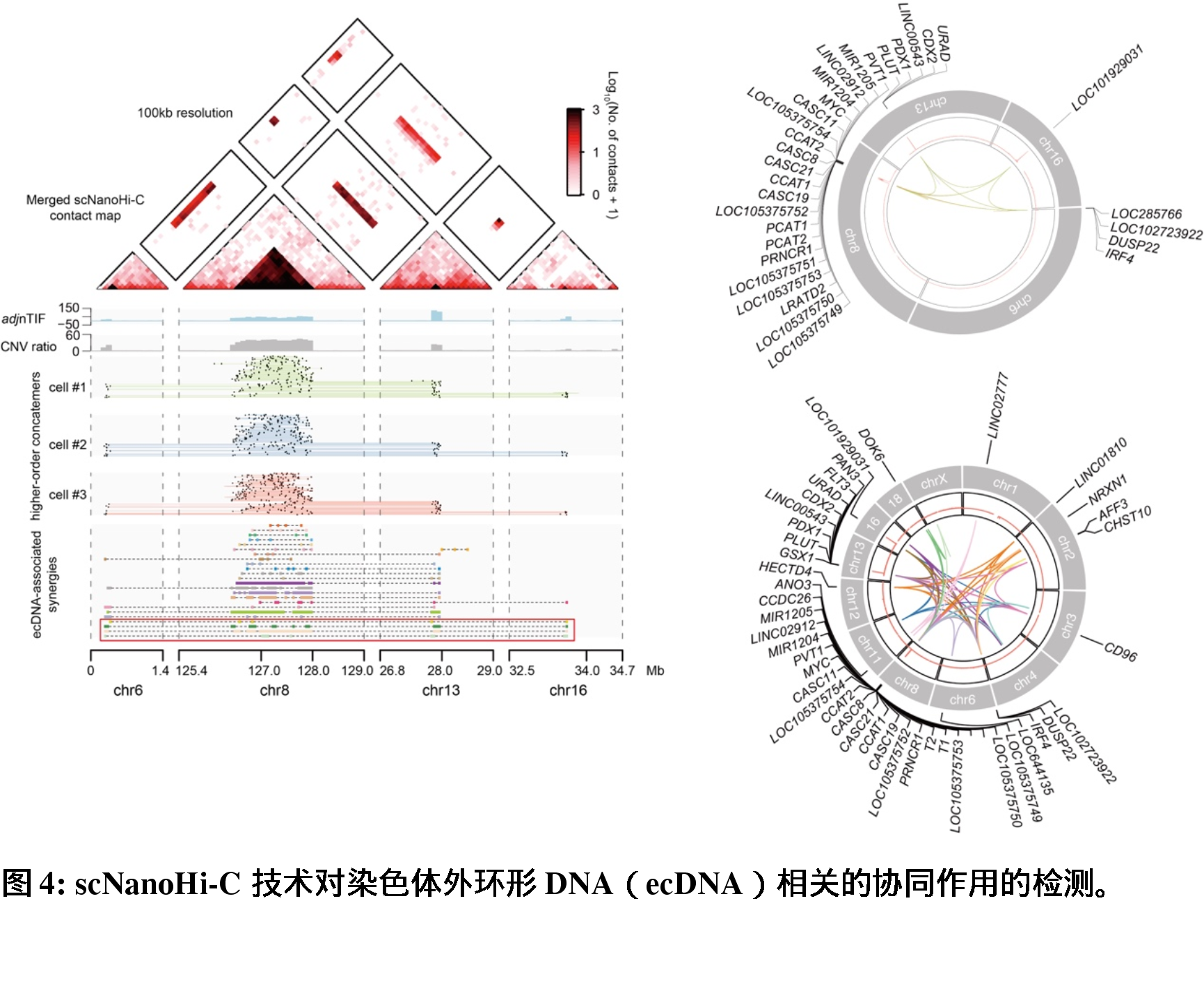
scNanoHi-C能够揭示不同基因组区域之间的协同调控关系以及染色体外环形DNA与线性基因组间的复杂相互作用。
倾向于形成高阶相互作用的一组基因组位点称为“基因组协同调控区域”。该研究针对scNanoHi-C的数据特点对鉴定基因组协同调控区域的算法进行了优化,并将该算法运用到GM12878细胞活跃启动子和增强子的集合中,在全基因组范围内共鉴定出了917组增强子-启动子协同调控区域。其中,大约20%(187/917)的协同调控区域包含来自不同染色体的基因组位点(提示不同染色体之间的反式相互作用)。这些协同调控区域在活跃转录的基因组区域、淋巴细胞特异性转录因子和染色质环相关因子(CTCF等)的结合位点区域中高度富集。此外,在917个协同调控区域中,有167个被发现与GM12878细胞特异性的超级增强子有关。接着,该研究将scNanoHi-C运用到携带大量染色体外环形DNA(ecDNA)的COLO320DM人类结直肠癌细胞系中,检测到了染色体外环形DNA与线性基因组(染色体内的基因组)之间存在广泛的染色质高阶相互作用,并且首次在单个细胞中观察到四个主要的染色体外环形DNA的基因位点之间存在复杂的高阶相互作用。这些结果表明,染色体外环形DNA可能通过建立复杂的高阶染色质三维结构来驱动癌基因的过量表达。
scNanoHi-C能够高效辅助单细胞基因组从头组装。
在可用细胞数量有限的情况下,该研究表明使用scNanoHi-C辅助单细胞基因组(single-cell whole genome sequencing,scWGS)从头组装7可以大幅度提高组装质量。例如,使用20个单细胞的基因组长读长测序数据和12个单细胞的scNanoHi-C数据组装的人类基因组支架(scaffold)的NG50要优于使用30个单细胞的基因组长读长测序数据直接组装的效果(2.49 Mb vs. 1.34 Mb)
总之,scNanoHi-C具有很好的可扩展性和灵活性,在一次测序中可对少至几个单细胞或多达数千个单细胞进行染色质三维结构测序,并且实验流程相对简单、易于操作,仅需要基本的PCR仪等分子生物学设备,适合于各种生物学实验室使用。scNanoHi-C还是一种强大且多功能的工具,可用于在单细胞分辨率准确区分细胞类型、对单个二倍体细胞进行高效单倍型分型、检测单个正常细胞和肿瘤细胞中的基因组拷贝数变异和各种复杂结构变异以及高效辅助单细胞基因组从头组装。更重要的是,scNanoHi-C首次实现了在单个细胞中在全基因组水平对增强子-启动子的高阶直接相互作用的检测,在单个细胞中准确鉴定了高阶基因调控事件,同时能够对复杂的染色体外环形DNA与线性基因组间的高阶相互作用进行精准检测。总之,scNanoHi-C显示了单细胞长读长Hi-C测序技术在分析由高阶染色质三维结构介导的不同细胞间基因调控异质性方面的潜力,为将来进一步研究发育和疾病进展过程中高阶染色质结构变化机制,揭开基因组中各种复杂调控关系中的“暗物质”奠定了坚实的基础。
北京大学生物医学前沿创新中心、前沿交叉学科研究院生命科学联合中心博士生李文、生命科学学院博士生卢健森为该论文的共同第一作者,汤富酬为该论文通讯作者。该研究得到了国家自然科学基金基础科学中心项目、北京未来基因诊断高精尖创新中心、昌平实验室的资助,北京大学高通量测序平台以及北京大学“北极星”高性能计算平台的协助与支持,北京大学邢栋课题组为本研究提供了重要的帮助。
参考文献:
1、Hafner, A. & Boettiger, A. The spatial organization of transcriptional control. Nat Rev Genet, doi:10.1038/s41576-022-00526-0 (2022).
2、Oudelaar, A. M. & Higgs, D. R. The relationship between genome structure and function. Nat Rev Genet22, 154-168, doi:10.1038/s41576-020-00303-x (2021).
3、Furlong, E. E. M. & Levine, M. Developmental enhancers and chromosome topology. Science361, 1341-1345, doi:10.1126/science.aau0320 (2018).
4、Deshpande, A. S. et al. Identifying synergistic high-order 3D chromatin conformations from genome-scale nanopore concatemer sequencing. Nat Biotechnol40, 1488-1499, doi:10.1038/s41587-022-01289-z (2022).
5、Arrastia, M. V. et al. Single-cell measurement of higher-order 3D genome organization with scSPRITE. Nature Biotechnology40, 64-73, doi:10.1038/s41587-021-00998-1 (2021).
6、Fulco, C. P. et al. Activity-by-contact model of enhancer-promoter regulation from thousands of CRISPR perturbations. Nat Genet51, 1664-1669, doi:10.1038/s41588-019-0538-0 (2019).
7Xie, H. et al. De novo assembly of human genome at single-cell levels. Nucleic Acids Res50, 7479-7492, doi:10.1093/nar/gkac586 (2022).
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


