PNAS丨谭蔚泓、韩达合作解密sgc8c DNA适体的复杂三维结构和功能优化
来源:生物探索 2024-07-28 14:28
研究结果揭示了sgc8c DNA适体形成精密复杂的三通结构,它能巧妙地从不同区域招募十余个核苷酸汇聚于三通中心构成其“结构-功能”的关键位点,这些信息有效地指导了适体的功能优化。
核酸的三维结构和构象动态对其发挥精密的生物功能至关重要。核酸适体通过折叠成特定的三维结构实现对蛋白和小分子等标靶的高亲和力、高特异性识别。靶向膜蛋白的核酸适体在疾病检测和靶向治疗等领域展现出巨大潜力。通常,利用冷冻电镜、X射线晶体衍射和核磁共振(NMR)技术测定蛋白-核酸适体复合物的三维空间结构是研究适体识别机制和功能优化的最直接策略。然而,对于具有较大构象动态性的蛋白和核酸适体,往往不易获取准确的复合物结构信息。
生物大分子结构预测模型如AlphaFold 3、RoseTTAFoldNA等在RNA、RNA-蛋白复合物结构预测性能上有所提升,但对DNA复杂三维结构预测表现欠佳。这是因为相较于RNA,DNA形成长程三级相互作用(long-range tertiary interactions)的能力较弱,导致Protein Data Bank (PDB) 数据库中DNA复杂三维结构的数量极少。因此,如何在缺乏复合物结构信息的情况下研究重要DNA适体的识别机制和功能优化,是领域内亟待解决的关键问题。
通过分析PDB数据库中蛋白-DNA适体复合物结构,作者发现DNA适体通常以碱基配对区域作为“支架”以维持三维空间折叠,而利用非稳定配对区域的核苷酸作为结合位点。若与蛋白作用仅引起核酸适体在其结合位点或附近发生局部构象变化,而不破环原本的三维折叠,即可通过监测NMR化学位移扰动(CSPs)来探测核酸适体的结合位点,从而指导其功能优化。该思路也被Bhattacharya等人用于生物大分子的定向进化(Nature, 2022, 610, 389)。在此基础上,仅解析核酸适体的三维结构则是有意义的。Borggrafe等人利用NMR实验捕捉碱基配对信息以更好地约束构象动态性较高的区域,从而成功解析10-23 DNAzyme的溶液结构(Nature, 2022, 601, 144)。这些展现出NMR技术在研究生物大分子互作、功能核酸结构和定向进化方面的巨大优势。
受上述启发,中国科学院杭州医学研究所谭蔚泓院士/韩达研究员/郭沛副研究员团队提出了一种简单高效的NMR技术思路,在无需获取蛋白-核酸适体复合物结构的条件下探索核酸适体的结合机制和功能优化,相关工作以Structure-based investigation of a DNA aptamer targeting PTK7 reveals an intricate 3D fold guiding functional optimization为题发表在PNAS上。

Sgc8c是通过Cell-SELEX技术获得的靶向白血病细胞的DNA适体,其分子靶标为酪氨酸蛋白激酶7(PTK7)。PTK7是一种无催化活性的跨膜蛋白,在多种肿瘤细胞中高表达。鉴于sgc8c对靶蛋白/细胞的高亲和力和高特异性,它被广泛应用于疾病检测和靶向治疗。然而,sgc8c执行精密功能的结构基础尚不清楚,导致其功能优化受到限制。作者以sgc8c为研究对象,通过NMR CSPs和定点突变实验确定了sgc8c结合PTK7的关键位点,测定了sgc8c的三维空间结构,发现其中重要的长程三级相互作用,并基于结构-功能关系设计sgc8c优化体,显著提升了适体的热稳定性、生物稳定性以及蛋白/细胞靶标结合亲和力。
Sgc8c二级结构和关键结合位点:在模拟Cell-SELEX的盐离子和温度条件下,作者分析sgc8c NOESY核磁谱图中的亚氨基质子(G H1和T H3)信号区,捕捉到C3∙G40、G9∙C13、C10∙G12、C18∙G36、C19∙G35、G20∙C34、T2∙A41、T4∙A39、A5∙T38和A6∙T37共10对Watson-Crick碱基配对,确定了sgc8c的二级结构,包含P1、P2、P3三个配对区(图1A-B)。向sgc8c滴定PTK7后,10个亚氨基质子信号依旧存在(图1C),说明sgc8c在结合PTK7后仍能维持原本的三维空间折叠。利用滴定PTK7前后sgc8c G H1、T H3和T H7信号的化学位移变化绘制了CSPs图,其中G9 H1、G12 H1、T38 H3、G36 H1显示出相对较大的CSPs(图1D),指示这些核苷酸可能处于结合位点。通常来说,生物大分子的关键功能区域(如结合位点)对残基突变较为敏感。作者将CSPs较大的G9∙C13、C10∙G12、A5∙T38、C18∙G36进行突变,采用SPR和细胞流式测定适体与PTK7蛋白和CCRF-CEM细胞的结合,最终确定P2区的核苷酸(即G9∙C13、C10∙G12和T11)是sgc8c的关键结合位点和重要功能元件。
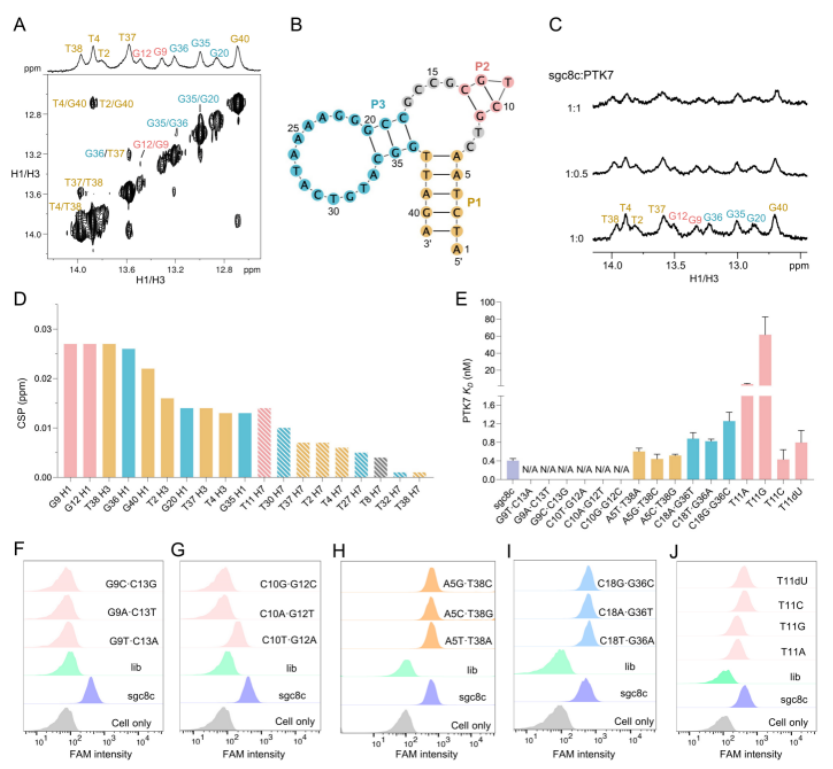
图1. sgc8c的二级结构和关键结合位点(Credit: PNAS)
Sgc8c三维空间结构:鉴于sgc8c在结合PTK7前后仍能维持其三维空间折叠,作者测定了sgc8c的溶液核磁结构(图2),揭示sgc8c折叠成复杂的三通(three-way junction)结构,包含P1区(核苷酸# 1-6、37-41)、P2区(核苷酸# 9-13)和P3区(核苷酸# 18-36)、J1连接区(核苷酸# 7-8)和J2连接区(核苷酸# 14-17)。有趣的是,sgc8c的P1和P3区形成跨度约53 Å的同轴堆叠,几乎呈共线排列。Passalacqua等人近期报道的lettuce DNA适体结构也有此特点(Nature, 2023, 618, 1078)。这种长跨度的同轴堆叠在RNA三通和四通结构中多见,而在DNA中却很少见。此外,作者还发现Mg2+对维持sgc8c三通结构和功能有重要作用。
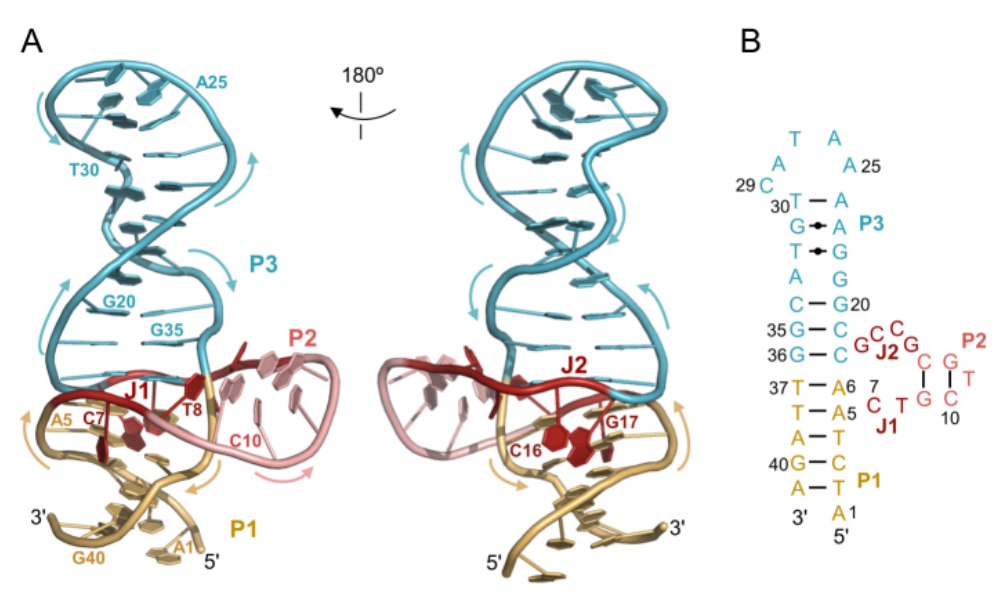 图2. Sgc8c的三维空间结构(PDB ID: 8Y0F)(Credit: PNAS)
图2. Sgc8c的三维空间结构(PDB ID: 8Y0F)(Credit: PNAS)
Sgc8c中的长程三级相互作用:作者在sgc8c三维结构中发现了DNA中少见的长程三级相互作用,例如C7与A39/T38形成氢键、G17与C3形成氢键。将C7或G17替换成缺碱基位点(dS)破坏了适体的三维空间折叠,丧失了对靶蛋白/细胞的结合能力。为了进一步确定C7或G17长程氢键作用的重要性,作者分别将C7突变成尿苷dU(缺少N4氨基)、G17突变成2-氨基嘌呤2-AP(缺少N1亚氨基)、G17突变成黄嘌呤X(缺少N2氨基),SPR结果显示G17X与PTK7无结合,C7dU、G17-2AP对PTK7的结合能力显著减弱,说明长程三级相互作用对sgc8c的结构和功能至关重要。
结构-功能关系指导的sgc8c优化策略:上述结果表明维持sgc8c结构和功能的重要核苷酸包括:构成结合位点的P2区、参与长程三级相互作用的C7和G17、形成重要碱基堆积的C19∙G35和G20∙C34,值得一提的是,sgc8c可将不同区域的十多个碱基募集在三通结构中心,构成其关键位点,这在DNA结构中非常罕见。Sgc8c关键位点中的核苷酸不耐受突变(图1,3),因此优化sgc8c应考虑对P1底部和P3顶部进行改造。
为提升适体的热稳定性和靶标结合亲和力,作者将P3顶部的AATA四核苷酸环(#25-28)替换为GAA三核苷酸环,同时保留或删除翻转在外侧的C29。GAA25-28和GAA25-29突变体能够维持原本的三维空间折叠,它们的Tm值较sgc8c分别升高约7和11 °C,其中GAA25-28表现出更强的蛋白结合亲和力,并在37 °C条件下与CCRF-CEM细胞结合的KD达到pM级别。为增强适体的生物稳定性,作者首先将P1末端的2至4对碱基替换为镜像核苷酸,得到L1-2、L1-3和L1-4突变体。L1-3 和L1-4虽能够抵抗酶切,但它们与蛋白/细胞的结合亲和力显著下降,这是因为镜像核苷酸破坏了C3和A39参与的重要长程氢键(图3)。为保护这些氢键,作者在P1末端延长2至3对镜像核苷酸,获得的L+2bp和L+3bp均有出色的抗酶切能力,且与蛋白/细胞的结合亲和力同sgc8c相当。最后,作者整合了GAA25-28变体和L+2bp、L+3bp修饰,获得sgc8c-x1和sgc8c-x2的优化适体。相较于sgc8c,优化后的适体表现出更高的热稳定性,更优的靶标结合亲和力、更强的抗核酸酶切能力和血清稳定性,可作为极具生物医学应用潜力的核酸分子工具。
该工作提出一种简单高效的NMR技术思路,用于在缺乏复合物三维结构信息的情况下探索适体的识别机理和功能优化。研究结果揭示了sgc8c DNA适体形成精密复杂的三通结构,它能巧妙地从不同区域招募十余个核苷酸汇聚于三通中心构成其“结构-功能”的关键位点,这些信息有效地指导了适体的功能优化。该工作为研究靶向膜蛋白的核酸适体结构与功能提供了新思路和技术路线,同时揭示了长程三级相互作用对塑造DNA分子复杂结构和精密功能的关键作用。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


