Nature子刊:西湖大学王寿文/李莉开发了首个基于表观突变、无需基因编辑的谱系追踪工具!
来源:iNature 2025-01-21 10:06
MethylTree 开启了在人类及更广泛领域进行高分辨率、非侵入性及多组学的谱系追踪的大门。
体内谱系追踪在揭示组织发育和稳态的基本原理方面具有巨大的潜力。然而,目前人类谱系追踪依赖于极其罕见的体细胞突变,其具有有限的时间分辨率和谱系准确性。
2025年1月16日,西湖大学王寿文、李莉共同通讯在Nature Methods在线发表题为“High-resolution, noninvasive single-cell lineage tracing in mice and humans based on DNA methylation epimutations”的研究论文,该研究开发了一种用于谱系追踪的新型计算工具——MethylTree,该工具无需基因编辑,即可在小鼠和人类中精准地、以多组学的方式实现高分辨率、非侵入性的单细胞谱系追踪。
在这里,研究人员基于DNA甲基化的频繁表型突变开发了一种通用谱系追踪工具,通过MethylTree实现。使用具有已知谱系和表型标签的单细胞全基因组DNA甲基化数据集,MethylTree跨不同细胞类型、发育阶段和物种以接近100%的准确度重建了谱系历史。在小鼠和人类血液中证明了基于表突变的单细胞多体谱系追踪,其中甲基树再现了造血中的分化层级。将MethylTree应用于人类胚胎,揭示了四细胞阶段的早期命运承诺。在天然小鼠血液中,研究人员鉴定了约250个造血干细胞克隆。MethylTree为高分辨率、非侵入性和多染色体谱系追踪打开了大门。

在过去的十年中,通过遗传操作追踪模式生物的谱系历史已经有了很大的改进。高分辨率谱系追踪可以通过用诱导的和可遗传的DNA突变标记单个细胞来实现,这可以在以后通过单细胞测序来描绘。最近开发了DARLIN,这是一种高效的谱系追踪小鼠模型,可以在规定的时间窗内诱导产生约1018个不同的谱系条形码。这些最新谱系追踪工具的应用揭示了关于细胞命运选择、细胞迁移动力学、癌症进化和克隆记忆的重要见解。
相比之下,由于基因操作被禁止,人类的血统追踪还不太发达。人类的细胞谱系可以从基因组的体细胞突变中推断出来。然而,这种方法需要单细胞衍生集落的全基因组DNA测序,这是低通量的,并且不提供细胞状态信息。虽然线粒体DNA (mtDNA)突变可用于以更高的产量追踪细胞谱系,但它们经历复杂的遗传和选择过程,可能导致谱系准确性差。由于极慢的体细胞突变(每个细胞分裂约109个核苷酸),这些方法可能无法在短得多的时间尺度(如几天)内解析谱系。非常需要开发一种替代的非侵入性谱系追踪方法,其提供高的“时间”谱系分辨率,实现接近100%的准确性,并且还与单细胞多组学分析兼容。
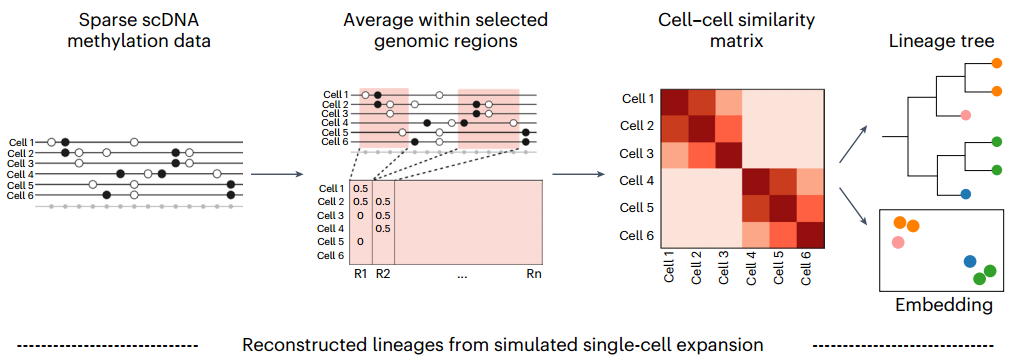
MethylTree的工作流程示意图(图源自Nature Methods)
在哺乳动物中,DNA甲基化主要发生在CpG二核苷酸的胞嘧啶残基上,由于表位突变的发生率约为0.001/CpG位点/分裂,这种甲基化会随着时间的推移而改变。最近在造血干细胞中发现,克隆记忆在DNA甲基化中稳定存在至少几个月,但在染色质可及性或基因表达中不存在。这激发了研究人员开发一种基于DNA甲基化表型突变的通用谱系追踪工具的极大兴趣。以前,在不同的环境中观察到克隆特异性DNA甲基化模式。大量甲基化数据已被用于追踪单个等位基因的进化并推断单细胞转换率。表突变已被用来推断单细胞谱系,主要是在癌症背景下。然而,由于缺乏来自中性标签的实际谱系的基准,这些推论并不令人信服。此外,已知癌细胞具有强烈的基因组不稳定性和高度异常的DNA甲基化。因此,从正常细胞的表型突变推断谱系仍然是一个巨大的挑战。
在这项研究中,研究团队开发了一种计算方法——MethylTree,该方法基于 DNA 甲基化上的频繁表观突变,利用具有已知谱系和表型标签的单细胞全基因组 DNA 甲基化数据集,以近乎 100% 的准确率推断不同细胞类型、发育阶段和物种之间的谱系关系。总的来说,MethylTree 开启了在人类及更广泛领域进行高分辨率、非侵入性及多组学的谱系追踪的大门。
参考消息:
https://www.nature.com/articles/s41592-024-02567-1#Sec40
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


