Nature子刊:腾讯AI Lab提出蛋白质相互作用研究AI模型
来源:AI Lab 2023-04-04 11:33
该研究将深度学习领域的层次图学习技术引入蛋白质相互作用(PPI)研究,提出了一种双视图层次图学习模型(HIGH-PPI),模型被证明在 PPI 的研究中具有更高的预测准确性和更好的可解释性。
蛋白质相互作用(PPI)可以说是人体最重要的分子事件之一,事关人体的生长发育、新陈代谢,是疾病治疗干预的重要来源,PPI 的失调会导致癌症等疾病的发生,因而该领域也是医药领域关注的研究热点。
为了更好地预测和解读 PPI,并深入挖掘相关分子的信息,2023 年 3 月,腾讯 AI Lab 联合香港科技大学、中国科学院大学相关团队,在 Nature Communications 期刊发表了题为:Hierarchical graph learning for protein–protein interaction 的研究论文。
该研究将深度学习领域的层次图学习技术引入蛋白质相互作用(PPI)研究,提出了一种双视图层次图学习模型(HIGH-PPI),模型被证明在 PPI 的研究中具有更高的预测准确性和更好的可解释性。

人类蛋白质组学数据分析表明,人体的 PPI 网络涉及约 65 万个相关接触位点。处理这样量级的数据,人工智能的效率要明显高于人类,相关技术引入可以更加高效、准确地帮助人们找出重要信息,促进靶向药物的开发和癌症等疾病的治疗。
这项研究是腾讯 AI Lab 对 PPI 层次问题建模的首次尝试,得到了审稿人的高度评价,被视为生物信息领域一项富有潜力的创新贡献,文章也得到了学界的认可,被选入 Nature Communications 年度编辑精选文章合集。
引入深度学习技术
首次提出模拟自然PPI层次结构的模型
过去的十年,为了高效且低成本地绘制出人类蛋白质相互作用组,研究领域越来越多地使用计算方法来自动预测 PPI,其中就包括深度学习的引入。但是,此前尚未出现用于模拟自然 PPI 层次结构的模型。
在 HIGH-PPI 模型中,腾讯 AI Lab 研究团队创建了一种层次图,包含蛋白质外顶视图和蛋白质内底视图。顶视图描述蛋白质之间的相互作用,每种蛋白质就是一个节点,蛋白质之间的相互作用就是图的边;而底视图描述每种蛋白质内部的信息,关键氨基酸或残基组合就是图的节点,物理位置相邻的残基以边相连。
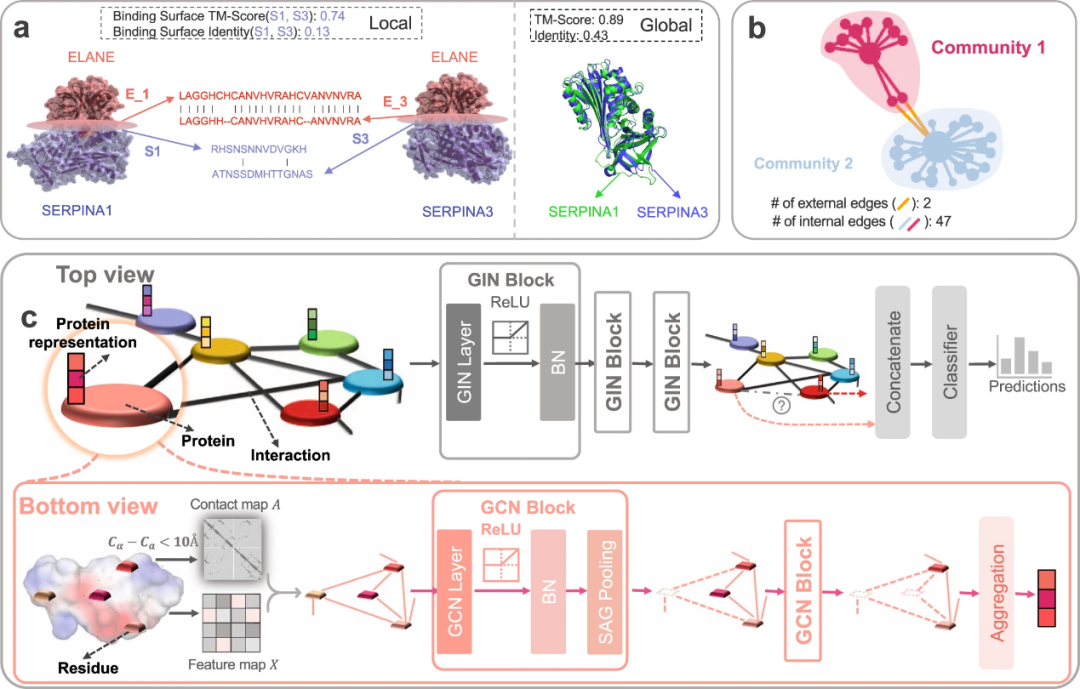
图1:蛋白质结构和网络结构都对PPI的准确预测至关重要。(a)蛋白质序列通常能提供关于PPI的细节信息,但它也可能导致PPI的预测准确性降低,例如不具备序列相似性的两个蛋白质(SERPINA1、3)可能和同一蛋白质(ELANE)在相同作用界面发生PPI;(b)包含网络结构信息的PPI,将蛋白质区分为不同的社区,社区内的蛋白质之间存在密集的交互,而社区之间通常只有微弱的连结;(c)HIGH-PPI具备双视图层次,顶视图包含网络结构信息,底视图包含蛋白质结构信息。
此前,业内学者大多关注 PPI 的外部层次(交互作用组学)或内部层次(蛋白质组学),并未考虑 PPI 本身的层次结构。
HIGH-PPI 模型受到生物学家研究方法的启发,使用两个图神经网络(GNN), 从两个视角分别进行学习。通过具体案例和统计实验,研究发现在端到端模型中,两个层次之间存在相互增益关系,缺失任一层次的结构信息都将损害机器学习模型的性能。
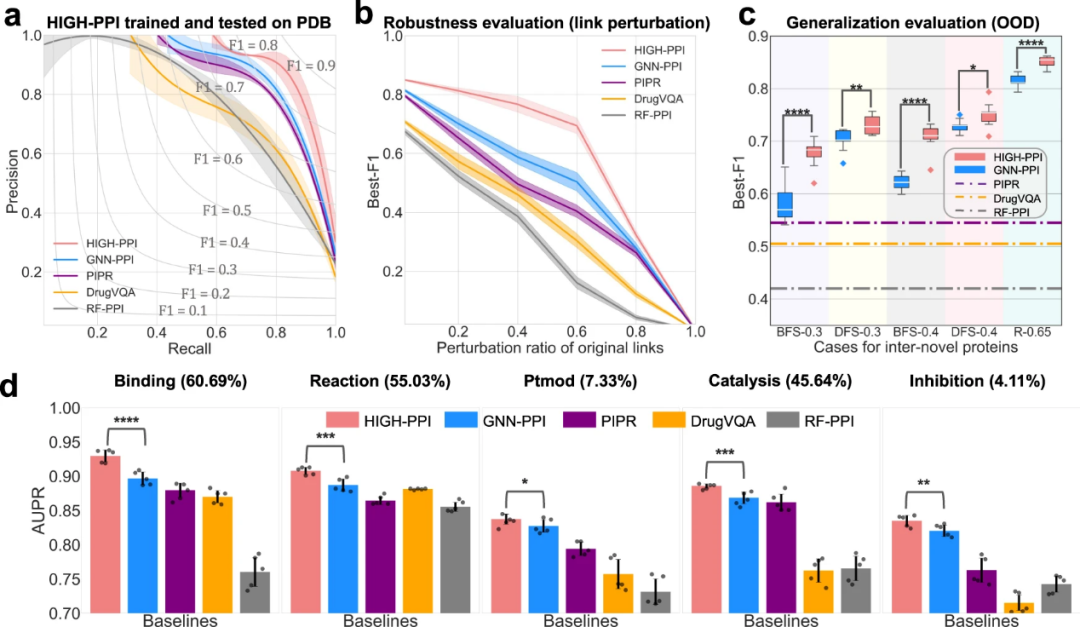
图2:将HIGH-PPI(红色)与四种主流模型 GNN-PPI、PIPR、DrugVQA和RF-PPI对比。(a)PPI预测精确率-召回率曲线;(b)PPI网络扰动下模型鲁棒性测试;(c)分布外场景下模型泛化性测试;(d)各PPI类别下精度测试。
据介绍,模型的架构设计主要有两个层面的考虑:
首先,在蛋白质组学层面,蛋白质序列信息通常能提供关于PPI的细节信息,但是在预测PPI方面准确率较低。例如,两种蛋白 SERPINA1、SERPINA3 分别与 ELANE 蛋白在同一局部位置发生相互作用,SERPINA1、3之间结构相似性较高,但是序列相似性较低(见图1a)。在这种情况下,结构信息对预测PPI很重要,仅依靠序列信息难以让模型作出准确的预测。
其次,在交互作用组学层面,模型关注 PPI 网络结构,倾向于将蛋白质划分为不同的社区,社区内的蛋白质之间存在密集的交互,而社区之间通常只有微弱的连结(见图1b)。先前研究已经证明,PPI网络的结构属性能够预测缺失的链接,发现未知的PPI。
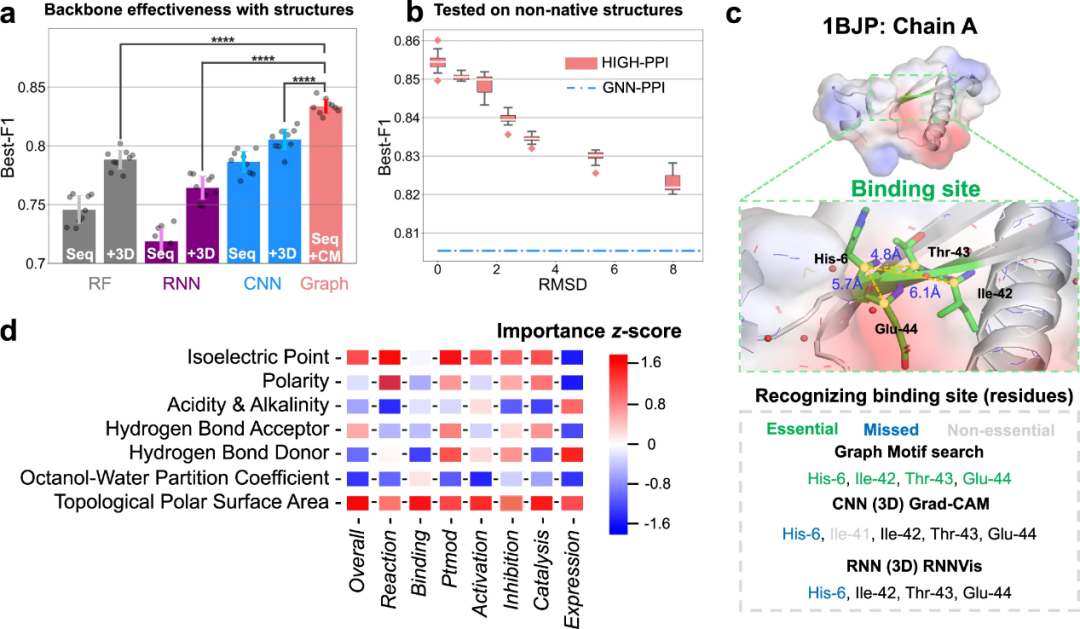
图3:蛋白内视图带来增益。(a)引入蛋白质3D信息显著提升预测性能,且在保证输入信息相同的前提下,图结构(Graph)相对循环神经网络(RNN)和卷积神经网络(CNN)具有明显优势;(b)和基线(蓝色点虚线)对比,采用相同的输入信息训练后,图结构数据对蛋白结构误差表现出高容忍度;此外,图结构数据还具备较高的可解释性,例如(c)精确识别蛋白中对接位点和(d)对残基的属性重要性打分。
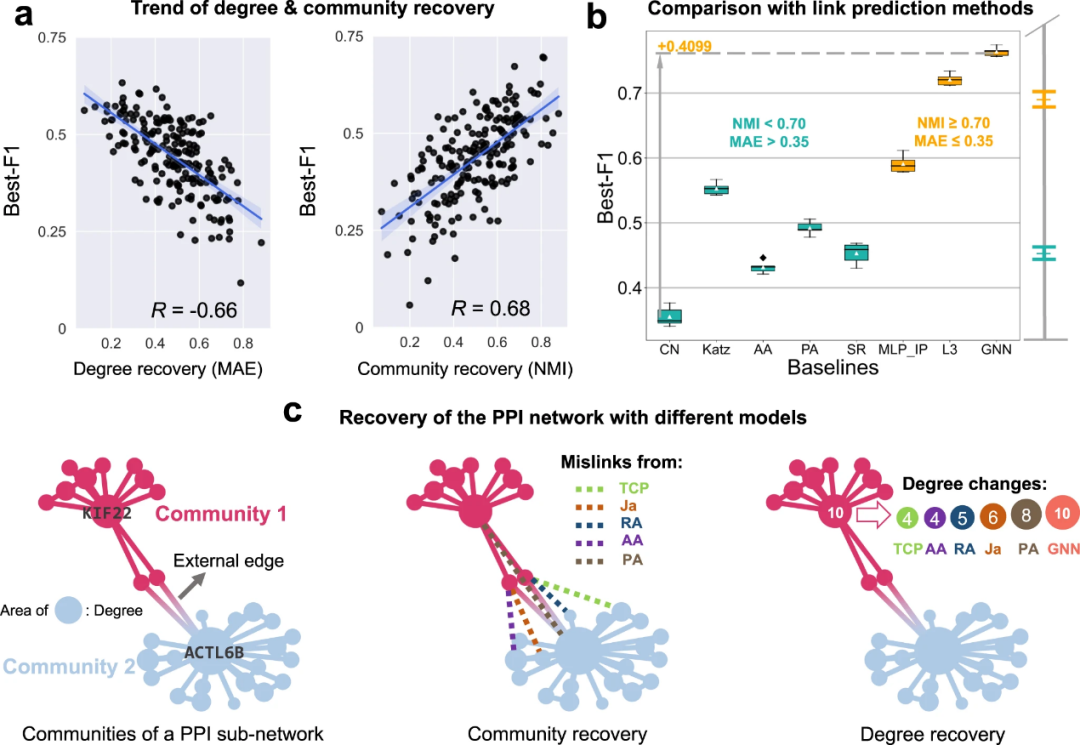
图4:交互作用组学信息的应用。(a)PPI网络节点度(左)和社区属性(右)分别与PPI预测结果F1分数的高相关性;(b)不同链路预测方法的F1分数,有效的链路预测方法都倾向于高度还原PPI网络的结构属性;(c)PPI网络示意图,每个节点的面积代表它的度值,只有两条外部的边连接检测到的两个社区(左),真实计算结果显示其他链路预测方法会产生错误的链路,这可能会破坏社区的划分(中),并且在恢复节点度方面存在缺陷(右)。
为训练和评估 HIGH-PPI,这项研究使用了 STRING 数据库中的人类 PPI 数据集,包含 1,690 种蛋白质和 7624 个 PPI。
对比发现,HIGH-PPI 的表现超出四种业内现有预测模型,和排名第二的 GNN-PPI 相比表现提升 4.42%。为了模拟实际应用中的数据不可靠性,研究还对PPI网络进行随机扰动,并测试其性能指标。实验证明,当测试集中存在 70% 的未知蛋白时,HIGH-PPI 仍可保持较高精度。
机器预测、实验验证,是未来生物、医药研究的重要趋势,为了让后续的验证实验更加高效,科学家需要能够充分理解模型的预测结果,这一指标称为模型的可解释性。例如,假设 HIGH-PPI 预测某个蛋白质对存在催化相互作用,但识别出与催化无关的重要位点,我们将很难相信模型的决定。
这项研究将模型经过学习预测的蛋白质功能域与真实数据进行对比,证明 HIGH-PPI 模型具有出色的可解释性,能够为后续的湿实验验证(实验室生化实验)提供可靠指导。例如,如果 HIGH-PPI 认为某个催化位点很重要,就可以针对特定位点,设计相应的验证实验。
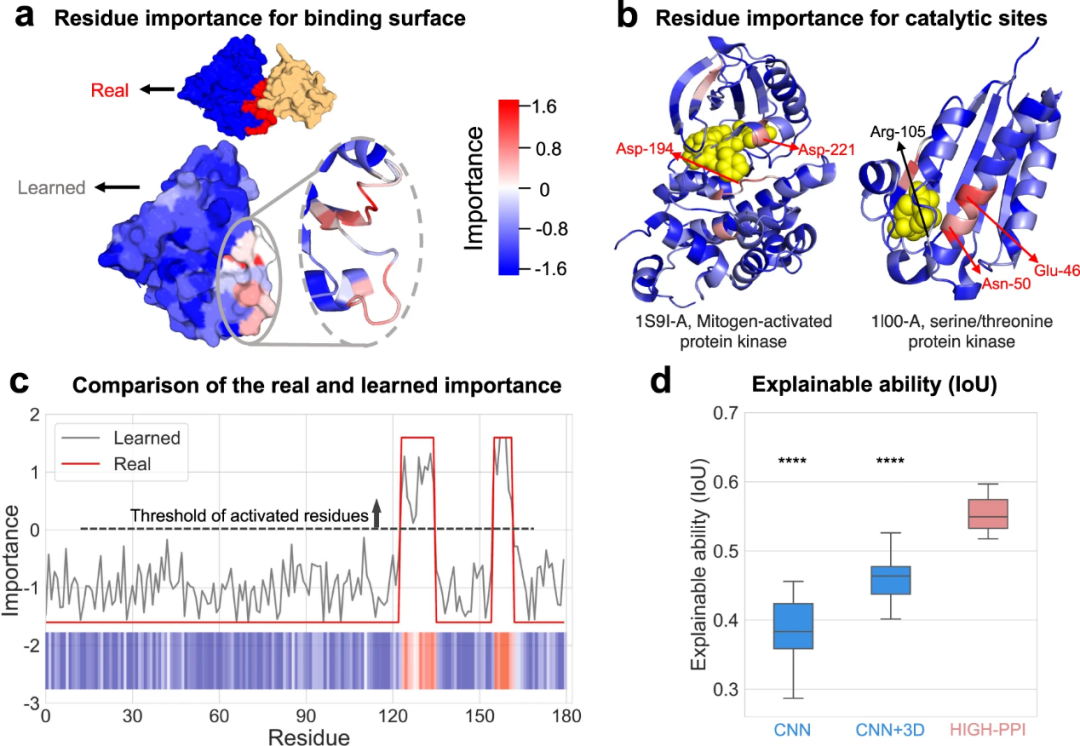
图5:在没有监督的情况下自动解释残基重要性。(a)对残基重要性评分时,目标蛋白质结合面上的残基获得高分(红色),其他残基获得低分(蓝色);(b)对两种蛋白质的残基进行重要性评分,正确识别为具有催化功能的残基叠加红色,错误识别叠加黑色;(c)将5a中模型预测的重要性评分(灰色折线)与该结合反应中的真实功能域(红色)进行对比;(d)通过计算对比真实功能域与模型预测的功能域,评估PPI预测结果的可解释性。
HIGH-PPI 模型是腾讯在 AI+ 生命科学研究领域的最新研究成果,此前,团队在蛋白质相关的药物发现领域已经取得诸多领先成果,发表多项关于蛋白质结构-功能关系研究的文章,如蛋白质高精度结构模型、疾病分类、蛋白动力学建模以及蛋白质组学数据等。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


