Nature:构建出在人类细胞中发现的300多种蛋白激酶的综合图谱
来源:生物谷原创 2023-01-20 12:45
在一项新的研究中,研究人员构建出一个包含在人类细胞中发现的300多种蛋白激酶的综合图谱,并确定了它们可能靶向和控制哪些蛋白。相关研究结果于2023年1月11日在线发表在Nature期刊上。
人类最重要的酶之一是蛋白激酶,它们是调节几乎所有细胞活动(包括生长、细胞分裂和代谢)的信号分子。这些细胞途径的功能失调可导致多种疾病,特别是癌症。
识别参与细胞功能障碍和癌症发展的蛋白激酶可以产生许多新的药物靶标,但是对于这些激酶中的绝大多数,科学家们并没有清楚地了解它们参与了哪些细胞途径,或者它们的底物是什么。
美国麻省理工学院精准癌症医学中心主任Michael Yaffe教授说,“我们有大量的癌症基因组测序数据,但我们缺少的是对癌症中信号通路和蛋白激酶激活状态的大规模研究。如果我们有这些信息,我们就会对如何治疗特定的肿瘤有更好的想法。”

在一项新的研究中,Yaffe、耶鲁大学医学院的Benjamin E. Turk博士和威尔康乃尔医学院的Lewis C. Cantley博士及其同事们构建出一个包含在人类细胞中发现的300多种蛋白激酶的综合图谱,并确定了它们可能靶向和控制哪些蛋白。这些信息可能帮助科学家们破译许多细胞信号通路,并帮助他们发现当细胞发送癌变或用特定药物治疗时,这些信号通路会发生什么变化。相关研究结果于2023年1月11日在线发表在Nature期刊上,论文标题为“An atlas of substrate specificities for the human serine/threonine kinome”。
可靠线索
人类基因组包括500多种蛋白激酶,它们通过给其他蛋白标记上称为磷酸基团的化学修饰来激活或失活这些蛋白。对于这些激酶中的大多数,它们所靶向的蛋白是未知的,尽管对MEK和RAF等激酶的研究已导致了抑制这些激酶的新癌症药物,其中MEK和RAF这两种激酶都参与了控制生长的细胞途径。
为了确定癌细胞中失调的其他途径,这些作者依靠使用质谱的磷酸化蛋白质组学(phosphoproteomics)来发现癌细胞或健康细胞中磷酸化程度较高的蛋白。然而,在此之前,还没有一种简单的方法来研究质谱数据,以确定哪些蛋白激酶负责磷酸化这些蛋白。正因为如此,这些蛋白在疾病中是如何受到调控或遭受异常调控的仍是未知的。
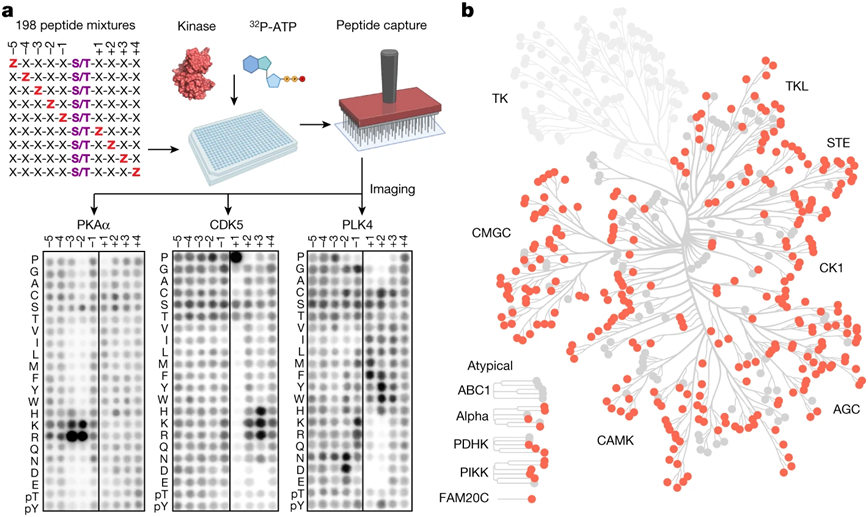
分析人类丝氨酸/苏氨酸激酶组的底物特异性。图片来自Nature, 2023, doi:10.1038/s41586-022-05575-3。
Yaffe说,“对于大多数被测量的磷酸肽(phosphopeptide),我们不知道它们在信号传导途径中的位置。我们没有有效线索用来观察这些磷酸肽,然后说,这是这些数据告诉我们的信号传导途径。原因是对于大多数蛋白激酶,我们不知道它们的底物是什么。”
在这项新的研究中,这些作者分析了两类激酶---丝氨酸激酶和苏氨酸激酶,这两类激酶构成了人体中大约85%的蛋白激酶。他们利用Cantley和Turk之前创建的一个肽库来寻找与激酶相互作用的基序,随后测量了这些肽与所有303种已知的丝氨酸激酶和苏氨酸激酶的相互作用。对于这两类激酶而言,通过使用一种计算模型来分析他们观察到的相互作用,他们能够识别出能够使人类细胞中已报道的9万个已知磷酸化位点中的每一个磷酸化的激酶。
令他们吃惊的是,这些作者发现许多具有非常不同的氨基酸序列的激酶已经进化到能够结合和磷酸化其底物上的相同基序。他们还发现,他们所研究的激酶中约有一半靶向三大类基序之一,而剩下的一半则靶向大约十几个小类基序之一。
解码网络
Yaffe说,这种新的激酶图谱可以帮助科学家们识别正常细胞和癌细胞之间,或经过治疗的癌细胞和未经治疗的癌细胞之间不同的信号通路。他说,“这个激酶基序图谱现在可以让我们解读信号传导网络。我们可以看看所有这些磷酸化的肽,我们可以把它们对应到一种特定的激酶上。”
为了展示这种方法,这些作者分析了用一种抗癌药物处理的细胞,这种药物抑制一种叫做Plk1的激酶,该激酶调节细胞分裂。当他们分析磷酸化蛋白的表达时,他们发现许多受影响的蛋白是由Plk1控制的,正如他们预期的那样。令他们吃惊的是,他们还发现这种处理增加了两种激酶的活性,这两种激酶参与了细胞对DNA损伤的反应。
Yaffe实验室如今有兴趣使用这种图谱来尝试寻找其他驱动癌症发展的功能失调的信号通路,特别是在某些没有发现遗传驱动因素的癌症类型中。
他说,“我们如今可以用磷蛋白组学说,也许在这个病人的肿瘤中,这些途径是上调的,或者这些途径是下调的。我们现在可以利用磷酸化蛋白质组学来判断,也许在这名患者的肿瘤中,这些通路上调或下调。它可能会确定在由不明显的遗传因素导致癌症的情况下驱动癌症的信号通路。”(生物谷 Bioon.com)
参考资料:
Jared L. Johnson et al. An atlas of substrate specificities for the human serine/threonine kinome. Nature, 2023, doi:10.1038/s41586-022-05575-3.
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


