Nat Methods:瞿昆/黎斌/陈发来合作系统性评估单细胞多组学分析算法
来源:生物探索 2024-10-02 09:07
结果显示,在蛋白质丰度预测方面,totalVI 和 scArches 表现最为优异;在染色质可及性预测中,LS_Lab 算法排名领先。
中国科学技术大学生命科学与医学部瞿昆教授课题组、北京生命科学研究所黎斌研究员课题组,以及中国科学技术大学数学科学学院陈发来教授课题组联合在Nature Methods期刊上发表了文章“Benchmarking algorithms for single-cell multi-omics prediction and integration”。他们通过对百万量级单细胞多组学数据进行分析,系统评估了14种单细胞模态预测算法和18种单细胞多组学整合算法的性能。

在本次研究中,团队收集了来自47个数据集的上百万个单细胞多组学数据,涵盖多个生物样本和实验平台。他们设计了一套全面的评估流程,结合算法的准确性、鲁棒性和计算资源消耗等多维度指标,系统评估了领域内最常用的算法。结果显示,在蛋白质丰度预测方面,totalVI 和 scArches 表现最为优异;在染色质可及性预测中,LS_Lab 算法排名领先。在多组学整合分析中,Seurat、MOJITOO 和 scAI 在垂直整合上表现突出,而 totalVI 和 UINMF 在水平整合和马赛克整合任务中展现了卓越性能。这一研究不仅为算法设计提供了新思路,还为未来多组学数据的分析和应用奠定了重要基础。为帮助科研人员选择合适的分析工具,研究团队在GitHub上发布了完整的分析流程、代码和测试数据集,供同行使用和改进。
研究团队还通过深入探讨这些算法的数学原理,发现降噪处理是提高单细胞数据预测精度的关键。在性能评估中,机器学习算法(如基于奇异值分解的LS_Lab 和 Guanlab-dengkw)以及基于概率模型的深度学习算法(如totalVI)均表现出显著优势。然而,研究还指出,现有模态预测算法在某些关键蛋白的预测性能上仍有待提升,染色质可及性预测的准确性也需进一步优化。
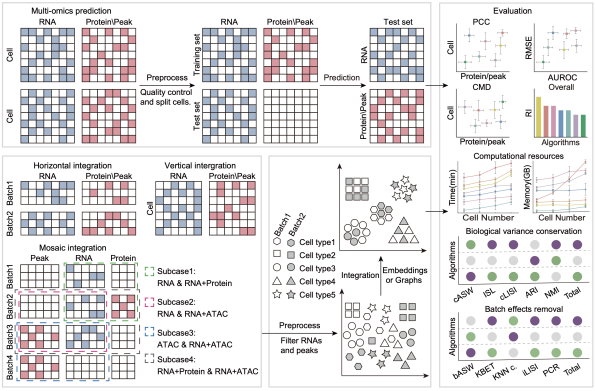
图1. 评估流程示意图(Credit: Nature Methods)
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


