Nat Chem Biol丨实现小鼠体内Cas9难编辑位点的高效编辑!杨辉/张海南/黄锦海团队开发出靶向更广的微型碱基编辑器
来源:生物探索 2024-08-18 09:10
该研究通过对新型IscB的挖掘和优化改造,开发出靶向范围更广的高活性、高特异性的迷你型碱基编辑工具IscB.m16*-BE,在基于AAV的基因治疗应用中显示出独特的优势和巨大的潜力。
CRISPR/Cas9系统自发现以来,得到快速发展已被广泛应用生命科学基础研究、基因治疗、动植物育种改良等领域【1】。基于Cas9切口酶(nCas9)与脱氨酶结构域/糖基化酶(MPG或UNG)的融合成的碱基编辑器(ABE,CBE,gGBE,gTBE),可高效实现A-to-G,C-to-T,C-to-G,G-to-C/T,以及T-to-G/C的碱基替换,为纠正突变的疾病位点提供了精准高效基因编辑工具【2-6】。然而由于Cas9的体积过大(1368个氨基酸),基于nCas9的碱基编辑器难以实现单个AAV(4.7kb) 的包装递送,极大限制了在体基因编辑的发展应用。近年来一系列紧凑型的Cas9蛋白【7-9】、Cas12f系列同源物【10-14】、以及其祖先蛋白TnpB【15,16】被报道,由于编辑活性有限,或缺乏HNH结构域而难以改造为缺口酶,都限制用于碱基编辑器的开发。2021年,张锋团队发现由IS200/IS605转座子超家族编码的IscB核酸酶,被认为Cas9的祖先蛋白,具有与Cas9相似的HNH和RuvC结构域,且仅有约500个氨基酸(约SpCas9的1/3大小)【17,18】,具有开发成微型碱基编辑器的潜力。
2023年,杨辉团队通过对OgeuIscB/ωRNA系统的工程化改造,开发出了高效的OgeuIscB变体(enOgeuIscB),并通过融合脱氨酶结构域,开发出高效迷你型碱基编辑器(miBE),推动了DNA单碱基编辑领域进入迷你型的“新时代”,具有极大的临床应用潜力【19】。然而,IscB/ωRNA系统需要严格的6位碱基靶序列邻近基序(TAM)来识别目标DNA,识别位点有限。因此,开发靶标识别范围更广的高效小型IscB碱基编辑器是十分必要。
2024年8月15日,辉大(上海)生物科技有限公司研发团队、复旦大学附属眼耳鼻喉科医院黄锦海团队和中科院脑科学与智能技术卓越创新中心杨辉团队合作在Nature Chemical Biology上发表题为Engineered IscB–ωRNA system with expanded target range for base editing的研究论文。该研究通过宏基因组数据挖掘,鉴定出19种具有不同TAM范围的新型IscB-ωRNA系统;综合RNA结构优化、蛋白质工程化改造、流式细胞术、脱靶检测等技术手段,成功获得在人类细胞内具有靶标识别范围广、更高效编辑活性的IscB系统(IscB.m16*);通过融合脱氨酶结构域,进一步开发出基于新型IscB的迷你型腺嘌呤和胞嘧啶碱基编辑器,并在哺乳动物细胞和小鼠疾病模型中包括SpCas9-BE无活性的疾病位点上均验证了其强大的碱基编辑效率和广泛的靶标识别能力,为未来精准基因治疗临床应用提供了强有力支持。

研究人员首先从200GB的宏基因组数据库中挖掘出19个未被表征的新型IscB系统,采用细菌耗竭实验鉴定相应的TAM序列;进一步利用荧光报告系统,筛选出10个具有真核细胞活性的IscB系统,其中IscB.m16表现出最高的编辑活性。为提高IscB.m16系统的活性并拓宽TAM范11:33:58围,研究人员对IscB.m16核酸酶进行RuvC结构域的精氨酸扫描突变和TAM识别相关位点的饱和突变,以及对其ωRNA进行茎环截短和碱基替换的优化改造。通过多轮迭代的高通量荧光报告系统筛选,最终获得了编辑活性高和TAM范围宽的IscB.m16变体(IscB.m16*,即IscB.m16RESH-ωRNA)。通过细菌耗竭TAM序列识别实验发现,相较于野生型IscB.m16的TAM位点MRNRAA扩展到NNNGNA。
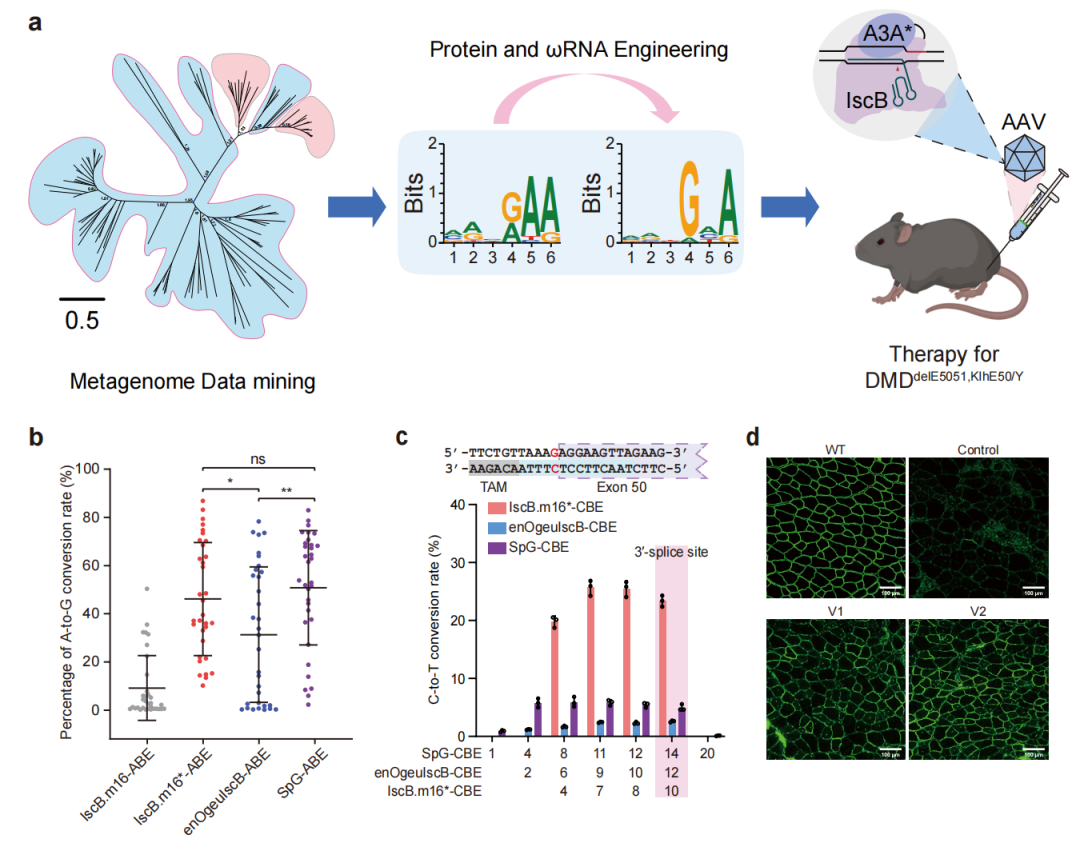
(Credit: Nature Chemical Biology)
在此基础上,研究人员构建了迷你型腺嘌呤碱基编辑器(IscB.m16*-ABE)和胞嘧啶碱基编辑器(IscB.m16*-CBE)。在哺乳动物细胞中,IscB.m16*-ABE的碱基编辑效率与SpG-ABE效率相当,显著高于已报道的enOgeuIscB-ABE且有更广的TAM兼容性。在人源化人源化杜氏肌营养不良症(DMD)小鼠疾病模型中,单AAV包装的IscB.m16*-CBE经注射至肌肉组织后,成功并高效的将小鼠肌纤维中dystrophin蛋白水平恢复至野生型小鼠的40%,为DMD患者提供了一种有希望的基因治疗策略。
总的来说,该研究通过对新型IscB的挖掘和优化改造,开发出靶向范围更广的高活性、高特异性的迷你型碱基编辑工具IscB.m16*-BE,在基于AAV的基因治疗应用中显示出独特的优势和巨大的潜力。
1. Porto, E.M., Komor, A.C., Slaymaker, I.M., and Yeo, G.W. (2020). Base editing: advances and therapeutic opportunities. Nature Reviews Drug Discovery 19, 839–859. https://doi.org/10.1038/s41573-020-0084-6.
2. Komor, A.C., Kim, Y.B., Packer, M.S., Zuris, J.A., and Liu, D.R. (2016). Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature 533, 420–424. https://doi.org/10.1038/nature17946.
3. Gaudelli, N.M., Komor, A.C., Rees, H.A., Packer, M.S., Badran, A.H., Bryson, D.I., and Liu, D.R. (2017). Programmable base editing of A•T to G•C in genomic DNA without DNA cleavage. Nature 551, 464–471. https://doi.org/10.1038/nature24644.
4. Kurt, I.C., Zhou, R., Iyer, S., Garcia, S.P., Miller, B.R., Langner, L.M., Grünewald, J., and Joung, J.K. (2021). CRISPR C-to-G base editors for inducing targeted DNA transversions in human cells. Nat Biotechnol 39, 41–46. https://doi.org/10.1038/s41587-020-0609-x.
5. Tong, H., Liu, N., Wei, Y., Zhou, Y., Li, Y., Wu, D., Jin, M., Cui, S., Li, H., Li, G., et al. (2023). Programmable deaminase-free base editors for G-to-Y conversion by engineered glycosylase. National Science Review, nwad143. https://doi.org/10.1093/nsr/nwad143.
6. Tong, H., Wang, H., Wang, X., Liu, N., Li, G., Wu, D., Li, Y., Jin, M., Li, H., Wei, Y., et al. (2024). Development of deaminase-free T-to-S base editor and C-to-G base editor by engineered human uracil DNA glycosylase. Nat Commun 15, 4897. https://doi.org/10.1038/s41467-024-49343-5.
7. Chen, S., Liu, Z., Xie, W., Yu, H., Lai, L., and Li, Z. (2022). Compact Cje3Cas9 for Efficient In Vivo Genome Editing and Adenine Base Editing. The CRISPR Journal 5, 472–486. https://doi.org/10.1089/crispr.2021.0143.
8. Davis, J.R., Wang, X., Witte, I.P., Huang, T.P., Levy, J.M., Raguram, A., Banskota, S., Seidah, N.G., Musunuru, K., and Liu, D.R. (2022). Efficient in vivo base editing via single adeno-associated viruses with size-optimized genomes encoding compact adenine base editors. Nat. Biomed. Eng 6, 1272–1283. https://doi.org/10.1038/s41551-022-00911-4.
9. Zhang, H., Bamidele, N., Liu, P., Ojelabi, O., Gao, X.D., Rodriguez, T., Cheng, H., Kelly, K., Watts, J.K., Xie, J., et al. (2022). Adenine Base Editing In Vivo with a Single Adeno-Associated Virus Vector. GEN Biotechnology 1, 285–299. https://doi.org/10.1089/genbio.2022.0015.
10. Hino, T., Omura, S.N., Nakagawa, R., Togashi, T., Takeda, S.N., Hiramoto, T., Tasaka, S., Hirano, H., Tokuyama, T., Uosaki, H., et al. (2023). An AsCas12f-based compact genome-editing tool derived by deep mutational scanning and structural analysis. Cell 186, 4920-4935.e23. https://doi.org/10.1016/j.cell.2023.08.031.
11. Kim, D.Y., Lee, J.M., Moon, S.B., Chin, H.J., Park, S., Lim, Y., Kim, D., Koo, T., Ko, J.-H., and Kim, Y.-S. (2022). Efficient CRISPR editing with a hypercompact Cas12f1 and engineered guide RNAs delivered by adeno-associated virus. Nat Biotechnol 40, 94–102. https://doi.org/10.1038/s41587-021-01009-z.
12. Kong, X., Zhang, H., Li, G., Wang, Z., Kong, X., Wang, L., Xue, M., Zhang, W., Wang, Y., Lin, J., et al. (2023). Engineered CRISPR-OsCas12f1 and RhCas12f1 with robust activities and expanded target range for genome editing. Nat Commun 14, 2046. https://doi.org/10.1038/s41467-023-37829-7.
13. Xu, X., Chemparathy, A., Zeng, L., Kempton, H.R., Shang, S., Nakamura, M., and Qi, L.S. (2021). Engineered miniature CRISPR-Cas system for mammalian genome regulation and editing. Molecular Cell 81, 4333-4345.e4. https://doi.org/10.1016/j.molcel.2021.08.008.
14. Wu, Z., Zhang, Y., Yu, H., Pan, D., Wang, Y., Wang, Y., Li, F., Liu, C., Nan, H., Chen, W., et al. (2021). Programmed genome editing by a miniature CRISPR-Cas12f nuclease. Nat Chem Biol 17, 1132–1138. https://doi.org/10.1038/s41589-021-00868-6.
15. Karvelis, T., Druteika, G., Bigelyte, G., Budre, K., Zedaveinyte, R., Silanskas, A., Kazlauskas, D., Venclovas, Č., and Siksnys, V. (2021). Transposon-associated TnpB is a programmable RNA-guided DNA endonuclease. Nature 599, 692–696. https://doi.org/10.1038/s41586-021-04058-1.
16. Kim, D.Y., Chung, Y., Lee, Y., Jeong, D., Park, K.-H., Chin, H.J., Lee, J.M., Park, S., Ko, S., Ko, J.-H., et al. (2022). Hypercompact adenine base editors based on transposase B guided by engineered RNA. Nat Chem Biol 18, 1005–1013. https://doi.org/10.1038/s41589-022-01077-5.
17. Altae-Tran, H., Kannan, S., Demircioglu, F.E., Oshiro, R., Nety, S.P., McKay, L.J., Dlakić, M., Inskeep, W.P., Makarova, K.S., Macrae, R.K., et al. (2021). The widespread IS200/IS605 transposon family encodes diverse programmable RNA-guided endonucleases. Science 374, 57–65. https://doi.org/10.1126/science.abj6856.
18. Schuler, G., Hu, C., and Ke, A. (2022). Structural basis for RNA-guided DNA cleavage by IscB-ωRNA and mechanistic comparison with Cas9. Science 376, 1476–1481. https://doi.org/10.1126/science.abq7220.
19. Han, D., Xiao, Q., Wang, Y., Zhang, H., Dong, X., Li, G., Kong, X., Wang, S., Song, J., Zhang, W., et al. (2023). Development of miniature base editors using engineered IscB nickase. Nat Methods. https://doi.org/10.1038/s41592-023-01898-9.
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


