生成式人工智能设计出对抗超级细菌的全新抗生素,还给出了合成路线
来源:生物世界 2024-03-30 09:03
研究结果表明,生成式人工智能具有从广泛的化学空间中设计结构新颖、可合成和有效的小分子抗生素候选物的潜力。
麦克马斯特大学和斯坦福大学的研究人员在 Nature 子刊 Nature Machine Intelligence 上发表了题为:Generative AI for designing and validating easily synthesizable and structurally novel antibiotics 的研究论文。
该研究开发了一种新的生成式人工智能(Generative AI)模型——SyntheMol,可以从近300亿个分子的化学空间中设计易于合成的新型化合物。研究团队应用SyntheMol设计能够抑制超级细菌鲍曼不动杆菌的分子,从中合成了58个生成的分子并验证了其中6个结构新颖的分子对鲍曼不动杆菌和其他几种系统发育不同的细菌病原体表现出抗菌活性。
该研究表明了生成式人工智能从巨大的化学空间中设计结构新颖、易于合成和有效的小分子抗生素候选物的潜力,代表了将生成式人工智能用于抗生素发现和药物发现的实际应用迈出的重要一步。

鲍曼不动杆菌(Acinetobacter baumannii),是出了名的难以根除,会导致肺炎、脑膜炎和感染伤口,所有这些都可能导致死亡。世界卫生组织(WHO)已经将鲍曼不动杆菌确定为世界上最危险的抗生素耐药细菌之一。
抗生素是一种很独特的药物,一旦开始在临床上使用,我们就要倒计时它的失效时间。因为细菌会迅速进化出抗性。因此,我们需要一个强大的抗生素管线,需要快速、低成本地发现新型抗生素,而这就是人工智能能够发挥关键作用的地方。
在这项研究中,研究团队开发了一种生成式人工智能模型——SyntheMol,以快速、低成本地访问数百亿个有前途的分子。他们从132000个分子片段的库中提取,这些片段就像乐高积木一样组合在一起,但本质上却非常不同。然后将这些分子片段与一组13个化学反应进行交叉参照,从而识别出300亿个片段的双向组合,以设计具有最有前途的抗菌特性的新分子。该模型设计的每个分子都被传递到另一个经过训练的AI模型中,以预测毒性。
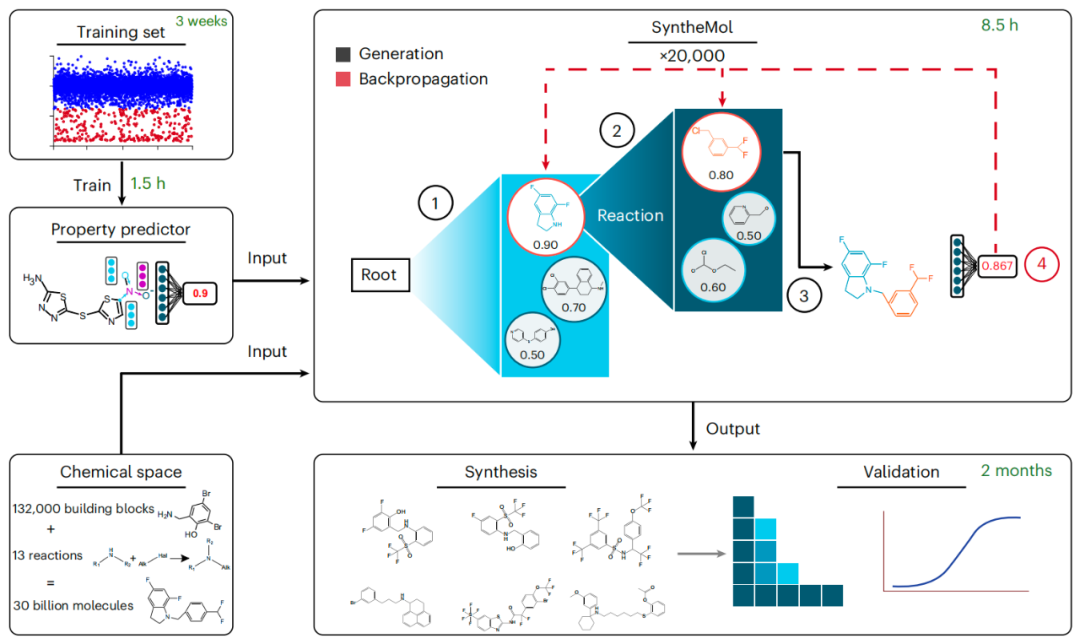
生成式人工智能用于抗生素发现
接下来,研究团队合成了58个由SyntheMol生成的分子,并进行了实验验证,其中6个结构新颖的分子对鲍曼不动杆菌和其他几种系统发育不同的细菌病原体(肺炎克雷伯菌、甲氧西林耐药金黄色葡萄球菌等)表现出强大的抗菌活性,且无毒。这些结果表明了生成式人工智能具有从广泛的化学空间中设计结构新颖、可合成和有效的小分子抗生素候选物的潜力。
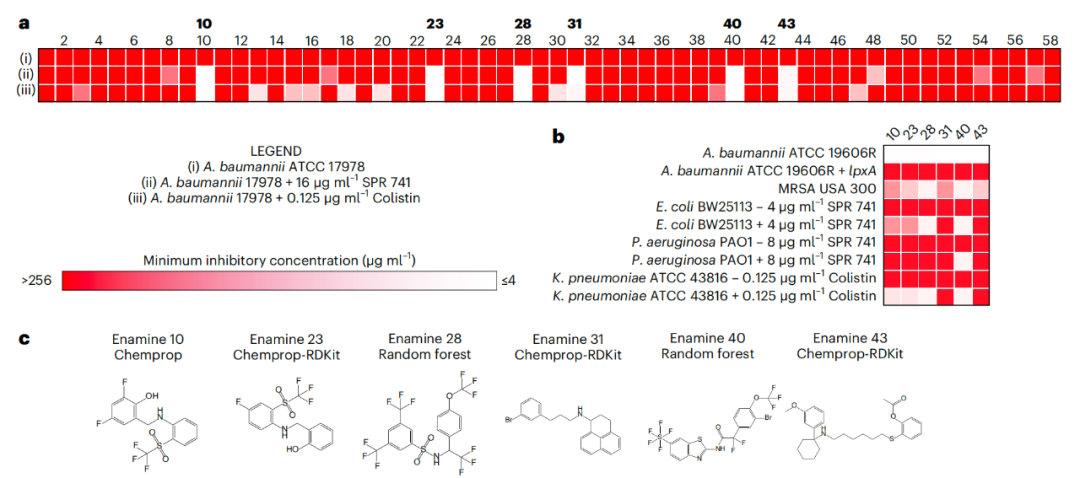
生成分子的体外验证实验
目前,SyntheMol模型还有一些不足之处,例如,它没有被编程来产生高水溶性的分子,上述6个新分子中有4个水溶性不足,因此只有2个可以在小鼠模型中进行体内测试。
虽然还需要更多的研究来将这些生成的新分子转化为可行的抗生素候选者,并改进生成式人工智能方法用于可合成分子的设计,但这项工作代表了将生成式人工智能用于抗生素发现和药物发现的实际应用迈出的重要一步。
论文共同通讯作者 James Zou 表示,SyntheMol不仅设计了有前途的候选抗生素分子,而且还生成了如何制造每个新分子的配方。生成这种配方是一种新的方法和游戏规则改变者,因为化学家并不知道如何制造AI设计的分子。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


