Nat Cell Biol: 孟飞龙/叶菱秀团队发现C-to-G碱基编辑产生双链断裂,导致删除、翻转和易位
来源:生物探索 2024-01-26 10:13
这项工作表明,CGBEs可以启动有害的中间DSBs,因此需要仔细考虑治疗应用,并且HMCES辅助的CGBEs有望成为更安全的工具。
中国科学院分子细胞科学卓越创新中心孟飞龙及上海交通大学叶菱秀共同通讯在Nature Cell Biology (IF 21.3)在线发表题为“C-to-G editing generates double-strand breaks causing deletion, transversion and translocation”的研究论文,该研究进行了深入的结果分析和遗传剖析,揭示了C-to-G BEs (CGBEs)产生大量的中间双链断裂(DSBs)。不完美的DSB末端连接导致末端切除、模板插入或末端填充期间的异常翻转导致小的缺失。在Cas9/脱氨酶起源的编辑靶点和非靶点之间检测到染色体易位。DNA修复因子的遗传筛选揭示了基本位点加工在DSB形成中的核心作用。自杀酶HMCES屏蔽基本位点减少了CGBEs引发的DSBs,提供了一种有效的方法来减少DSBs引发的事件,而不影响取代。这项工作表明,CGBEs可以启动有害的中间DSBs,因此需要仔细考虑治疗应用,并且HMCES辅助的CGBEs有望成为更安全的工具。

碱基编辑器(BEs)在没有双链DNA切割的情况下进行碱基替换,因此与链切割编辑器相比,提供了一种更安全的治疗性基因组编辑方法。然而,不希望的副产品,如意外的替换、插入或缺失,在各种基因组环境中都会发生,并可能在临床环境中产生有害的后果。副产物通常被称为“随机”产物,因为它们通常以1%或更低的频率出现,这使得在常规分析中研究它们具有挑战性。此外,BEs通过Cas依赖性或非依赖性机制产生大量脱靶基因组突变。胞苷编辑对基因组改变的影响,包括插入/缺失(indels)和染色体结构变异(SVs),可能被低估了。
虽然优化的Cas和/或脱氨酶模块可以减少碱基编辑副产物,但DNA修复因子提供了最小化副产物或指导结果的替代方法。例如,可选的尿嘧啶糖基酶抑制剂(UGI)或尿嘧啶糖基酶(UNG)模块可以改变胞嘧啶BEs (CBEs)或C-to-G BEs (CGBEs) 的取代结果。此外,在胞嘧啶碱基编辑中加入细菌双链断裂(DSB)末端结合蛋白Gam已被证明可以减少indels。然而,Gam并不能消除DSB的形成,持续的DSB可能会导致其他类型的基因组不稳定,如易位。
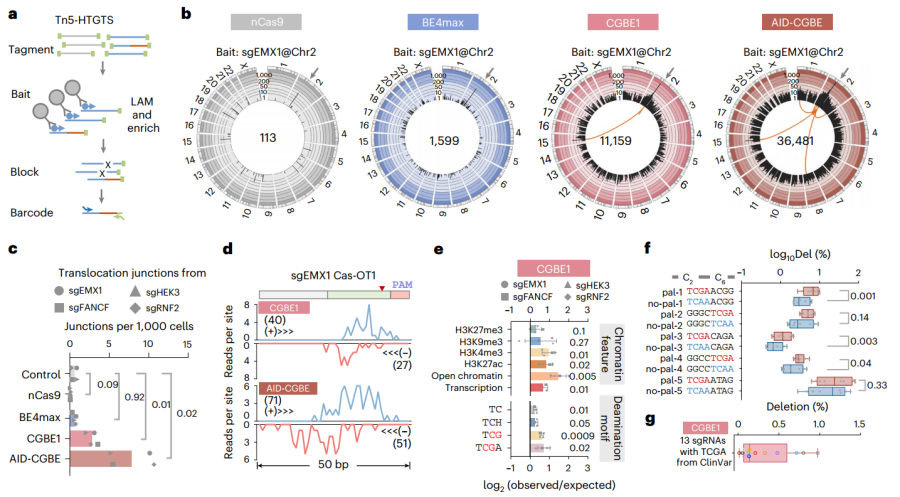
CGBEs引发的染色体易位检测(Source: Nature Cell Biology )
为了分析低频胞苷编辑副产物并剖析潜在的DNA修复机制,研究人员在几个位点上对胞苷编辑结果进行了深度测序,并使用集中聚集规律间隔短回文重复序列(CRISPR)筛选研究了DNA修复途径。研究发现胞苷BEs,特别是CGBEs,产生大量的中间DSBs,这些DSBs可以进一步加工为缺失、模板插入、异常翻转或染色体易位。基于所描绘的DNA修复途径,发现自杀酶HMCES可以最大限度地减少DSB的产生,并进一步开发了HMCES辅助的CGBEs。虽然CGBEfH/pH不能完全消除DSBs,但DNA修复信息为CGBE优化提供了未来的思路,并且在不久的将来可以开发出具有背景副产物和更高翻转纯度的完美颠换编辑器。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


