北航李春燕团队全面总结组学技术在增强子研究中的应用
来源:生物世界 2023-12-06 16:23
增强子是基因转录的重要调控元件,组学技术的应用加快了对增强子在基因调控中的作用和机制的阐明。
北京航空航天大学李春燕课题组(博士研究生王棨临为第一作者)在 Briefings in Bioinformatics 期刊发表了题为:Integrative approaches based on genomic techniques in the functional studies on enhancers 的综述论文。
该综述全面总结了目前组学技术在增强子研究中的应用,梳理了常用的增强子数据库和多组学数据整合方法,以及归纳了目前增强子整合技术遇到的挑战。

组学技术在增强子研究中的应用
测序技术的广泛应用为增强子研究提供了丰富的分子信息。基因组学、表观基因组学、转录组学和CRISPR编辑技术在增强子研究中具有巨大的应用价值。
基因组学方法可分为两类:全基因组测序(WGS)和全外显子组测序(WES),主要针对DNA序列信息进行测序;高通量染色体捕获技术(Hi-C)为代表的方法主要针对DNA结构信息进行测序。增强子研究中,全基因组关联研究(GWAS)或表达数量性状位点(eQTL)分析方法可用于探讨增强子区段变异与基因表达之间的关系,助力预测增强子靶基因或功能。Hi-C技术则可通过染色体拓扑结构域(TAD)结构预测物理空间中增强子与靶基因的结合。此外,Hi-C衍生技术如ChIA-PET、HiChIP等可在高分辨率下检测特定的蛋白质介导的染色质环,为增强子分析提供支持。
表观遗传学方法是指不改变DNA序列而调节基因转录或翻译过程进而调控表型特征的一种调控机制。在增强子研究中,表观基因组学方法可以根据研究对象的不同分为四类:染色质可及性、DNA修饰、DNA相互作用和RNA相互作用。
1)真核生物染色质开放状态被认为是转录的先决条件,DNase-seq、MNase-seq、FAIRE-seq和ATAC-seq是四种常见的鉴定染色质开放状态的测序技术。
2)DNA甲基化近些年发现和增强子的活性成负相关。
3)DNA与其他分子(如蛋白质、DNA和RNA)之间的相互作用可以改变DNA的结构或结合亲和力。
这些过程还可以导致增强子的功能改变。ChIP-seq和CUT&Tag是最常用于鉴定转录因子在DNA上的结合位点以及研究DNA与特定组蛋白修饰相互作用的测序方法。FiTAc-seq和FiTAc-seq被开发应用于FFPE(福尔马林固定和石蜡包埋)样本的组蛋白结合信号分析。除此之外,Circle-seq和GRID-seq等技术可以被用来从DNA-DNA和DNA-RNA相互作用角度对增强子的活性改变进行研究。细胞核中的RNA分子通过分子内碱基配对形成二级结构,从而发挥其生物学功能。eRNA和启动子上游反义RNA形成增强子-启动子环来激活转录。因此,解码RNA的高级结构对于理解潜在机制至关重要。RIC-seq可以准确捕获RNA的二级结构并通过杂交序列识别RNA-RNA相互作用,有助于研究eRNA在启动子活性中的调控作用。
转录组方法可以直接检测基因或增强子的表达。在增强子研究中,常用的方法包括 RNA-seq、微阵列和STARR-seq。RNA-seq是一种广泛应用于转录组研究的测序技术,可全面分析基因表达水平。微阵列技术则通过检测基因组中的基因表达水平,揭示增强子与基因表达的关系。STARR-seq是一种针对增强子活性开发的测序方法,通过大规模并行的报告测定法,可定量评估增强子活性,并确定转录增强子的位置。
基因编辑技术与二代测序技术相结合,可实现与特定表型相关的增强子在基因组水平的平行筛选。CRISPR/Cas9技术已被广泛应用于增强子的筛选、功能验证、靶基因鉴定等领域。此外,基于CRISPR的高通量增强子筛选技术,如CRISPR-flowfish,以及 Perturb-seq(也称为CRISPR-seq和CROP-seq)等方法,为研究增强子功能提供了有力工具。
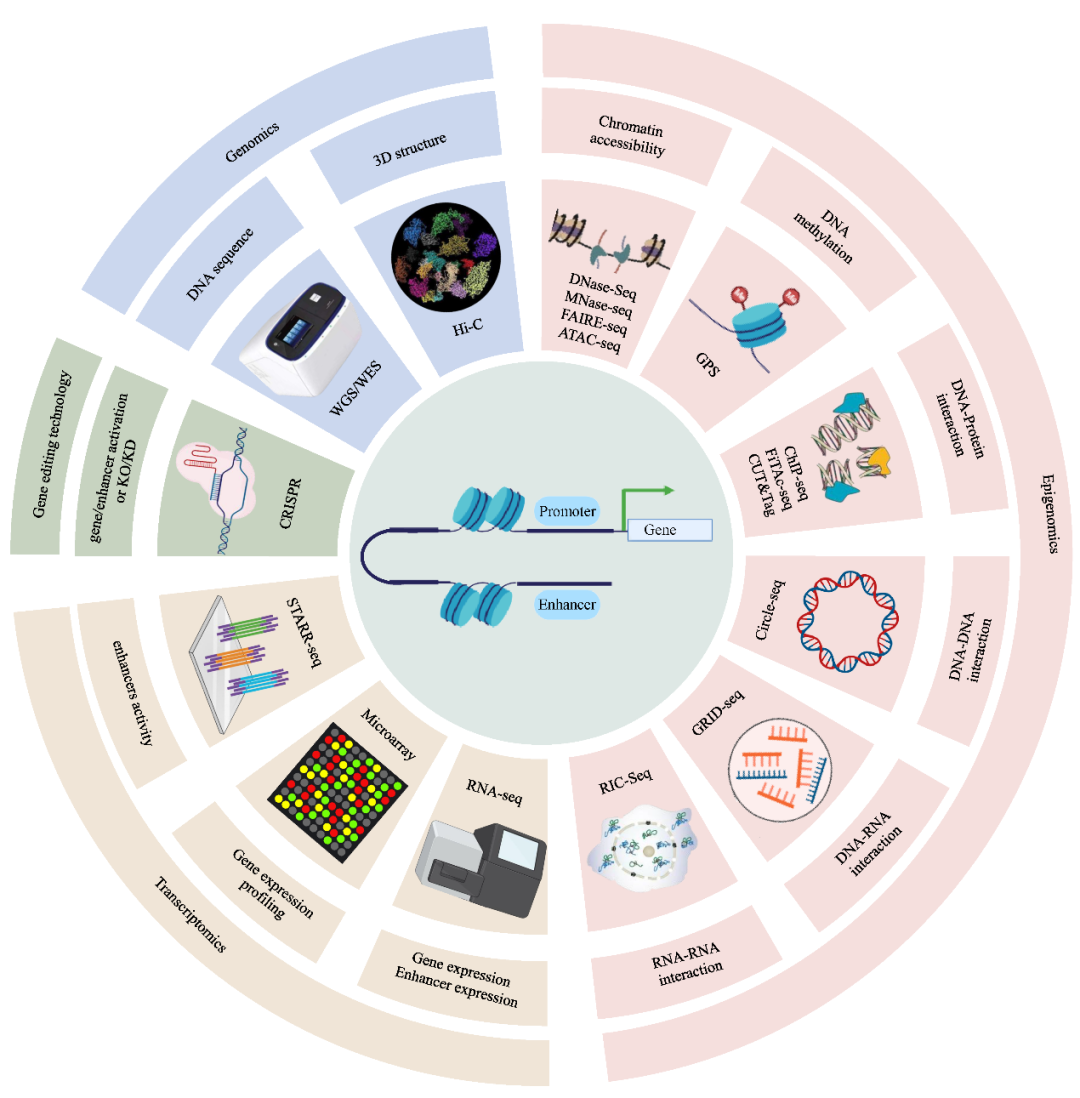
不同组学方法和CRISPR基因编辑技术在增强子研究中的应用
多组学集成方法
近年来,数学、统计学和计算科学的进步为多组学分析的整合提供了基础。根据是否采用神经网络,多组学集成方法可分为两大类:传统机器学习模型和基于神经网络的深度学习模型。
传统机器学习方法,如逻辑回归、随机森林和朴素贝叶斯等,被用于预测和分类未知数据。根据数据是否需要人工标注标签,可分为无监督、半监督和监督三种学习方式。无监督学习方法包括基于距离和基于相关性的方法。最早用于预测增强子靶基因的方法是基于距离的方法,该方法依赖于增强子和基因之间的基因组距离。这种方法假设增强子倾向于调节附近基因的表达。但该方法准确度不高,错误发现率(FDR)约为40%-73%。即使利用RNA表达数据筛选增强子的调控基因,准确性依然较低,FDR值在53%至77%之间。并且基于距离的方法也不能考虑远端调控相互作用以及多个增强子靶向同一启动子的情况。基于相关性的方法在基于距离的方法基础上发展而来,结合特征间的相关性(如组蛋白修饰、增强子和启动子的染色体开放程度信号、基因转录水平等)提高预测精度。
半监督学习采用涵盖未标记和标记数据的算法进行训练,在缺乏足够标记数据集用于监督学习时,这种方法具有优势。相较于监督学习,半监督学习有助于降低过拟合,提高模型稳定性。监督学习依赖于高置信度的正标签和负标签训练数据集(增强子和非增强子)。根据模型所采用的算法,半监督学习和监督学习可分为基于回归的方法和分类器训练方法。基于回归的方法(如McEnhancer、JEME、FENRIR和FOCS)整合了增强子和启动子的特征或基因表达,以确定增强子与靶基因之间的调控关系。分类器训练方法则利用实验识别的增强子-启动子相互作用作为金标准集。通过学习序列和表观遗传修饰特征,可以训练分类器来预测给定增强子-启动子对是否存在相互作用。
深度学习在增强子研究中的应用日益广泛。神经网络在增强子研究中具有显著优势,如能跨不同细胞类型进行预测,降低计算和时间成本。卷积神经网络(CNN)已成为增强子研究中的常用方法,包括DNABERT、iEnhancer-GAN、gcmerge、GraphReg、EPIVAN和DeepTACT等多种模型。DNABERT作为一种预训练双向编码器,可通过学习DNA序列信息揭示不同顺式DNA之间的潜在关联。iEnhancer-GAN集成了词嵌入和序列生成对抗网络,以预测增强子-靶基因相互作用的结合强度。DeepTACT应用自引导深度学习模型,整合基因组序列和染色质可及性数据来预测增强子-启动子相互作用。GC-MERGE是一个基于图形的深度学习框架,通过图形卷积网络解码Hi-C图谱,以捕获潜在基因组空间结构。它通过模拟表观遗传修饰信号和DNA序列信息来预测由远端增强子调控的靶基因。除CNN外,基于深度神经网络(DNN)的架构也用于学习增强子特征,如EP-DNN、ES-ARCNN等。虽然深度学习在增强子预测应用中优于许多传统计算机方法,但过参数化和模型性能有限等问题仍然存在,可解释性也落后于传统统计方法。随着新型深度学习方法的不断发展,有望在增强子研究中实现更优雅的应用。
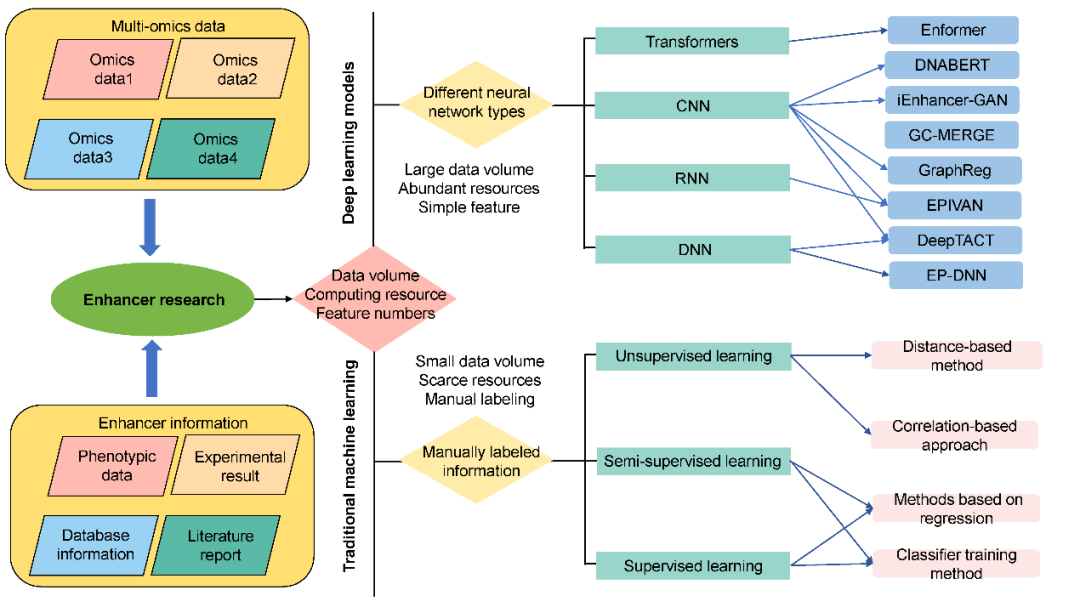
增强子研究中多组学整合方法分类
多组学整合技术遇到的挑战
目前多组学整合技术在增强子研究中面临5大挑战。不同组学数据之间的准确性差异、缺失值、模型性能的评价标准、模型可解释性、计算和储存成本。
1)来自不同来源的多组学数据信噪比和准确性存在差异。增加样本数量和改进实验设计可以提高不同组学分析的统计能力。然而根据MultiPower软件的统计,DNA-seq和ChIP-seq需要接近RNA-seq样本量的两倍以上才能达到相同的统计效果。此时可以考虑通过欠采样来平衡样本量或通过平衡偏差-方差权衡来评估机器学习的性能。
2)在测序过程中,实验随机误差或固有的技术缺陷会导致缺失值的出现。在分析过程中应重新评估多组学数据的分布特征,进行敏感性分析,以评估缺失值输入对下游分析的影响。对输入数据的验证是评估任何填补缺失值算法性能的重要步骤。
3)不同算法的侧重点完全不同,选择适合多组学分析的算法至关重要。
4)可解释性是指在一个系统中可以观察到因果关系的程度。影响模型可解释性的因素包括数据、模型架构和算法。人工标记数据,简化网络架构,采用可解释的人工智能架构,使用可解释的算法可以提高模型的可解释性。但过度追求可解释性可能会导致模型性能下降。
5)大多数集成算法需要高计算能力和相当大的存储容量来存储日志、结果和分析过程。高性能计算基础设施、云计算解决方案和先进的统计方法都是降低计算和存储成本的有效途径。
总结
增强子是基因转录的重要调控元件,组学技术的应用加快了对增强子在基因调控中的作用和机制的阐明。这篇综述讨论了四种组学技术(基因组学、表观基因组学、转录组学和CRISPR基因编辑)的应用和局限性。随着新的组学技术的发展,越来越多的高质量数据集的可用性,对多组学分析的需求持续增长。使用机器学习对多组学数据进行整合和分析,可以有效提高增强子预测模型的准确性。然而组学技术在增强子研究领域的应用仍然具有挑战性。尽管多组学数据存在广泛的异质性和质量差异,但随着测序技术的应用越来越广泛,数据的质量和数量都在不断提高,以应对当前的挑战。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


