Science:利用一种新的多尺度光催化邻近标记技术揭示细胞表面上和细胞之间的蛋白邻居
来源:生物谷原创 2024-08-06 11:14
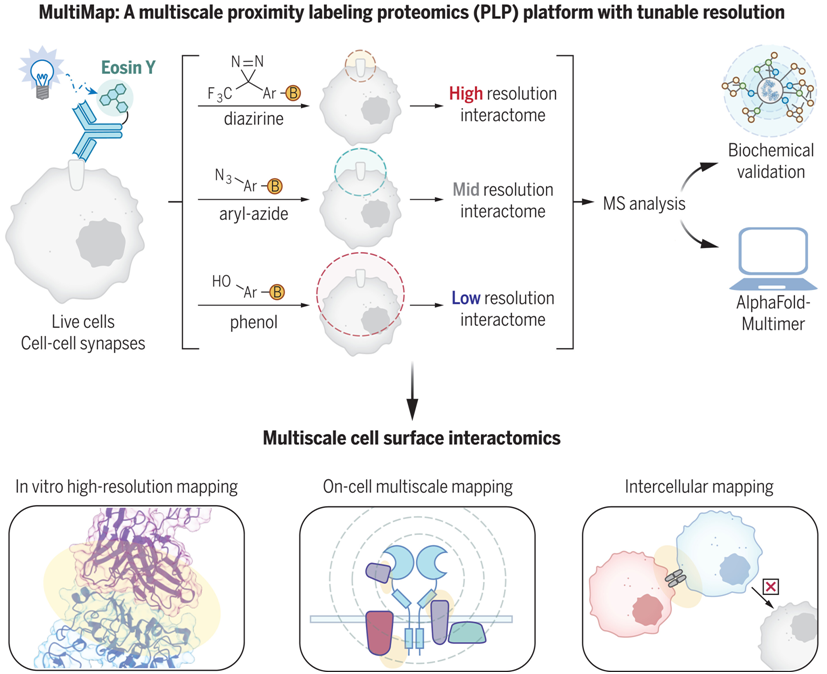
研究人员在一项新的研究中展示了一种名为 MultiMap 的新型 PLP 平台,它能以可调节的分辨率和集成的目标验证工作流程进行简便的相互作用组分析。

细胞膜蛋白质组是细胞通信的主要生物枢纽,然而人们对细胞表面上和细胞之间的动态蛋白邻居(protein neighborhoods)及其功能的了解才刚刚开始。近邻标记蛋白质组学(proximity labeling proteomics, PLP)是一种在原位鉴定瞬时蛋白相互作用组(protein interactome)的强大方法,对于治疗靶点发现和蛋白功能研究至关重要。第一代近邻标记方法(比如 APEX 和 BioID)使用酶催化,需要进行细胞工程。
由于所使用的探针的反应半衰期较长,它们提供的相互作用组图谱具有较宽的空间分辨率(高达 3000 Å)。近期开发的光催化近邻标记方法(如μMap)通过使用高活性的双吖丙啶探针(diazirine probe),提供了一种分辨率更高(约 100 Å)的替代策略。然而,目前的 PLP 方法仅限于基于所选探针及其标记半径的单一分辨率。没有一种方法能覆盖所有的标记长度尺度,因此,用户需要在这些近邻标记方法之间进行切换,以研究不同的生物系统。

为了以高置信度鉴定蛋白相互作用组,来自加州大学旧金山分校的研究人员在一项新的研究中展示了一种名为 MultiMap 的新型 PLP 平台,它能以可调节的分辨率和集成的目标验证工作流程进行简便的相互作用组分析。MultiMap 依靠发现的单一有机光催化剂 Eosin Y 激活所有主要类型的光学探针——双吖丙啶、芳基叠氮化物(aryl-azide)和苯酚,其中每种光学探针的标记半径各不相同。这种市场上可买到的光催化剂可以通过简便的生物偶联方法与感兴趣的抗体结合,使得人们能够捕捉和可视化观察蛋白的邻居,而无需在细胞表面上和细胞之间进行大量的蛋白工程实验。
相关研究结果发表在2024年7月19日的Science期刊上,论文标题为“Multiscale photocatalytic proximity labeling reveals cell surface neighbors on and between cells”。
MultiMap 使得这些作者能够探索肿瘤学的一个重要治疗节点——表皮生长因子受体 (EGFR) 的邻居。他们使用简单的两步生物偶联法将 Eosin Y 结合到抗体上,发现了 20 多个高置信度的分子邻居,其中大部分是以前未发现的。这些分子邻居包括一种磷酸酶、多种稳定蛋白和表皮生长因子受体底物,它们在功能上有关联,但在结构上没有显示出联系。
利用多尺度光催化邻近标记方法绘制的高分辨率相互作用组图谱
图片来自Science, 2024, doi:10.1126/science.adl5763
此外,通过使用作为综合邻近分析工作流程的一个新部分的AlphaFold-Multimer 进行二元相互作用的计算机预测,进一步提供了EGFR周围重要的有可能作为治疗靶点的分子邻居的可检验结构模型。他们还将 MultiMap 扩展到绘制细胞之间的邻居,展示了由包括双特异性 T 细胞接合子(BiTE)和嵌合抗原受体(CAR)在内的治疗模式诱导的四种不同的细胞-细胞突触。他们证实,MultiMap 能以高选择性标记相互接触的细胞和蛋白邻居,可用于不同的突触发现。
总之,这些作者开发了一种多尺度 PLP 方法,用于细胞表面和细胞间的相互作用组分子。在这些不同的生物场景中,MultiMap 是一个用途广泛、适应性强的相互作用组研究平台。它使用一种光催化剂、一个工作流程和最少的蛋白工程,提供了大量高分辨率和低分辨率的邻近信息。MultiMap 大大简化了光催化 PLP 工作流程。他们设想这一平台将加速未来的相互作用组研究,让科学家们更好地了解细胞膜上的水平信号传递。(生物谷Bioon.com)
参考资料:
Zhi Lin et al. Multiscale photocatalytic proximity labeling reveals cell surface neighbors on and between cells. Science, 2024, doi:10.1126/science.adl5763.
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


