Nature子刊:清华大学曾坚阳团队开发预测T细胞受体和抗原表位互作的AI模型
来源:生物世界 2023-04-10 10:47
清华大学交叉信息研究院曾坚阳团队开发了TEIM模型,通过小样本学习(Few-shot learning)的思想来准确预测TCR-epitope在残基水平的相互作用和结合信息。
T细胞受体和抗原表位的相互作用是机体免疫系统的重要组成部分,这种相互作用可以激活T细胞,从而识别和攻击感染机体的病原体。因此,研究T细胞受体和抗原表位的结合位点是理解机体免疫系统如何工作的关键。
近日,清华大学交叉信息研究院曾坚阳研究组在 Nature 子刊 Nature Machine Intelligence 上发表了题为:Characterizing the interaction conformation between T cell receptors and epitopes with deep learning 的研究论文。该研究成功开发了能预测T细胞受体和抗原表位相互作用的深度学习模型。

T细胞在适应性免疫反应中对表位的识别是至关重要的。这种识别是通过主要组织相容性复合体(MHC)分子呈递的致病抗原(epitope)与T细胞受体(TCR)相互作用来实现的,从而激活细胞介导的免疫,消除感染细胞或活化相应的免疫细胞。
因此,深入了解TCR-epitope的结合机制对于癌症免疫学、自身免疫抗原发现和疫苗设计具有重要意义。然而,由于这种识别机制的内在复杂性,TCR-epitope 相互作用的实验检测和确定通常既费时又昂贵。
为了解决这一问题,清华大学交叉信息研究院曾坚阳团队开发了TEIM模型,通过小样本学习(Few-shot learning)的思想来准确预测TCR-epitope在残基水平的相互作用和结合信息。模型首先在粗粒度的TCR-epitope序列结合信息上进行预训练,然后在细粒度的残基相互作用信息上进行进一步微调。验证试验和分析表明,TEIM模型具有优良的预测性能。
进一步,通过三个应用场景:突变序列的结合构象预测、TCR库的结合模式分析和结合机制的挖掘,研究团队进一步说明了TEIM模型可以帮助研究者更全面和深入地解析T细胞受体和抗原表位的相互作用。
该研究有效使用深度学习框架建模多层级的T细胞受体和抗原表位的相互作用,并且融合了大规模预训练思想和少样本学习策略,为揭示T细胞识别表位这一生物问题提供了有效工具。
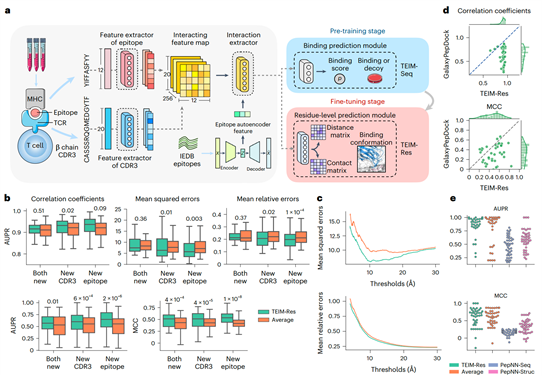
图1. TEIM模型结构和评估效果 (a) TEIM-Seq 和 TEIM-Res 的模型结构以及训练流程。 除了最后一个模块外,TEIM-Seq 和 TEIM-Res 模型共享相似的架构。 它们都有特征提取器来分别学习 CDR3 和抗原表位的序列特征,然后将它们扩展到不同的维度以形成交互特征图。 接下来,主要由二维卷积神经网络(2D CNN)组成的交互提取器用于提取成对残基交互信息。 此外,由自动编码器生成的表位特征向量被馈送到交互提取器中以获取全局表位信息。 最后,TEIM-Seq 使用结合预测模块聚合所有成对交互来预测结合得分(即结合概率),而 TEIM-Res 使用由 2D CNN 层组成的残基级预测模块来预测距离矩阵和接触矩阵。 训练流程包括两个阶段:首先在序列级结合数据上预训练 TEIM-Seq,然后在残基粒度的结合数据上微调 TEIM-Res。 (b) TEIM-Res 和其它基线方法相比,在三种不同数据划分设置下的性能表现。(c) TEIM-Res 和平均基线的不同距离阈值内残基对的均方/相对误差。 (d) GalaxyPepDock 和 TEIM-Res 之间的性能比较。 (e) TEIM-Res、平均基线和 PepNN 在预测结合矩阵任务上的性能比较。
清华大学交叉信息研究院曾坚阳副教授和赵诞助理研究员为该论文共同通讯作者,清华大学交叉信息研究院的硕士毕业生彭鑫港(现为北京大学智能学院和人工智能研究院博士生)和2021级博士研究生雷逸品为论文共同第一作者。该研究由国家自然科学基金杰出青年项目、青年基金项目、科技部重点研发项目以及南京图灵人工智能研究院支持。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


