Science:张锋一次性发现了188种新型CRISPR-Cas系统
来源:Brainology 2023-11-27 09:35
这项研究也提示我们,该研究发现的CRISPR相关系统代表着与RNA指导机制相关的多样化生化活动的未开发宝库,具有巨大的生物技术发展潜力。
CRISPR基因编辑先驱张锋在 Science 发表了题为:Uncovering the functional diversity of rare CRISPR-Cas systems with deep terascale clustering 的研究论文。
该研究开发了一种新的搜索算法——基于快速局部敏感哈希聚类算法(FLSHclust),使用该算法对三个主要的公共数据库进行挖掘,这些数据库包含各种不同寻常的细菌的数据(包括在煤矿、啤酒厂、南极湖泊和狗唾液中发现的细菌),从中识别出了188种新型CRISPR系统,并对其中4个系统进行了详细表征,这些新系统可能被用来编辑哺乳动物细胞,其脱靶效应比目前的CRISPR-Cas9系统要少,也有可能在被用于诊断或用来记录细胞内部活动。
这项研究凸显了CRISPR前所未有的多样性和灵活性,也表明了大多数CRISPR系统是罕见的,只在不寻常的细菌和古细菌中发现。随着可用来搜索数据库的不断增长,可能还有更多罕见系统被发现。

张锋教授表示,生物多样性是一个宝库,随着对更多的基因组和宏基因组样本进行测序,我们对FLSHclust等更好的工具的需求越来越大,以搜索序列空间来寻找“分子宝石”。
搜索CRISPR
CRISPR,即规律间隔成簇短回文重复序列(Clustered Regularly Interspaced Short Palindromic Repeats),是一种细菌和古菌的防御系统,已被设计成多种用于基因组编辑和诊断的工具。
为了从蛋白质和核酸序列数据库挖掘新型CRISPR系统,研究团队开发了一种基于局部敏感哈希(locality-sensitive hashing,LSH)的算法,将相似但并不完全相同的对象聚类在一起。使用这种方法,可以在几周时间内探测到数十亿个蛋白质和DNA序列,而以前寻找相同对象的方法需要数月时间。研究团队进一步设计了自己的算法——基于快速局部敏感哈希聚类算法(FLSHclust),来搜索与CRISPR相关基因。
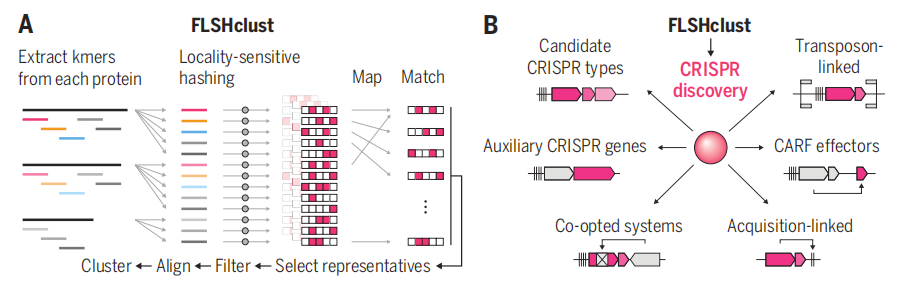
发现新系统
使用FLSHclust算法,他们发现的数千个CRISPR系统,可以分为几个现有类别以及许多新的类别。他们在实验室中更详细地研究了其中4个新系统。

该研究鉴定了一种IV型CRISPR系统——DinG-HNH,该系统具有插入到CRISPR相关DNA损伤诱导基因G(DinG)样螺旋酶中的HNH核酸酶结构域。该系统表现出RNA引导的PAM序列依赖的定向双链DNA(dsDNA)降解,这需要ATP水解和DinG-HNH蛋白的HNH核酸酶功能。 这也是首次证明具有特定干扰机制的IV型系统。
该研究还鉴定了两种I型CRISPR系统——Cas8-HNH和Cas5-HNH,它们含有插入在Cascade的不同亚基中的HNH核酸酶结构域。这两个系统都进行精确的双链DNA切割和单链DNA切割。并进一步证明了这两种系统都可以应用于人类细胞的基因组编辑,并且该系统具有高度特异性,它们使用一个长达32个碱基对的gRNA,而不是像Cas9那样的20个核苷酸的gRNA。由于这些I型CRISPR系统的gRNA更长,因此可被用于开发更精准基因编辑技术,从而减少脱靶编辑的可能性。由于这两个系统与CRISPR-Cas9的大小相似,它们可以被现有的基因递送技术递送到动物或人类细胞中。
该研究还观察到Cas5-HNH系统对单链DNA具有旁系切隔活性(Collateral Activity)。也就是该系统可以切割目标位点附近的DNA序列。张锋团队此前使用具有这一特点的CRISPR-Cas13开发了SHERLOCK系统,能够通过快速感应单个核酸分子来实现病原体的快速灵敏检测。研究团队认为,该系统也可以用于开发类似的诊断技术。
研究团队还探索了候选的VII型CRISPR系统,包括一个最小的Cas7-Cas5效应复合物,以及一个包括β-CASP结构域的独特干扰蛋白,这些系统可能来源于III-E型CRISPR系统,并且是RNA靶向的,因此可能用于RNA编辑。
挖掘数据
研究团队表示,该研究开发的FLSHclust算法在大型序列数据库挖掘中具有广泛应用前景,除了CRISPR系统,还可以帮助搜寻其他生化系统。任何人都可以使用这种搜索算法来研究蛋白质如何进化或发现新的基因。
这项研究也提示我们,该研究发现的CRISPR相关系统代表着与RNA指导机制相关的多样化生化活动的未开发宝库,具有巨大的生物技术发展潜力。
这项研究不仅说明了CRISPR系统的多样性,而且大多数都是罕见的,只在不寻常的细菌中才能被发现。例如,其中一些系统只在生活在煤矿水中的细菌中存在。如果不是使用这种算法,我们可能永远不会看到这些系统,而拓宽取样多样性对继续扩大我们所能发现的CRISPR系统多样性很重要。
值得一提的是,2023年6月27日,中国科学院遗传与发育生物学研究所高彩霞研究组在 Cell 期刊发表了题为:Discovery of deaminase functions by structure-based protein clustering 的研究论文。
该研究创新性地运用人工智能(AI)辅助的大规模蛋白结构预测,建立起全新的基于三级结构的高通量蛋白聚类分析方法,实现了脱氨酶功能结构的深入挖掘,鉴定到完全区别于已知脱氨工具酶的全新底盘元件,并成功开发了一系列具有我国自主知识产权的新型碱基编辑工具。

版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


