Genome Biology:基于零膨胀概率主成分分析模型的微生物组数据去噪新方法mbDenoise
来源:上海交大 2022-04-29 09:07
该研究提出基于零膨胀概率主成分分析的统计模型和变分近似算法对微生物组数据进行去噪,对揭示微生物组数据潜在的生物学信号有重要意义。
近日,国际权威期刊《Genome Biology》在线发表了上海交通大学生命科学技术学院王涛团队的研究成果“mbDenoise: microbiome data denoising using zero-inflated probabilistic principal components analysis”。该研究提出基于零膨胀概率主成分分析的统计模型和变分近似算法对微生物组数据进行去噪,对揭示微生物组数据潜在的生物学信号有重要意义。生命科学技术学院博士生曾燕燕为第一作者,生命科学技术学院王涛教授和美国耶鲁大学赵宏宇教授为通讯作者,生命科学技术学院李婧教授和韦朝春教授为合作者。
微生物组学是综合研究群居在某个生态系统的微生物群落,依托自身丰富的遗传及代谢功能,与特定环境中的其他生命体或环境因子发生互利或相克作用的新兴前沿学科。微生物组研究已积累了海量的测序数据,正面临着从数量到质量、从结构到功能研究的关键转变过程。但是,微生物组数据分析仍然面临许多技术挑战。主要挑战之一是物种或基因特征表包含大量的零,其中一些是生物零,而另外一些是非生物零。此外,微生物组数据还存在测序深度不均匀、过度离散和数据冗余等问题。这些干扰因素会引入大量噪声,如果不加以解决,会直接影响下游数据分析的准确性。
为了解决微生物组数据中的干扰因素并将真实的生物信号提取出来,该研究开发了mbDenoise,一种用于去除微生物组数据噪声的潜变量建模方法。该方法基于一个概率生成模型,在样本和物种或基因之间借用信息,从而实现生物信号与技术噪声分离(图1)。物种或基因丰度数据由零膨胀负二项模型生成,用于克服数据的过度离散和稀疏性问题,并区分生物零和非生物零。在此基础上,通过在负二项分布引入样本特异性效应,用于减轻测序深度差异导致的技术偏差。进一步地,假设生物信号对应嵌入高维特征空间的低维潜在表示,不仅刻画了微生物组数据的冗余性,而且克服了高维度和特征之间相互关系复杂的问题。该研究将上述生成模型称为零膨胀概率主成分分析(ZIPPCA)。mbDenoise通过变分近似算法拟合该模型,然后利用后验均值恢复真实丰度矩阵,实现数据去噪目标。
该研究使用大量模拟实验和真实数据广泛评估了mbDenoise的性能。整体来说,mbDenoise在参数估计、潜变量预测以及微生物群落组成估计方面具有较高的准确性。同时,在多样性分析和差异丰度分析等下游分析中,mbDenoise与其他方法相比表现更为优越。该研究提供的R软件包可在https://github.com/YanyZeng/mbDenoise获取。
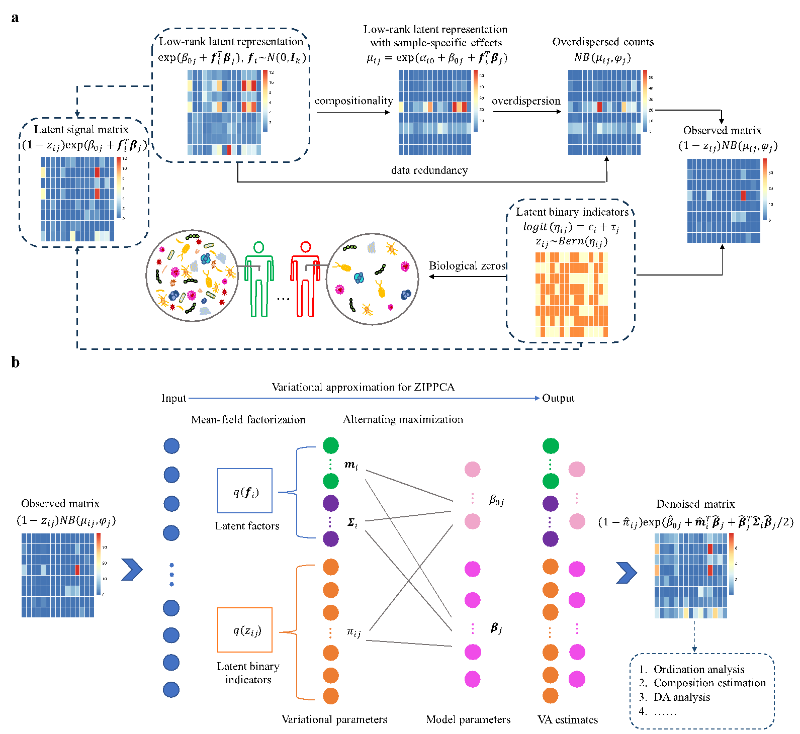
图1.mbDenoise及其噪声模型的概述
a.mbDenoise区分了生物零和非生物零,并假设生物信号对应嵌入高维特征空间的低维潜在表示,从而体现微生物组数据的冗余性。该方法基于零膨胀概率主成分分析(ZIPPCA)模型,考虑了微生物群落物种或基因丰度数据的成分性、稀疏性和过度离散等特征。b.假设输入数据(即丰度矩阵或特征表)是来自ZIPPCA模型的样本。mbDenoise通过变分近似算法拟合该模型,并将潜在信号矩阵的后验均值估计作为去噪输出,可以用于多个下游分析任务。
上海交通大学王涛团队研究方向为生物统计和高维数据统计推断。近5年来,发表微生物组数据挖掘与统计分析方法学论文10余篇,包括Journal of the American Statistical Association (2022, 2017), Annals of Applied Statistics (2017), Biometrics (2020, 2019, 2017), Briefings in Bioinformatics (2022), Bioinformatics (2022, 2021)。主要成果涉及微生物组数据预处理、数据降维与可视化、多元统计建模与分析、关联分析、整合进化树结构的预测建模与分析等。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


