肝脏芯片新进展!Anal Chem:开发出一种创新的堆叠阵列芯片,能成功模拟人体内的复杂炎症微环境
来源:生物谷原创 2025-01-30 13:27
本研究成功在堆叠阵列芯片上构建iDILI模型,可有效区分致特发性与固有性肝损伤的药物。以尼美舒利为例探究机制,发现其通过激活TNF通路、促进巨噬细胞M1极化等引发肝损伤,且炎症与药物处理存在动态平衡。

在药物研发和临床治疗领域,药物性肝损伤(DILI)一直是个令人头疼的问题。其中,特发性药物性肝损伤(iDILI)由于发病机制难以捉摸,缺乏明确的剂量-反应关系,不仅威胁患者健康,也给科研和医疗工作带来巨大挑战。
近期,一项发表在Anal Chem的研究Establishment of an Idiosyncratic Drug-Induced Liver Injury Model on a Stacked Array Chip for Identification of CCL5-Mediated Paracrine Dynamics为我们带来了新的解决方案。

以往研究iDILI,主要依赖动物模型和体外细胞模型。动物模型存在药物消耗量大、物种差异等问题,限制了高通量药物筛选以及对分子和细胞层面机制的深入探究。体外细胞模型,如二维细胞培养模型,无法模拟体内复杂的炎症微环境,难以真实反映iDILI的发病过程。尽管器官芯片技术逐渐兴起,但此前的芯片在灵活性和兼容性方面存在不足,且一直没有与动态炎症相关的iDILI芯片模型。
本研究研发出一种堆叠阵列芯片,它由上层的肝脏芯片和下层的巨噬细胞芯片组成。研究人员先将肝细胞与特定混合液滴在肝脏芯片的微柱阵列上,形成3D肝脏微组织。同时,对巨噬细胞进行预处理后,接种到巨噬细胞芯片。然后将两者组装并共培养,设置不同处理组,成功构建了iDILI模型。该模型细胞活力接近100%,并且确定了诸如血清需求、细胞接种密度等优化参数,在多细胞系和原代细胞中都验证了其可行性。

图 1. 堆叠阵列芯片上iDILI模型的构建
为了验证模型的有效性,研究人员挑选了10种代表性药物,其中8种为iDILI药物,2种为固有DILI药物。实验结果显示,iDILI药物与脂多糖(LPS)共处理时,3D肝脏微组织的肝毒性明显高于单独给药组;而固有DILI药物共处理组与单独给药组相比,肝毒性没有显著增加,这表明该模型能够准确区分两类药物。
以尼美舒利(NIM)为例,研究人员进一步探究iDILI的发病机制。RNA测序和RT - qPCR分析发现,NIM与LPS共处理会激活TNF通路,引发肝脏微组织的炎症损伤。同时,CCL5等炎症因子水平上升,CCL5通过与CCR1、CCR3和CCR5受体结合,促使巨噬细胞向M1极化。此外,通过灵活操作芯片,研究人员发现NIM诱导iDILI的机制符合部分假设,炎症预激活和NIM预处理之间存在动态平衡,这种平衡影响着炎症相关基因的表达。
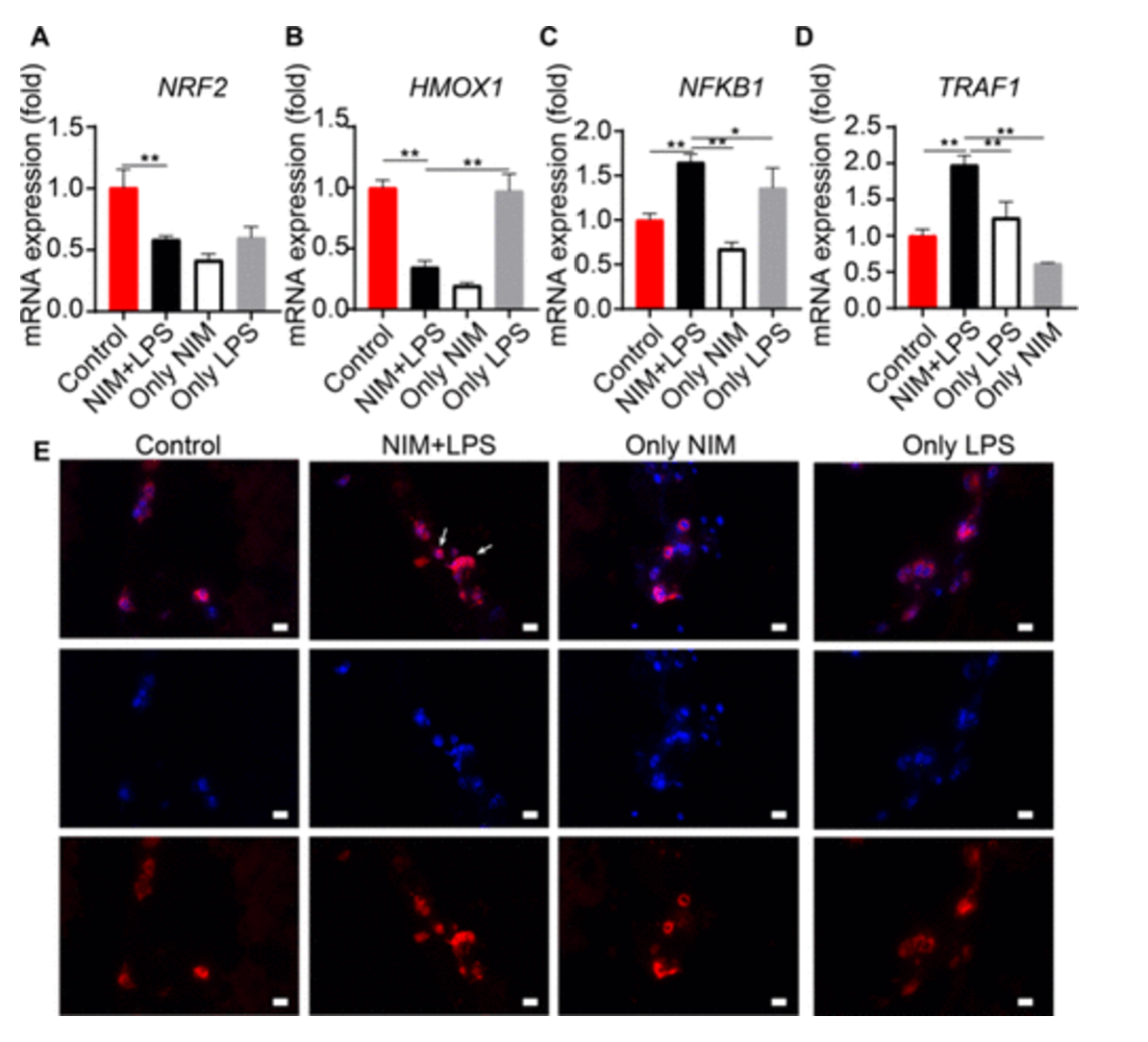
图 2. NIM诱导的iDILI促进3D肝脏微组织的炎症损伤
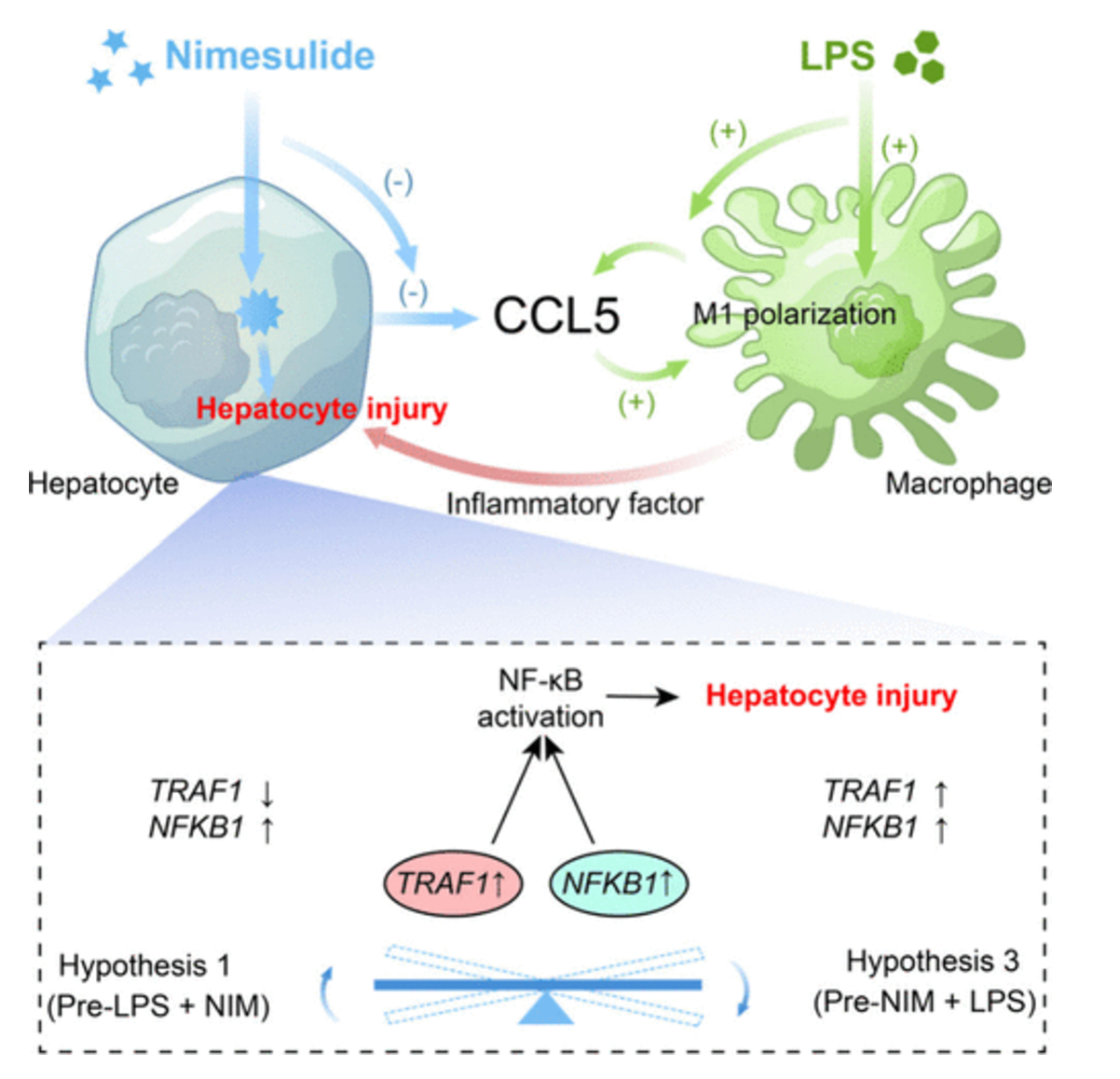
图 3. NIM诱导iDILI的机制示意图
这项研究构建的iDILI芯片模型具有重要意义。它能够模拟体内的动态微环境,准确区分iDILI药物,且芯片设计灵活,兼容性良好。通过该模型,我们对iDILI的发病机制有了更深入的理解,为iDILI药物筛选提供了有力工具,有望加速药物研发进程,降低临床用药风险。未来,随着科研的不断深入,通过整合患者来源细胞等方式,该模型的预测能力将进一步提升,为攻克iDILI带来更多希望,让更多患者受益于精准的医疗干预。(生物谷Bioon.com)
参考文献:
Hou Y, Gao Z, Tu P, Ai X, Jiang Y. Establishment of an Idiosyncratic Drug-Induced Liver Injury Model on a Stacked Array Chip for Identification of CCL5-Mediated Paracrine Dynamics. Anal Chem. 2024;96(49):19370-19377. doi:10.1021/acs.analchem.4c03552
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


