关于导向编辑与碱基编辑领域的系列突破性研究进展
来源:正序生物 2022-04-11 11:13
这项研究结果不仅为环形RNA的研究提供了新的思路,也拓展了碱基编辑器在生命科学研究领域的应用。
近年来,基因编辑技术发展迅速。基于CRISPR/Cas系统的新型基因编辑技术,因其高效、不产生DNA双链断裂、无需供体DNA等优势,正逐渐成为基因编辑领域的明星,在生物医药业、畜牧业和农业等领域均有广泛的应用场景。
目前,根据编辑原理的不同,新型编辑系统主要分为碱基编辑系统(Base Editor,BE)和导向编辑系统(Prime Editor,PE)两种。然而,PE目前能够介导的DNA插入片段较短、在很多疾病相关位点编辑效率很低、在人类细胞中是否会产生脱靶效应等问题制约着其在临床中的应用和推广,BE在更加广泛的研究领域内的创新性应用也有待探索。
为了开发更高效、安全、可靠的碱基基因编辑技术,上海科技大学生命科学与技术学院陈佳教授科研团队、复旦大学生物医学研究院杨力教授科研团队、武汉大学医学研究院殷昊教授科研团队、上海科技大学免疫化学研究所杨贝教授科研团队等进行了一系列研究,在检测现有导向编辑技术的安全性、提高导向编辑系统的编辑能效、扩大碱基编辑系统的运用范围等方面取得了突破性进展,将导向编辑在疾病相关靶点的编辑效率真正提高到可成药水平,为相关领域的临床应用提供了坚实的技术基础。
检测导向编辑系统的安全性
导向编辑系统(Prime Editor,PE)的工作原理是将CRISPR系统中的sgRNA改造成pegRNA(prime editing guide RNA),pegRNA不仅包含sgRNA中的靶向DNA结合区域,还携带有引物结合位点(primer binding site, PBS)区域和逆转录的模板序列(RTT, reverse transcription template),可以和逆转录酶(RT)相结合,实现将pegRNA中RTT的序列写入靶向位点的DNA序列中去。2019年,杨力教授、杨贝教授、陈佳教授在Cell期刊发表专评论文,介绍导向编辑系统的工作原理。

然而,PE系统的安全性目前还缺乏足够全面的证明。虽然已有报道表明PE系统只会产生少量的pegRNA依赖性的脱靶突变;但是其是否会在人类细胞中产生另外一种更为严重的、与pegRNA序列无关并难以预测的随机突变——pegRNA非依赖性的脱靶突变,还有待验证。
2022年3月14日,陈佳教授、杨力教授与上海科技大学生命科学与技术学院黄行许教授合作,在CRISPR Journal期刊在线发表了题为“Genomic and Transcriptomic Analyses of Prime Editing Guide RNA–Independent Off-Target Effects by Prime Editors”的研究论文。
该研究论文建立了一种可以在细胞水平检测和分析PE系统在全基因组和全转录组范围内脱靶效应的方法,并证实PE3逆转录酶部分在开展编辑过程中的高度特异性。

为了全面有效地评估PE的脱靶效应,科研人员首先在293FT细胞中构建了一种背景突变数量极低的细胞系293FTA3-/-,并分别用PE3、hA3A-BE3以及Cas9三种不同的编辑器对其进行编辑,之后检测这三种编辑器所编辑过的单克隆细胞中的突变数量。所有实验均在全基因组和转录组范围内进行。(图1A)
结合基因组和转录组数据,对端粒完整性、内源性逆转录作用元件的活性、可变剪接事件的发生以及基因表达模式改变等进行了全方位的比对和分析之后,科研人员发现,PE3在靶向位点处介导的高效编辑的同时(图1B),并未在全基因组(图1C)和全转录组(图1D)范围内检测到PE3产生的pegRNA非依赖性的脱靶效应。由此,证明了PE3系统的高特异性 (Gao et al, CRISPR J 2022)。
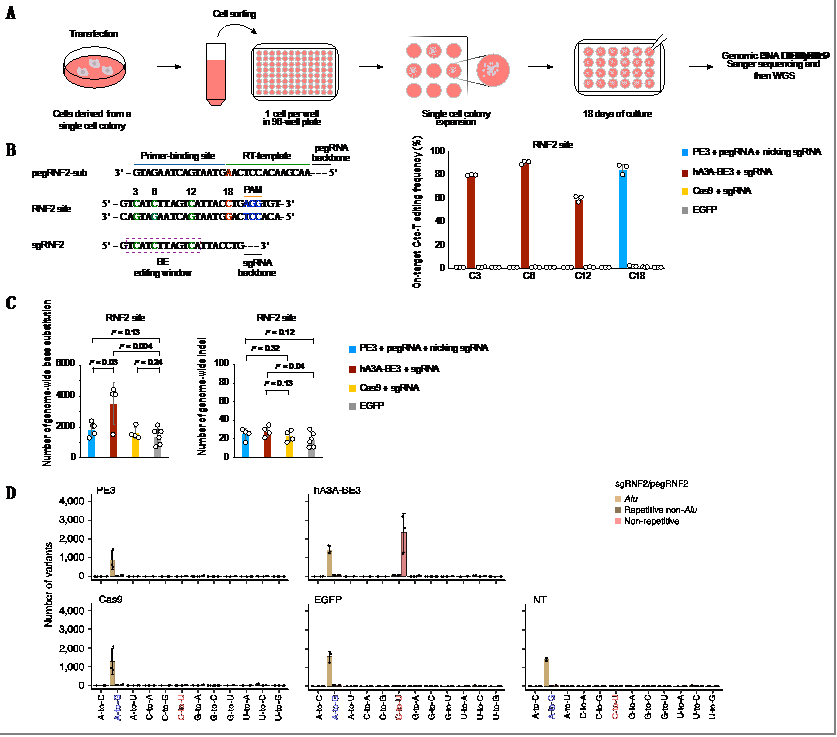
图1:全基因组和全转录组水平未检测到PE3存在pegRNA非依赖性脱靶突变
A)在细胞水平上通过检测编辑的单克隆细胞来分析PE是否存在脱靶突变方法的模式图。
B)PE3在靶向位点的编辑效率。
C)全基因组范围内未检测到PE3存在脱靶突变。
这些结果为利用PE3开展高精度的基因编辑应用提供了理论基础。
导向编辑系统的升级改造
为了提升PE系统的编辑能效,科研团队还进行了更深一步的探索。
2022年2月28日,武汉大学医学研究院殷昊教授和张楹教授在Nature Methods期刊在线发表了题为“Efficient targeted insertion of large DNA fragments without DNA donors”的最新研究进展,科研团队开发了一种新型导向编辑系统——GRAND editing,通过引入一对pegRNAs并对其进行巧妙设计,实现了无需DNA供体的情况下,在基因组DNA上精确地插入长序列片段的新策略。

该项研究利用一对特殊设计的pegRNAs构建了新型导向编辑系统——GRAND editing(genome editing by RTTs partially aligned to each other but nonhomologous to target sequences within duo pegRNA),以实现目的片段的靶向插入。插入片段长度由PE3介导的44 bp扩展到GRAND editing介导的1000 bp,大大扩展了导向编辑系统在进行靶向插入编辑时的应用用范围。
经过特殊设计的paired pegRNAs 的3’端携带的逆转录模板中含有一段碱基互补配对区域,并且两个RTTs和基因组的靶向位点均无任何同源序列。当RT酶通过pegRNA 3’末端的引物结合位点开始逆转录之后,产生两条末端互补配对的单链DNA,进而产生一段稳定的双链DNA结构。这样的编辑链在与原始的基因组链的竞争中具备优势,从而实现了外源片段的高效插入。同时,因为GRAND editing中的逆转录模板不需要与基因组同源配对,可以在flap长度相同的情况下插入更长的外源片段(图2)。
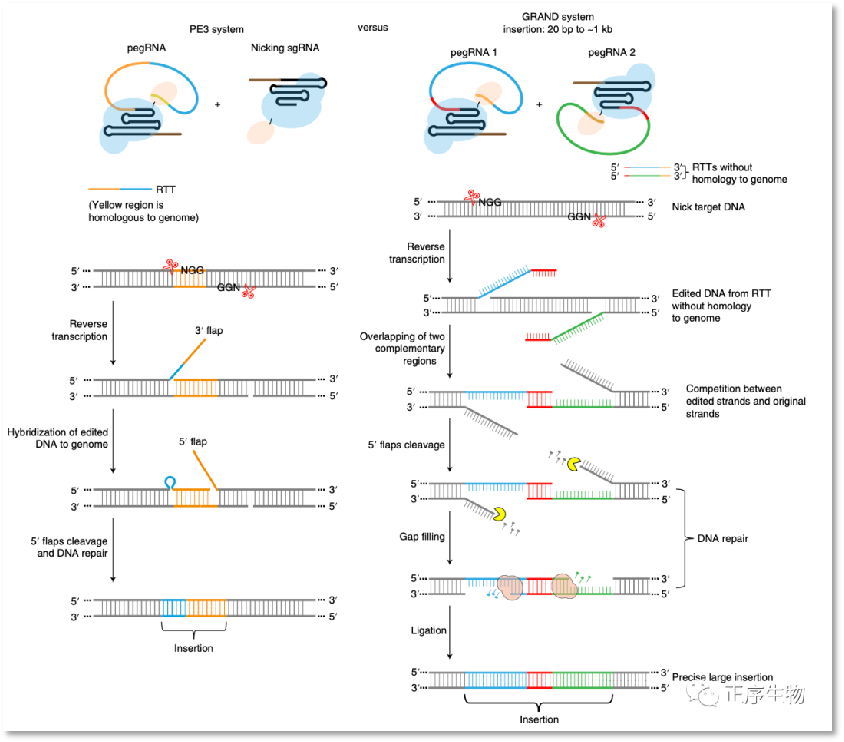
图2:PE3(左)和GRAND editing(右)靶向插入DNA片段的示意图
研究团队利用GRAND editing系统成功靶向插入bsd基因,并通过基因插入补全缺失部分基因片段的EGFP基因,在多个内源性位点上均实现了高效靶向插入DNA,并且其编辑的副产物占比极低。同时,证实了GRAND editing可以在多种细胞系的多个位点以及在非分裂细胞中均能实现长序列的精准插入。
GRAND editing系统为科研工作者进行基因功能相关的研究以及分子生物学的研究提供了新方法,也极大的拓展了遗传病以及罕见病的基因治疗手段和相关的药物以及治疗管线开发的思路。
除了增加PE系统在靶向位点插入DNA片段的长度,针对PE系统编辑效率极低的位点,研究团队也对PE系统进行了创新性升级。
2022年3月29日,陈佳教授、杨力教授与上海第一人民医院孙晓东教授合作在Nature Communications期刊在线发表了题为“Genomic and Transcriptomic Analyses of Prime Editing Guide RNA–Independent Off-Target Effects by Prime Editors”的研究论文,通过规律性引入同义突变和pegRNA骨架优化,开发了新型导向编辑系统sPE和aPE,优化的sPE系统将碱基转换效率平均提高353倍(最高至4976倍),优化的aPE系统将碱基插入/缺失效率平均提高了2.77倍(最高至10.6倍)。

针对PE介导的单碱基转换,科研人员通过在pegRNA的逆转录模板中规律性的引入同义突变,开发了spegRNA,并与PE3系统整合构建了sPE3。研究发现,当引入1到2个同义突变,且位于1,5,6或同时位于2和5,3和6位时,可以获得效率最高的spegRNA。经过优化的spegRNA可将碱基转换效率平均提高353倍(最高至4976倍),并在原始版本的PE3无明显编辑(编辑效率低于3%)位点处将编辑效率提高至40%到80%(图3)。
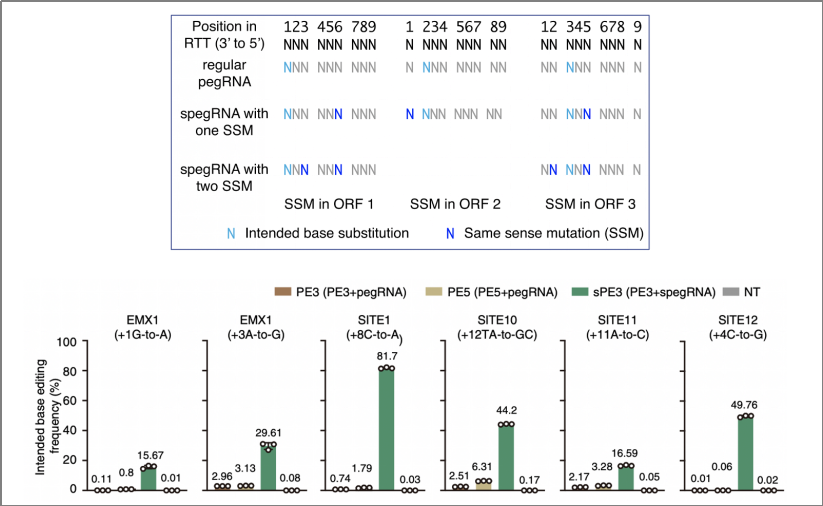
图3:spegRNA可介导更高效率的碱基转换
针对PE3介导的碱基精确插入/缺失,科研人员在分析了pegRNA骨架之后,将发夹结构中G/A改造为C/G碱基对,以提高pegRNA稳定性,从而建立了apegRNA,并将其与PE3系统整合构建了aPE3。经过检测,优化的apegRNA将PE3介导的碱基插入/缺失效率平均提高了2.77倍(最高至10.6倍)(图4)。

图4:apegRNA能够介导更高效率的碱基精确插入/缺失
同时,spegRNA和apegRNA联合使用可进一步提高碱基转换和插入/缺失效率,spegRNA和apegRNA也能够与PE5系统整合(sPE5与aPE5)并提高PE5的编辑效率(Li et al, Nat Commun 2022)。
碱基编辑系统的创新性应用
除了在PE系统研发上的系列性成果,科研团队之前已深入研究并开发BE系统多年。BE系统主要由 sgRNA 和碱基编辑器两部分组成,其中碱基编辑器一般由改造的Cas9 蛋白(dCas9或nCas9)、胞嘧啶脱氨酶和尿嘧啶糖苷酶抑制子三者构成。sgRNA 通过与靶位点互补配对,引导碱基编辑器结合到靶位点发挥作用。
许多环状RNA(circRNAs)是由前体mRNAs外显子的反向剪接产生的,通常与同源线性RNA共同表达。由于基因之间存在序列重叠,目前还没有找到针对环状RNA特异性敲除的方法。
2022年1月10日,杨力教授、陈佳教授与中科院分子细胞卓越创新中心(原上海生化细胞所)陈玲玲研究员合作,在Genome Biology期刊在线发表了题为“Knockout of circRNAs by base editing back-splice sites of circularized exons”的研究进展,报道了利用hA3A-BE碱基编辑底层平台专利技术实现环形RNA在基因组水平敲除的新策略。

该研究成果的成功发表得益于科研团队在碱基编辑领域和环形RNA领域的独特优势,紧密结合各实验室的研究积累,基于反向剪接位点具有与经典剪接位点(5'-AGgt---agGT-3')一致的特征,利用碱基编辑器针对反向剪接位点开展单碱基突变的策略,从而实现了在基因组水平的环形RNA敲除新方法(图5)。
由于环形RNA与其同源线性mRNA在一级序列上的一致性,当使用CBE(实现反向互补链的C到T编辑)或ABE(编码链的A到G编辑)碱基编辑器改变这些共用剪接位点时,环形RNA及其对应的线性RNA同时被抑制,因此不能简单地利用碱基编辑器进行环形RNA的敲除。
而为了实现针对环形RNA的特异性敲除,应设计只针对在环形RNA中存在的外显子反向剪接位点进行突变。研究人员通过计算分析鉴定,发现了一类特殊的环形RNA新分子,其包含只在环形RNA中特异存在的外显子,并利用碱基编辑器对相应反向剪接位点进行突变,实现了针对环形RNA的特异敲除(图5)。利用这一方法,研究人员也在293FT中筛选发现了一个具有影响细胞增殖功能的环形RNA新分子。
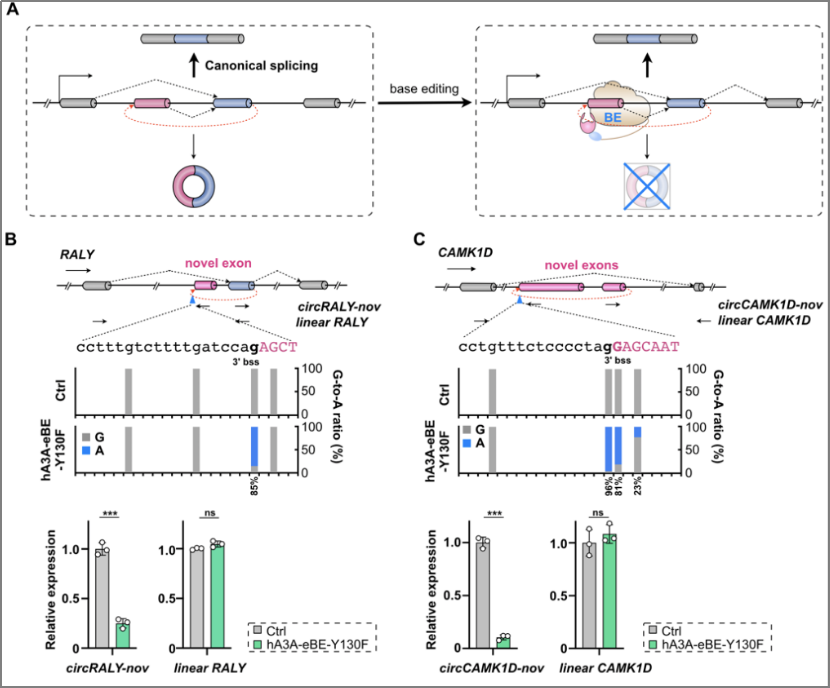
图5:通过编辑环形RNA特异存在的外显子的反向剪接位点敲除环形RNA
这项研究结果不仅为环形RNA的研究提供了新的思路,也拓展了碱基编辑器在生命科学研究领域的应用。
上海科技大学生命科学与技术学院陈佳教授、复旦大学生物医学研究院杨力教授、武汉大学医学研究院殷昊教授、上海科技大学免疫化学研究所杨贝教授,分别在基因编辑、DNA损伤修复、生物信息学、RNA生物学、药物递送、疾病动物模型、蛋白质工程等生物科技领域深耕多年。科研团队开发了全球先进的碱基编辑系统,并拥有覆盖全球15个国家和地区的10余项技术专利和靶点专利。目前,团队创建了多种基于新型碱基编辑系统的疗法,这些疗法相比传统基因编辑疗法更加安全、可靠、高效,尤其在控制脱靶效应的安全性和体内编辑效率方面具有显著优势。相关研究多次发表于Nature Biotechnology, Nature Cell Biology, Nature Methods, Nature Structural & Molecular Biology, Nature Biomedical Engineering, Nature Communications, Genome Biology等国际学术期刊,受到行业的广泛关注。
Cell论文链接:
https://doi.org/10.1016/j.cell.2019.11.030
CRISPR Journal论文链接:
https://www.liebertpub.com/doi/10.1089/crispr.2021.0080
Nature Methods论文链接:
https://www.nature.com/articles/s41592-022-01399-1
Nature Communications论文链接:
https://www.nature.com/articles/s41467-022-29339-9
Genome Biology论文链接:
https://genomebiology.biomedcentral.com/articles/10.1186/s13059-021-02563-0
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


