中山大学科研人员报道代谢组学AI分析新方法
来源:网络 2024-10-14 10:22
DeepMSProfiler利用来自多家医院的859份肺腺癌患者、良性肺结节患者和健康人群的人血清样本进行训练和测试,在独立测试集中成功甄别了不同组别的代谢组学特征,AUC值达0.99。
中山大学中山医学院李伟忠团队与中山大学肿瘤防治中心黄蓬、胡寓旻团队合作,在Nature Communications发表了题为An end-to-end deep learning method for mass spectrometry data analysis to reveal disease-specific metabolic profiles 的研究成果。该研究开创性地提出可解释性集成深度学习方法DeepMSProfiler,可端对端地进行非靶向代谢组学数据分析。
DeepMSProfiler方法能够直接将原始的LC-MS下机数据转换为样本所属疾病类别、疾病相关的关键代谢物和疾病相关的代谢物-蛋白质网络等结果,提供了全面的疾病相关代谢组图景。DeepMSProfiler无需使用传统的代谢组学分析过程中的峰提取和识别步骤,从而避免了复杂分析过程中的误差累积,更充分地利用了根据现有数据库无法鉴定出来的未知代谢信号。重要的是,DeepMSProfiler有效地克服了来自不同医院之间的批次效应问题。
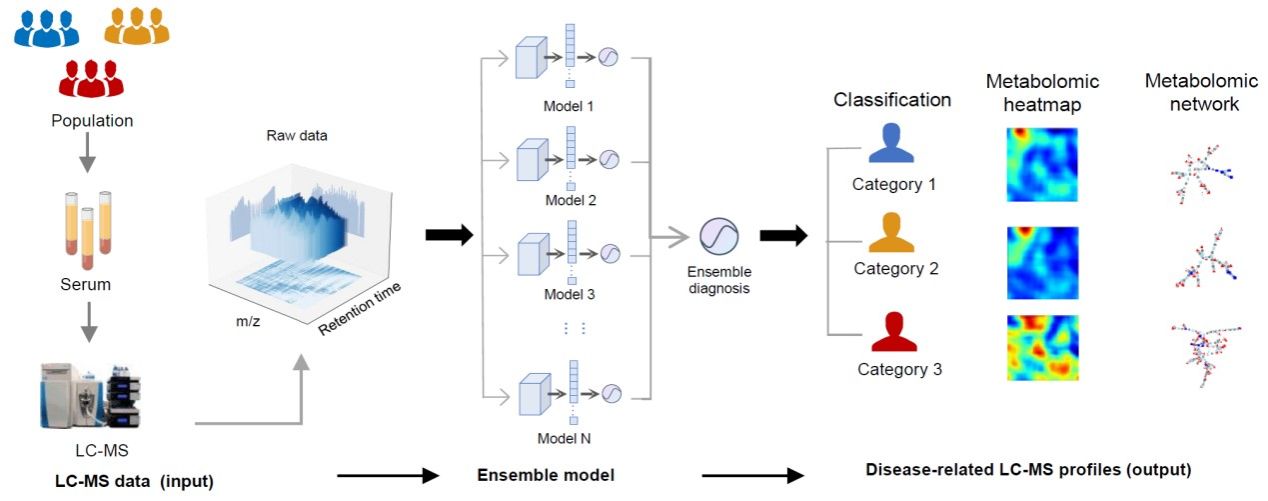
DeepMSProfiler利用来自多家医院的859份肺腺癌患者、良性肺结节患者和健康人群的人血清样本进行训练和测试,在独立测试集中成功甄别了不同组别的代谢组学特征,AUC值达0.99,并在早期肺腺癌诊断中实现了高达96.1% 的准确率。此外,该方法在来自928种细胞系的泛癌脂质代谢组学数据上进行了扩展分析,揭示了23种癌症类型共同相关的代谢物和蛋白质,展现了出色的泛化能力以及在多种疾病诊断中的应用潜力。
简而言之,该研究为代谢组学领域首个端对端分析方法,性能指标显著性超越传统方法,为基于代谢组学的疾病诊断和生物机制发现提供了一个直接且可靠的方法。
该研究得到了国家重点研发计划、广东省基础与应用基础研究基金、广州国家实验室重大项目等项目资助。中山医学院博士研究生邓永洁为该论文第一作者,中山医学院李伟忠教授与中山大学肿瘤防治中心黄蓬教授及胡寓旻研究员为该论文共同通讯作者。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


