全文编译!我国科学家发表Science论文,揭示SARS-CoV-2病毒RNA依赖性RNA聚合酶的三维结构,助力开发新的疫苗和药物
来源:本站原创 2020-04-23 13:18
2020年4月23日讯/生物谷BIOON/---由新型冠状病毒SARS-CoV-2(之前称为2019-nCoV)引起的2019年冠状病毒病(COVID-19)于2019年12月出现,此后成为全球大流行病。据报道,SARS-CoV-2是β冠状病毒(betacoronavirus)属的一个新成员,与严重急性呼吸综合征(SARS)冠状病毒(SARS-CoV)和几种
2020年4月23日讯/生物谷BIOON/---由新型冠状病毒SARS-CoV-2(之前称为2019-nCoV)引起的2019年冠状病毒病(COVID-19)于2019年12月出现,此后成为全球大流行病。据报道,SARS-CoV-2是β冠状病毒(betacoronavirus)属的一个新成员,与严重急性呼吸综合征(SARS)冠状病毒(SARS-CoV)和几种蝙蝠冠状病毒密切相关。与SARS-CoV和中东呼吸综合征(MERS)冠状病毒(MERS-CoV)相比,SARS-CoV-2表现出更快的人际传播,从而导致世界卫生组织(WHO)宣布为世界性的公共卫生紧急事件。
冠状病毒使用一种多亚基复制/转录复合物。作为病毒多聚蛋白(polyprotein)ORF1a和ORF1ab的裂解产物而产生的一组非结构蛋白(nsp)组装在一起以促进病毒复制和转录。其中的一个关键组分---RNA依赖性RNA聚合酶(RdRp, 也称为nsp12)---催化病毒RNA合成,因而在SARS-CoV-2的复制和转录周期中起着至关重要的作用,它在这个过程中可能需要作为辅因子的nsp7和nsp8的协助。因此,nsp12被认为是瑞德西韦(remdesivir, 也称为GS-5734)等核苷酸类似物抗病毒抑制剂的主要靶点,其中瑞德西韦已显示出治疗SARS-CoV-2感染的潜力。
为了指导药物设计,在一项新的研究中,来自中国清华大学、上海科技大学、南开大学、天津大学和中国科学院生物物理研究所的研究人员使用了两种不同的方案:一种不存在DTT(数据集1),另一种存在DTT(数据集2),并利用低温电镜(cryo-EM)技术解析出nsp12与它的辅因子nsp7和nsp8形成复合物时的三维结构。相关研究结果于2020年4月10日在线发表在Science期刊上,论文标题为“Structure of the RNA-dependent RNA polymerase from COVID-19 virus”。
细菌表达的全长SARS-CoV-2 nsp12(残基S1-Q932)与nsp7(残基S1-Q83)和nsp8(残基A1-Q198)孵育在一起,然后纯化出所形成的复合物。在这种复合物的存在下制备出低温电镜网格,初步筛选后发现这种网格具有良好的分散性和极佳的颗粒密度。在收集和处理7994个显微影片后,这些研究人员在2.9埃的分辨率下实现对nsp12单体分别与nsp12-nsp8二聚体和 nsp8单体形成的复合物的三维重建,正如之前在SARS-CoV中观察到的那样。除了nsp12-nsp7-nsp8复合物,他们还观察到了对应于nsp12-nsp8二聚体以及单个nsp12单体的单颗粒类型,但这些都没有给出原子分辨率下的三维重建。然而,nsp12-nsp7-nsp8复合物的三维重建提供了完整的结构分析信息。
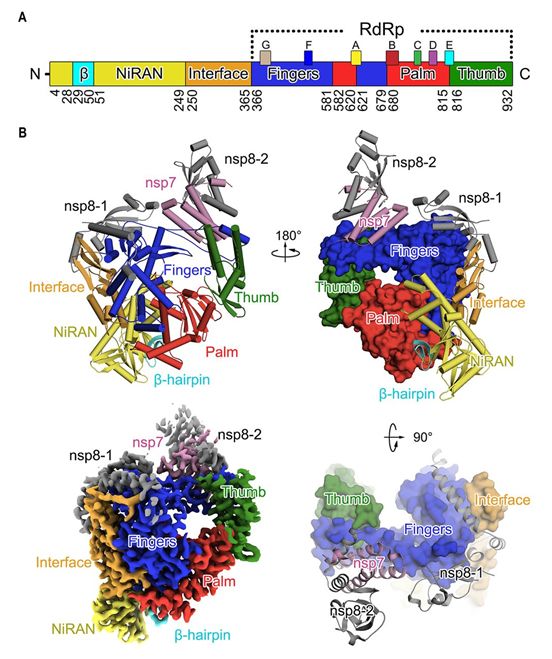
SARS-CoV-2 nsp12的结构包含一个“右手”RdRp结构域(残基S367-F920),以及一个套病毒(nidovirus)独特性的N端延伸结构域(残基D60-R249),其中这种延伸结构域采用了套病毒RdRp相关核苷酸转移酶(nidovirus RdRp-associated nucleotidyltransferase, NiRAN)结构。这种聚合酶结构域(即RdRp结构域)和NiRAN结构域由一个界面结构域(interface domain, 残基A250-R365)连接(图1A和图1B)。在清晰的低温电镜图的指导下构建出的一个额外的N端β发夹(D29-K50),插入到由RdRp结构域中的掌状亚结构域和NiRAN结构域夹持的凹槽中(图2)。这种nsp7-nsp8二聚体显示出类似于SARS-CoV nsp7-nsp8二聚体的保守结构。相比于nsp7-nsp8二聚体,与nsp12结合的nsp8单体的N端螺旋的定位发生了偏移。在nsp8的N端解析出的13个额外的氨基酸残基表明它的著名的 “高尔夫球杆(golf club)”形状的长“轴”是弯曲的。
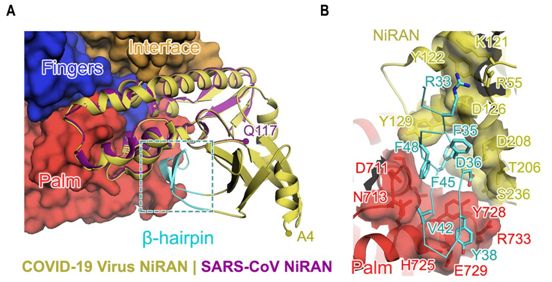
SARS-CoV-2 nsp12-nsp7-nsp8复合物的整体结构与SARS-CoV中的相似,1078个Cɑ原子的均方根偏差(root mean square deviation, rmsd)为0.82。然而,有区分这两者的关键特征。通过低温电镜图,这些研究人员得以构建出SARS-CoV-2 nsp12(包括除S1-D3和G897-D901以外的所有氨基酸残基)的完整结构。相比之下,SARS-CoV nsp12中的前116个氨基酸残基没有被解析出来。在SARS-CoV-2中解析出的NiRAN结构域的部分(残基4至28和51至249)由8个螺旋组成,在N端有一个五股β折叠(five-stranded β-sheet)(图2A)。在SARS-CoV-12病毒结构中,他们另外解析了残基A4-R118。这些构成了具有5个反平行β链和两个螺旋的结构体。残基N215-D218在SARS-CoV-2病毒nsp12中形成了一个β链,而这些残基在SARS-CoV nsp12中不那么有序排列。这个区域与包括残基V96-A100的链接触,从而有助于稳定其构象。因此,这四条链形成了一个紧凑的半β桶结构。因此,他们将残基A4-T28和Y69-R249确定为完整的冠状病毒NiRAN结构域。通过对N末端残基的解析,他们还能够确定一个N末端的β发夹(D29至K50,图1A和2A)。这个β发夹插入到由RdRp结构域中的掌状亚结构域和NiRAN结构域夹持的凹槽中,并形成一组紧密接触,以稳定整体结构(图2B)。另一点需要注意的是,他们观察到C301-C306和C487-C645在没有DTT的情况下形成二硫键。然而,在DTT存在的情况下,螯合锌离子存在,并且与在SARS-CoV中观察到的位置相同。
这种聚合酶结构域采用病毒聚合酶家族的保守结构,由三个亚结构域组成:手指亚结构域(残基L366-A581和K621-G679)、掌状亚结构域(残基T582-P620和T680-Q815)和拇指亚结构域(残基H816-E920)(图1)。这些催化性的金属离子在合成RNA的病毒聚合酶的几个结构中观察到,不过在引物-模板RNA和核苷酸三磷酸(NTP)不存在的情形下是观察不到的。
SARS-CoV-2病毒RdRp结构域的活性位点由掌状亚结构域中保守的聚合酶基序A-G形成,并且它的构型类似于其他的RNA聚合酶(图1A和3A)。由氨基酸残基611-TPHLMGWDYPKCDRAM-626组成的基序A包含经典的二价阳离子结合残基D618,这在大多数病毒聚合酶中是保守的,包括HCV ns5b(残基D220)和脊髓灰质炎病毒3Dpol(残基D233)(图3,B和C)和基序C(残基753-FSMMILSDDAVVCFN-767)包含在两个β链之间的转弯处的催化残基(759-SDD-761)。这些催化残基在大多数病毒RdRp---比如HCV ns5b中的317-GD-319和脊髓灰质炎病毒3Dpol中的327-GD-329---中也是保守的:第一个残基是丝氨酸或甘氨酸。
在这种结构中,与其他RNA聚合酶一样,模板/引物进入通道、NTP进入通道和新生链出口通道都带正电荷,是溶剂可接触到的,并汇集在一个中央空腔中,在这个中央空腔中,这些RdRp基序介导模板引导的RNA合成(图3D)。模板/引物进入通道、NTP进入通道和新生链出口通道的配置与SARS-CoV中的相类似,也与HCV聚合酶和脊髓灰质炎病毒聚合酶等其他RNA聚合酶相类似(图3,B和C)。NTP进入通道由一组亲水性残基形成,包括基序F中的K545、R553和R555。据预计RNA模板通过由基序F和G夹持的凹槽进入由基序A和C组成的活性位点。基序E和掌状亚结构域支持着引物链。产物-模板杂交体通过RNA聚合酶前侧的RNA出口通道退出这个活性位点。

作为2-乙基丁基L-丙氨酸磷酰胺(2-ethylbutyl L-alaninate phosphoramidate)前体药物的Sp异构体,瑞德西韦(remdesivir, 也称为GS-5734)已被报道能够抑制SARS-CoV-2病毒增殖,因此具有临床潜力。这些研究人员将根据这项研究的结果简要地讨论它的潜在结合和抑制机制。链终止核苷酸类似物的疗效需要病毒RdRp来识别并成功地将它们的活性形式整合到生长中的RNA链上。索非布韦(sofosbuvir),即2'-F-2'-C-甲基尿苷单磷酸酯(2'-F-2'-C-methyluridine monophosphate),是一种靶向HCV ns5b的前体药物,已被批准用于治疗慢性HCV感染。它通过结合HCV ns5b聚合酶的催化位点而发挥作用。
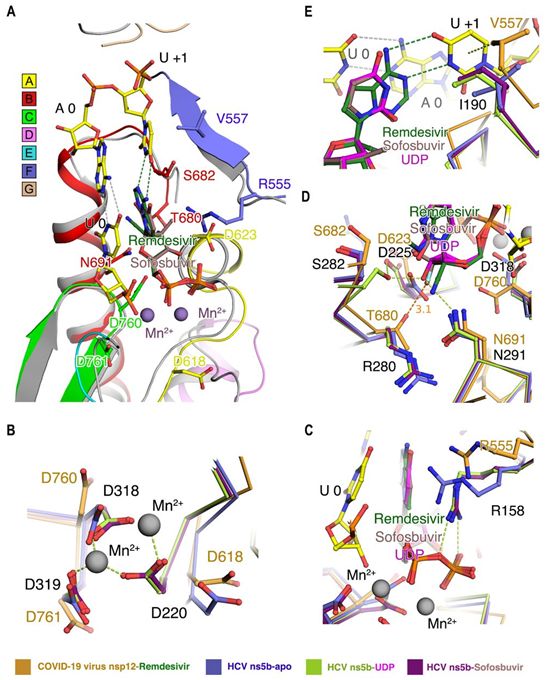
鉴于瑞德西韦和索非布韦都是核苷酸类似物,以及SARS-CoV-2 nsp12和HCV ns5b聚合酶的催化位点在结构上的保守性,他们基于与结合到HCV ns5b上的索非布韦的叠加,对二磷酸瑞德西韦(remdesivir diphosphate)与SARS-CoV-2 nsp12之间的结合进行了建模(图4A)。总的来说,他们发现SARS-CoV-2 nsp12与ns5b的空载状态(Apo state)的相似度最高。鉴于ns5b在空载/延伸/抑制状态下的构象变化,这似乎表明催化残基D760、D761和典型的D618将发生构象变化,以便与这些二价阳离子形成配位键(图4B)。后者将与基序F中的变构残基R555一起锚定进入的核苷酸或抑制剂的磷酸基团(图4C)。在HCV ns5b延伸复合物或它与二磷酸索非布韦(pp-sofosbuvir)形成的复合物的结构中,一个关键的特征是整合到RNA链上的二磷酸索非布韦与N291(相当于SARS-CoV-2中的N691)相互作用,但由于它的糖基上发生氟取代,它不能与S282和D225连接成氢键网络(图4D),不过这是让进入的天然核苷酸保持稳定所必需的。然而,瑞德西韦保留了一个完整的核糖基团,所以它可能能够像天然底物一样利用这个氢键网络。此外,SARS-CoV-2 nsp12中的T680也可能与瑞德西韦的2'羟基形成氢键,当然也可能与进入的天然NTP形成氢键(图4D)。此外,基序F中的V557的疏水侧链很可能让+1模板RNA碱基尿苷保持稳定,使得尿苷与进入的三磷酸瑞德西韦(ppp-remdesivir)进行碱基配对(图4E)。
SARS-CoV-2在全球范围内的迅速传播突显了开发新的冠状病毒疫苗和药物的必要性。SARS-CoV-2病毒聚合酶nsp12看起来是开发新药物的一个很好的靶点,特别是考虑到瑞德西韦等先导抑制剂已经以化合物的形式存在。考虑到核苷类似物的结构相似性,这项研究中讨论的结合模式和抑制机制也可能适用于其他的此类药物或候选药物,包括在临床试验中证明有效的法匹拉韦(Favipiravir)。除了其他的有前景的药物靶标(比如SARS-CoV-2的主蛋白酶)之外,这个靶点还可以潜在地支持开发抗冠状病毒混合物,从而潜在地可用于发现广谱抗病毒药物。(生物谷 Bioon.com)
参考资料:
Yan Gao et al. Structure of the RNA-dependent RNA polymerase from COVID-19 virus. Science, Published online: 10 Apr 2020, doi:10.1126/science.abb7498.
冠状病毒使用一种多亚基复制/转录复合物。作为病毒多聚蛋白(polyprotein)ORF1a和ORF1ab的裂解产物而产生的一组非结构蛋白(nsp)组装在一起以促进病毒复制和转录。其中的一个关键组分---RNA依赖性RNA聚合酶(RdRp, 也称为nsp12)---催化病毒RNA合成,因而在SARS-CoV-2的复制和转录周期中起着至关重要的作用,它在这个过程中可能需要作为辅因子的nsp7和nsp8的协助。因此,nsp12被认为是瑞德西韦(remdesivir, 也称为GS-5734)等核苷酸类似物抗病毒抑制剂的主要靶点,其中瑞德西韦已显示出治疗SARS-CoV-2感染的潜力。
为了指导药物设计,在一项新的研究中,来自中国清华大学、上海科技大学、南开大学、天津大学和中国科学院生物物理研究所的研究人员使用了两种不同的方案:一种不存在DTT(数据集1),另一种存在DTT(数据集2),并利用低温电镜(cryo-EM)技术解析出nsp12与它的辅因子nsp7和nsp8形成复合物时的三维结构。相关研究结果于2020年4月10日在线发表在Science期刊上,论文标题为“Structure of the RNA-dependent RNA polymerase from COVID-19 virus”。
细菌表达的全长SARS-CoV-2 nsp12(残基S1-Q932)与nsp7(残基S1-Q83)和nsp8(残基A1-Q198)孵育在一起,然后纯化出所形成的复合物。在这种复合物的存在下制备出低温电镜网格,初步筛选后发现这种网格具有良好的分散性和极佳的颗粒密度。在收集和处理7994个显微影片后,这些研究人员在2.9埃的分辨率下实现对nsp12单体分别与nsp12-nsp8二聚体和 nsp8单体形成的复合物的三维重建,正如之前在SARS-CoV中观察到的那样。除了nsp12-nsp7-nsp8复合物,他们还观察到了对应于nsp12-nsp8二聚体以及单个nsp12单体的单颗粒类型,但这些都没有给出原子分辨率下的三维重建。然而,nsp12-nsp7-nsp8复合物的三维重建提供了完整的结构分析信息。

图1.SARS-CoV-2病毒nsp12-nsp7-nsp8复合物的三维结构。图片来自Science, 2020, doi:10.1126/science.abb7498。
SARS-CoV-2 nsp12的结构包含一个“右手”RdRp结构域(残基S367-F920),以及一个套病毒(nidovirus)独特性的N端延伸结构域(残基D60-R249),其中这种延伸结构域采用了套病毒RdRp相关核苷酸转移酶(nidovirus RdRp-associated nucleotidyltransferase, NiRAN)结构。这种聚合酶结构域(即RdRp结构域)和NiRAN结构域由一个界面结构域(interface domain, 残基A250-R365)连接(图1A和图1B)。在清晰的低温电镜图的指导下构建出的一个额外的N端β发夹(D29-K50),插入到由RdRp结构域中的掌状亚结构域和NiRAN结构域夹持的凹槽中(图2)。这种nsp7-nsp8二聚体显示出类似于SARS-CoV nsp7-nsp8二聚体的保守结构。相比于nsp7-nsp8二聚体,与nsp12结合的nsp8单体的N端螺旋的定位发生了偏移。在nsp8的N端解析出的13个额外的氨基酸残基表明它的著名的 “高尔夫球杆(golf club)”形状的长“轴”是弯曲的。

图2.N端NiRAN结构域和β发夹的结构。图片来自Science, 2020, doi:10.1126/science.abb7498。
SARS-CoV-2 nsp12-nsp7-nsp8复合物的整体结构与SARS-CoV中的相似,1078个Cɑ原子的均方根偏差(root mean square deviation, rmsd)为0.82。然而,有区分这两者的关键特征。通过低温电镜图,这些研究人员得以构建出SARS-CoV-2 nsp12(包括除S1-D3和G897-D901以外的所有氨基酸残基)的完整结构。相比之下,SARS-CoV nsp12中的前116个氨基酸残基没有被解析出来。在SARS-CoV-2中解析出的NiRAN结构域的部分(残基4至28和51至249)由8个螺旋组成,在N端有一个五股β折叠(five-stranded β-sheet)(图2A)。在SARS-CoV-12病毒结构中,他们另外解析了残基A4-R118。这些构成了具有5个反平行β链和两个螺旋的结构体。残基N215-D218在SARS-CoV-2病毒nsp12中形成了一个β链,而这些残基在SARS-CoV nsp12中不那么有序排列。这个区域与包括残基V96-A100的链接触,从而有助于稳定其构象。因此,这四条链形成了一个紧凑的半β桶结构。因此,他们将残基A4-T28和Y69-R249确定为完整的冠状病毒NiRAN结构域。通过对N末端残基的解析,他们还能够确定一个N末端的β发夹(D29至K50,图1A和2A)。这个β发夹插入到由RdRp结构域中的掌状亚结构域和NiRAN结构域夹持的凹槽中,并形成一组紧密接触,以稳定整体结构(图2B)。另一点需要注意的是,他们观察到C301-C306和C487-C645在没有DTT的情况下形成二硫键。然而,在DTT存在的情况下,螯合锌离子存在,并且与在SARS-CoV中观察到的位置相同。
这种聚合酶结构域采用病毒聚合酶家族的保守结构,由三个亚结构域组成:手指亚结构域(残基L366-A581和K621-G679)、掌状亚结构域(残基T582-P620和T680-Q815)和拇指亚结构域(残基H816-E920)(图1)。这些催化性的金属离子在合成RNA的病毒聚合酶的几个结构中观察到,不过在引物-模板RNA和核苷酸三磷酸(NTP)不存在的情形下是观察不到的。
SARS-CoV-2病毒RdRp结构域的活性位点由掌状亚结构域中保守的聚合酶基序A-G形成,并且它的构型类似于其他的RNA聚合酶(图1A和3A)。由氨基酸残基611-TPHLMGWDYPKCDRAM-626组成的基序A包含经典的二价阳离子结合残基D618,这在大多数病毒聚合酶中是保守的,包括HCV ns5b(残基D220)和脊髓灰质炎病毒3Dpol(残基D233)(图3,B和C)和基序C(残基753-FSMMILSDDAVVCFN-767)包含在两个β链之间的转弯处的催化残基(759-SDD-761)。这些催化残基在大多数病毒RdRp---比如HCV ns5b中的317-GD-319和脊髓灰质炎病毒3Dpol中的327-GD-329---中也是保守的:第一个残基是丝氨酸或甘氨酸。
在这种结构中,与其他RNA聚合酶一样,模板/引物进入通道、NTP进入通道和新生链出口通道都带正电荷,是溶剂可接触到的,并汇集在一个中央空腔中,在这个中央空腔中,这些RdRp基序介导模板引导的RNA合成(图3D)。模板/引物进入通道、NTP进入通道和新生链出口通道的配置与SARS-CoV中的相类似,也与HCV聚合酶和脊髓灰质炎病毒聚合酶等其他RNA聚合酶相类似(图3,B和C)。NTP进入通道由一组亲水性残基形成,包括基序F中的K545、R553和R555。据预计RNA模板通过由基序F和G夹持的凹槽进入由基序A和C组成的活性位点。基序E和掌状亚结构域支持着引物链。产物-模板杂交体通过RNA聚合酶前侧的RNA出口通道退出这个活性位点。

图3.RdRp核心区域。图片来自Science, 2020, doi:10.1126/science.abb7498。
作为2-乙基丁基L-丙氨酸磷酰胺(2-ethylbutyl L-alaninate phosphoramidate)前体药物的Sp异构体,瑞德西韦(remdesivir, 也称为GS-5734)已被报道能够抑制SARS-CoV-2病毒增殖,因此具有临床潜力。这些研究人员将根据这项研究的结果简要地讨论它的潜在结合和抑制机制。链终止核苷酸类似物的疗效需要病毒RdRp来识别并成功地将它们的活性形式整合到生长中的RNA链上。索非布韦(sofosbuvir),即2'-F-2'-C-甲基尿苷单磷酸酯(2'-F-2'-C-methyluridine monophosphate),是一种靶向HCV ns5b的前体药物,已被批准用于治疗慢性HCV感染。它通过结合HCV ns5b聚合酶的催化位点而发挥作用。
鉴于瑞德西韦和索非布韦都是核苷酸类似物,以及SARS-CoV-2 nsp12和HCV ns5b聚合酶的催化位点在结构上的保守性,他们基于与结合到HCV ns5b上的索非布韦的叠加,对二磷酸瑞德西韦(remdesivir diphosphate)与SARS-CoV-2 nsp12之间的结合进行了建模(图4A)。总的来说,他们发现SARS-CoV-2 nsp12与ns5b的空载状态(Apo state)的相似度最高。鉴于ns5b在空载/延伸/抑制状态下的构象变化,这似乎表明催化残基D760、D761和典型的D618将发生构象变化,以便与这些二价阳离子形成配位键(图4B)。后者将与基序F中的变构残基R555一起锚定进入的核苷酸或抑制剂的磷酸基团(图4C)。在HCV ns5b延伸复合物或它与二磷酸索非布韦(pp-sofosbuvir)形成的复合物的结构中,一个关键的特征是整合到RNA链上的二磷酸索非布韦与N291(相当于SARS-CoV-2中的N691)相互作用,但由于它的糖基上发生氟取代,它不能与S282和D225连接成氢键网络(图4D),不过这是让进入的天然核苷酸保持稳定所必需的。然而,瑞德西韦保留了一个完整的核糖基团,所以它可能能够像天然底物一样利用这个氢键网络。此外,SARS-CoV-2 nsp12中的T680也可能与瑞德西韦的2'羟基形成氢键,当然也可能与进入的天然NTP形成氢键(图4D)。此外,基序F中的V557的疏水侧链很可能让+1模板RNA碱基尿苷保持稳定,使得尿苷与进入的三磷酸瑞德西韦(ppp-remdesivir)进行碱基配对(图4E)。

图4.瑞德西韦在SARS-CoV-2病毒nsp12中的掺入模型。图片来自Science, 2020, doi:10.1126/science.abb7498。
SARS-CoV-2在全球范围内的迅速传播突显了开发新的冠状病毒疫苗和药物的必要性。SARS-CoV-2病毒聚合酶nsp12看起来是开发新药物的一个很好的靶点,特别是考虑到瑞德西韦等先导抑制剂已经以化合物的形式存在。考虑到核苷类似物的结构相似性,这项研究中讨论的结合模式和抑制机制也可能适用于其他的此类药物或候选药物,包括在临床试验中证明有效的法匹拉韦(Favipiravir)。除了其他的有前景的药物靶标(比如SARS-CoV-2的主蛋白酶)之外,这个靶点还可以潜在地支持开发抗冠状病毒混合物,从而潜在地可用于发现广谱抗病毒药物。(生物谷 Bioon.com)
参考资料:
Yan Gao et al. Structure of the RNA-dependent RNA polymerase from COVID-19 virus. Science, Published online: 10 Apr 2020, doi:10.1126/science.abb7498.
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
87%用户都在用生物谷APP 随时阅读、评论、分享交流 请扫描二维码下载->


