研究解析参与植物胁迫应答的蛋白激酶-底物网络
来源:分子植物卓越中心 2020-02-12 09:24
1月28日,《美国国家科学院院刊》(PNAS)杂志在线发表了中国科学院分子植物科学卓越创新中心上海植物逆境生物学研究中心朱健康研究组、王鹏程研究组、美国普渡大学教授W. Andy Tao研究组合作的题为Mapping proteome-wide targets of protein kinases in plant stress re
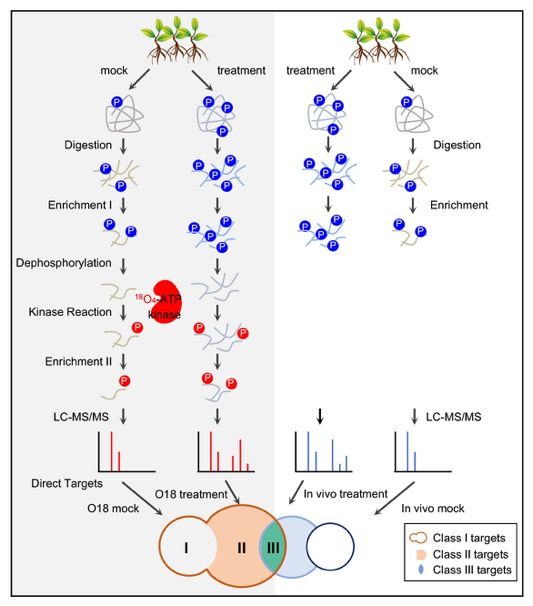
1月28日,《美国国家科学院院刊》(PNAS)杂志在线发表了中国科学院分子植物科学卓越创新中心上海植物逆境生物学研究中心朱健康研究组、王鹏程研究组、美国普渡大学教授W. Andy Tao研究组合作的题为Mapping proteome-wide targets of protein kinases in plant stress responses 的研究论文。该论文报道了一种新的蛋白激酶底物的鉴定方法,并对参与逆境胁迫应答的9个重要蛋白激酶的底物进行了系统分析。
蛋白磷酸化是重要的蛋白翻译后修饰之一。已知一些重要的蛋白激酶,如MPK6,SOS2,SnRK2等参与了植物对于不良环境的适应以及抵御病原体侵染的过程。干旱、冷、活性氧、病原体侵染等生物和非生物胁迫激活这些蛋白激酶,通过蛋白磷酸化调控底物蛋白的生物学功能,影响基因表达、代谢、物质转运等许多生理过程。目前,对这些蛋白激酶的底物及调控的下游生物学过程仍缺乏了解。
该研究以胁迫处理后的植物总蛋白为材料,富集磷酸化肽段,并通过磷酸酶去除肽段上的磷酸。以去磷酸化的肽段为底物池,利用原核表达的蛋白激酶和稳定同位素标记的ATP进行体外激酶反应。对反应产物进行二次磷酸化肽段富集和质谱检测,从而鉴定蛋白激酶的直接底物。同时,通过与体内胁迫处理后的磷酸化组学结果比较,进一步增加结果的可靠度(图1)。
通过这一方法,该研究解析了在氧化胁迫、冷、高盐和激发子处理下MPK6、渗透胁迫处理下SnRK2.4、ABA处理下SnRK2.6(OST1)和CKL2、盐处理下SOS2等9个重要植物蛋白激酶的约5000个可能底物。初步揭示了这些蛋白激酶直接的靶蛋白,为深入研究这些蛋白激酶的功能及其调控的下游生理过程提供了系统信息。
植物逆境中心的生物信息学平台基于这一研究的大规模数据,创建了蛋白激酶底物数据库(protein kinase–substrate database,KSDB, http://ksdb.psc.ac.cn)。KSDB数据库提供相关蛋白的磷酸化位点、可能的上游激酶、胁迫处理诱导的磷酸化变化等信息。
这一研究为在蛋白组学水平上鉴定蛋白激酶底物提供了一个高灵敏度、快速可行的方法。可以以少量植物为材料,在较短时间内对特定蛋白激酶的底物进行解析。同时这一研究提供的大量蛋白激酶-底物信息也为深入研究植物应答环境胁迫的分子机制提供了重要参考资源。(生物谷Bioon.com)
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


