Nature:给生命“天书”装个“Ctrl+F”——科学家把67万亿碱基压缩进硬盘,一键搜索全球基因库
来源:生物谷原创 2025-10-10 14:23
这项研究不仅仅是一项技术成就,其更像是一座连接海量生物数据与真实世界应用的桥梁。它告诉我们,那个曾经看似遥不可及的“谷歌 for DNA”的梦想,如今已在技术上成为可能。
想象一下,你面前摆着一套由67万亿个字母写成的“生命天书”,这就是全球公共基因数据库中储存的原始DNA数据总量,这个数字相当于把人类基因组重复打印2000多万次,其庞大程度几乎超出了人类直观的理解范围。
在新冠疫情中,我们见证了基因测序如何快速锁定病毒变种,为全球防控提供关键信息;然而,这只是冰山一角,从追踪抗生素耐药菌的全球传播到解密癌症的基因根源,再到绘制人体微生物地图,现代生命科学正日益依赖于对这些海量基因数据的挖掘。但一个巨大的矛盾随之而来:数据正以每45个月翻一番的速度疯狂增长,而我们分析其的能力却像试图用吸管喝干大海一样,传统的检索方式如同在图书馆里逐页翻找特定句子,效率低下,成本高昂,让绝大多数宝贵数据“沉睡”在硬盘的海洋里。
怎么办?是时候为生命的天书打造一个强大的“搜索引擎”了。

MetaGraph:把“基因宇宙”装进口袋的魔法
近日,一篇发表在国际杂志Nature上题为“Efficient and accurate search in petabase-scale sequence repositories”的研究报告中,来自苏黎世联邦理工学院等机构的科学家们就开发出了一种名为MetaGraph的方法,这项研究的核心目标非常明确,即要让任何人都能像使用谷歌搜索一样,快速、准确、低成本地在全球基因数据库中搜寻特定的DNA、RNA或蛋白质序列;这意味着一线医生或许能瞬间比对肿瘤患者的基因突变,环保科学家能快速监测水体中的抗生素耐药基因,研究人员能发现物种间未知的基因交流,这不仅是技术上的突破,更是推动精准医疗、公共卫生和基础科研民主化的关键一步。
神奇的“压缩与索引”魔法
开发MetaGraph方法的核心思想可以理解为两步:1)建造“乐高式”的基因图谱:其不像传统方法那样存储完整的基因序列,而是将所有的DNA/RNA数据“打碎”成更短的片段(称为k-mer),再像用乐高积木拼装一样构建成一个巨大的、包含所有片段关系的图谱(即“de Bruijn图”),这个图谱只存储每个独特“积木”一块,并记录它们的拼接方式,天然去除了大量重复数据;2)贴上“智能标签”:MetaGraph还为图谱中的每一个“积木”贴上标签,注明其来自哪个样本、哪个物种、甚至其表达量;然后利用高效的压缩算法将这些标签信息压缩到极致。
实验材料与流程:给全球基因数据“瘦身”
文章中,研究人员整合了来自七大公共数据库的数据,包括:1)对象:高达67 Petabases(相当于6700万亿个碱基) 的原始基因序列,涵盖了病毒、细菌、真菌、植物、动物和人类,包含超过1880万个独特的DNA和RNA序列集;2)流程:简单来说,就是对每个样本的数据分别构建小图谱并进行“清洗”(去除测序错误带来的噪音),然后将所有小图谱合并成一个全局的、标注好的超级图谱,最后进行极致压缩;3)技术:基于前沿的注释德布鲁因图和高效稀疏矩阵压缩技术。
从“不可能”到“触手可及”
这项研究得出的结论堪称颠覆:1)极致的压缩:整个公共基因数据库的索引最终可以压缩到约223 TB,这意味着,只需要几块消费级硬盘(总成本约2500美元)就能装下整个“基因宇宙”的搜索目录;2)低廉的搜索成本:搜索变得极其便宜;对于大型查询能精确匹配序列的成本可低至每百万碱基0.74美元,即使是针对整个数据库的小型查询(1兆碱基左右),成本也仅需约100美元;3)强大的应用案例:研究人员演示了如何在1小时左右,在一个计算节点上,从24万个肠道微生物样本中快速找出抗生素耐药基因与特定噬菌体的关联,这在传统方法下需要处理数百TB数据,几乎不可能实现。
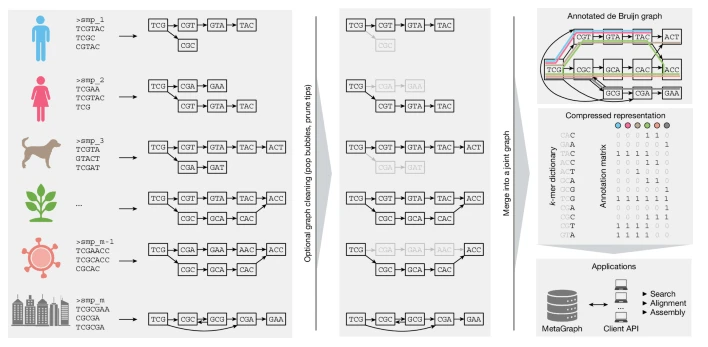
MetaGraph的框架
开启生物“大数据”搜索时代
这项研究不仅仅是一项技术成就,其更像是一座连接海量生物数据与真实世界应用的桥梁。它告诉我们,那个曾经看似遥不可及的“谷歌 for DNA”的梦想,如今已在技术上成为可能。其能将曾经专属于拥有超级计算机的大型机构的分析能力“下放”到普通实验室甚至个人研究者的手中,这极大地降低了生命科学研究的门槛,有望催生更多意想不到的发现。
未来,我们可以预见:1)疾病监测实时化:新病原体一经发现,即可在全球数据库中进行地毯式搜索,追溯其来源和传播路径;2)药物研发加速:快速筛选与疾病相关的基因靶点,并评估其在全球人群中的分布;3)生物多样性研究深化:轻松探索不同环境中所有生物的基因宝藏。
MetaGraph如同为人类点亮了一盏在基因数据黑暗宇宙中航行的明灯,它让我们相信,在生命这本浩瀚的天书中寻找任何一个关键“句子”都将不再是大海捞针,而是轻松的“一键搜索”。一个真正由数据驱动的生命科学新时代正由此开启。(生物谷Bioon.com)
参考文献:
Karasikov, M., Mustafa, H., Danciu, D. et al. Efficient and accurate search in petabase-scale sequence repositories. Nature (2025). doi:10.1038/s41586-025-09603-w
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


