研究人员设计出基于图表示学习和蛋白质语言模型的深度生成算法
来源:中国科大 2024-12-29 10:56
该研究成果推进了深度生成模型用于功能蛋白质设计,为进一步剖析蛋白质设计规律并开展生物实验验证奠定了基础
近日,中国科学技术大学认知智能全国重点实验室教授刘淇指导的博士研究生张载熙,联合美国哈佛大学医学院教授Marinka Zitnik课题组,设计了基于图表示学习和蛋白质语言模型的深度生成算法PocketGen,生成了与小分子结合的蛋白质口袋序列和空间结构。实验验证显示,PocketGen在生成成功率和效率方面均超过传统方法。相关研究成果以Efficient Generation of Protein Pockets with PocketGen为题,发表在《自然-机器智能》(Nature Machine Intelligence)上。
研发适用于科学发现任务的人工智能算法如功能蛋白质设计是重要的研究方向。在药物发现和生物医疗领域,设计与小分子结合的功能蛋白质具有积极意义。而基于能量优化和模板匹配的传统方法计算速度慢、成功率低。基于深度学习的模型存在分子-蛋白质复杂相互作用建模难、序列-结构依赖关系学习难等问题。因此,亟待发展高效、高成功率且能够准确反映物理化学规律的蛋白质口袋生成算法。
该团队在前期蛋白质口袋生成工作FAIR和PocketFlow的基础上,研发出PocketGen。PocketGen可以基于蛋白质框架和结合小分子生成蛋白质口袋序列和结构。PocketGen主要由双层图Transformer编码器和蛋白质预训练语言模型组成。受蛋白质固有的层级结构启发,双层图Transformer编码器包括氨基酸层级编码器和原子层级编码器,学习不同细粒度的相互作用信息,更新氨基酸/原子表示和坐标。在蛋白质预训练语言模型中,PocketGen高效微调ESM2模型,辅助氨基酸序列预测。具体方法为PocketGen固定大部分模型层不变,仅微调部分适应层参数,计算序列-结构信息交叉注意力,增强序列-结构一致性。实验显示,PocketGen模型亲和力和结构合理性等指标超过传统方法,在计算效率方面亦有大幅提高。
进一步,该研究在芬太尼和艾必克等小分子结合蛋白质口袋设计任务中进行验证,并与生成模型RFDiffusion、RFDiffusionAA等比较,验证了PocketGen的有效性。同时,研究将PocketGen产生的注意力矩阵与基于第一性原理和力场模拟分析软件得到的结果进行对比展示,发现基于深度学习的PocketGen具有较好可解释性。
上述成果推进了深度生成模型用于功能蛋白质设计,为进一步剖析蛋白质设计规律并开展生物实验验证奠定了基础,展现了人工智能方法在解决药物研发和生物工程领域重要科学问题方面的优势。
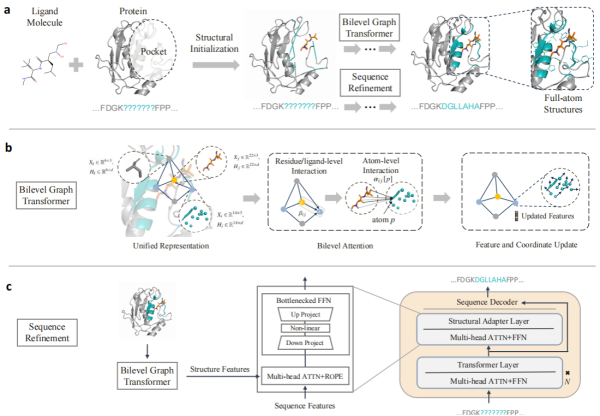
(a)利用PocketGen进行蛋白质序列-结构共同设计;(b)双层图Transformer编码器;(c)蛋白质预训练语言模型用于序列预测及高效微调技术
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


