清华大学刘俊杰为基因编辑做减法,无需蛋白质的基于RNA核酶的新一代基因编辑工具
来源:生物世界 2024-02-05 17:58
该研究阐明了HYER的DNA靶向切割机制,其与众所周知的RNA引导的CRISPR-Cas核酸酶相似,使用可重编程的单链RNA进行DNA识别,并通过水解来切割DNA。
清华大学生命学院刘俊杰(Jun-Jie Gogo Liu)课题组在国际顶尖学术期刊 Science 期刊发表了题为:Hydrolytic endonucleolytic ribozyme (HYER) is programmable for sequence-specific DNA cleavage 的研究论文。
该研究报道了一种新型基因编辑工具——HYER(水解型内切核酶),仅由具有催化活性的RNA(核酶)构成,HYER可序列特异性地切割RNA和DNA底物,并在哺乳动物细胞基因组中产生位点特异性的编辑。研究团队还通过理性设计,获得了工程化HYER,具有更高的特异性和灵活性。这也是科学界首次报道具有DNA水解切割能力的核酶。
HYER对底物的识别和切割仅依赖于RNA分子,而无需蛋白核酸酶参与,由于具有良好的可编程性,HYER有望成为继CRISPR之后的新一代基因编辑底盘工具。


刘俊杰研究员
刘俊杰,2011本科毕业于四川大学,2017年博士毕业于清华大学,此后曾在诺奖得主、CRISPR基因编辑先驱 Jennifer Doudna 教授实验室进行博士后研究,在此期间作为第一作者发表 Nature 论文,开发了一种基于CRISPR-Cas12e的小型化基因编辑工具——CasX,CasX转化的基因编辑疗法公司Scribe Therapeutics已完成超1亿美元融资,并与礼来、赛诺菲等制药巨头达成大额合作。
2020年10月,刘俊杰加入清华大学生命学院,成立了独立实验室。2023年1月,刘俊杰团队在 Cell Research 期刊发表论文,开发了基于CRISPR-Cas12l的小型化基因编辑工具——Casπ。2023年6月,刘俊杰团队在 Cell 期刊发表论文,解析了R2逆转座子在基因组上跳跃和切割DNA的分子机制,为开发基于逆转座子的新型基因编辑工具奠定了基础。
值得一提的是,在这篇 Cell 论文中,刘俊杰团队还将来自低等真核生物的R2逆转座子与其祖先(原核生物第二类内含子,GII intron)及哺乳动物中的LINE-1逆转座子进行了比较,发现第二类内含子向R2逆转座子及进一步向LINE-1逆转座子进化的过程中,RNA的结构性组分逐渐减少并被编码区域所取代,并且催化功能逐渐从以RNA为主导过渡到以蛋白质为主导。
1980年代,Thomas Cech 和 Sidney Altman 等人发现并证明了RNA也可以发挥催化作用,即所谓的核酶(Ribozymes),他们也因此获得了1989年的诺贝尔化学奖。
第二类内含子(GII intron)逆转座子,特别是它们编码的内含子RNA,是一类能够在宿主基因组通过“复制-粘贴”进行逆转座的可移动遗传元件,该元件通常编码一个兼具核酸内切酶(EN)和逆转录酶(RT)活性的蛋白质以及一个RNA分子,通过形成蛋白核酸复合物(RNP)来执行在宿主基因组中的逆转座扩增。
GII内含子可分为A、B、C三类,其中GII-C型内含子的大小仅为GII-A和GII-B的一半,被认为在进化上更古老。此前有研究发现,存在天然的缺乏开放阅读框(ORF)的GII内含子,它们被认为是GII内含子的“退化和无功能后代”,并且缺乏表征。迄今为止,广泛使用的GII内含子数据库仅列出了42个经生物信息学分析确定的缺乏ORF的GII-A和GII-B,尚未报道缺乏ORF的GII-C内含子。
刘俊杰团队对公开可用的细菌基因组序列进行初步筛选,发现许多缺乏ORF的GII-C型内含子,其中一些还在细菌基因组中存在多个拷贝,这表明,这些内含子编码的为唯一组分——RNA分子,可能在不依赖蛋白质的情况下仍然具有扩增位点识别和切割DNA的能力,从而通过“复制-粘贴”机制在基因组中进行拷贝扩增。
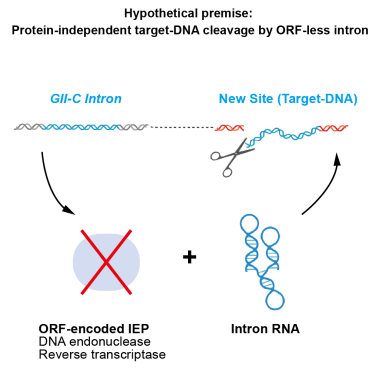
不含蛋白组分的GII内含子RNA在基因组中“复制-粘贴”的假设示意图
进一步研究显示,这些长度约600nt的RNA分子在广谱的离子浓度和温度范围内,具有显著的RNA和DNA水解切割活性。因此,研究团队将这些RNA分子命名为水解型内切核酶(Hydrolytic Endonucleolytic Ribozyme,HYER),这是科学界首次报道具有DNA水解切割能力的核酶(Ribozyme)。
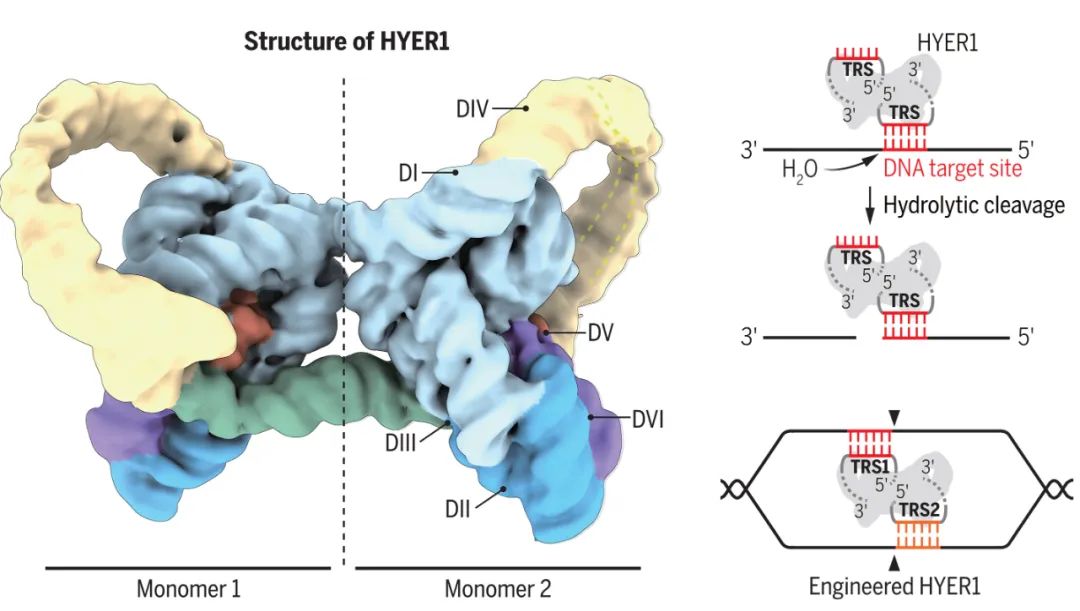
研究团队利用冷冻电镜获得了HYER1的高分辨三维结构(3.0Å分辨率),并解析了其水解切割DNA的机制,HYER1通过一段暴露的单链RNA(6nt)区域——目标识别位点(TRS),来识别并招募DNA底物,将DNA捕获在结构域V所形成的催化核心中,通过经典的双镁离子机制催化DNA水解,这表明了HYER是一种TRS引导的序列特异性DNA内切酶,其主要切割位点位于TRS-DNA互补区下游0或1 nt处。
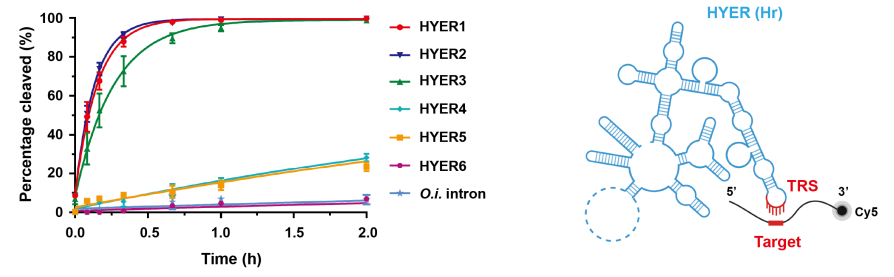
HYER的DNA切割能力
研究团队进一步验证了HYER在细胞内的DNA切割能力,在大肠杆菌和人HEK293T细胞内分别构建了ccdB毒蛋白报告系统和移码嘌呤霉素抗性基因(puro*)报告系统。结果显示,HYER可以在细胞内,尤其是真核细胞基因组中引入DNA双链断裂(DSB)并产生编辑效果。

HYER在大肠杆菌和人类细胞中切割DNA双链
天然HYER仅使用TRS识别6nt的DNA区域,这限制了其精准基因编辑的能力。研究团队基于HYER的三维结构,进行了多种理性设计。例如,可根据底物序列,灵活设计目标识别位点(TRS)的序列和长度;在TRS临近区域插入14nt的底物招募序列(Recruiting Sequence,RS),可明显提高HYER1的底物识别特异性和切割效率;对回文序列和TRS进行改造, HYER1则可形成带有两个不同TRS的异源二聚体,靶向双链DNA底物的不同区域,产生了具有5'突出、3'突出或平末端的定制化切割产物。这表明了HYER具有良好的可编程性,具有相当大的改造空间。
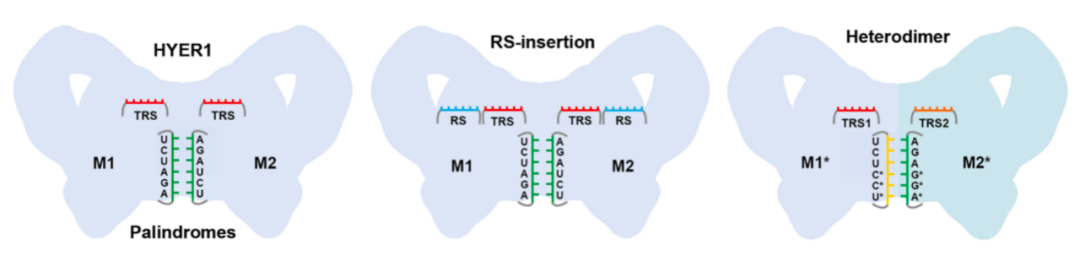
多种理性设计提高了HYER1的底物识别特异性和切割效率
最后,受“RNA世界”(RNA World)假说的启发,研究团队提出了第二类内含子(GII intron)中的“RNA的催化功能逐渐被蛋白质取代”的分子进化历程:在进化过程中,GII intron的结构域IV逐渐扩大,并产生了可编码短肽的开放阅读框(ORF),而这些短肽可作为顺式元件与内含子RNA相互作用,增强其结构稳定性和催化活性;随着ORF变得更长,更为成熟,其编码的蛋白质不仅起到稳定结构的作用,亦获得了DNA切割和逆转录活性,以替代RNA核酶行使催化功能。
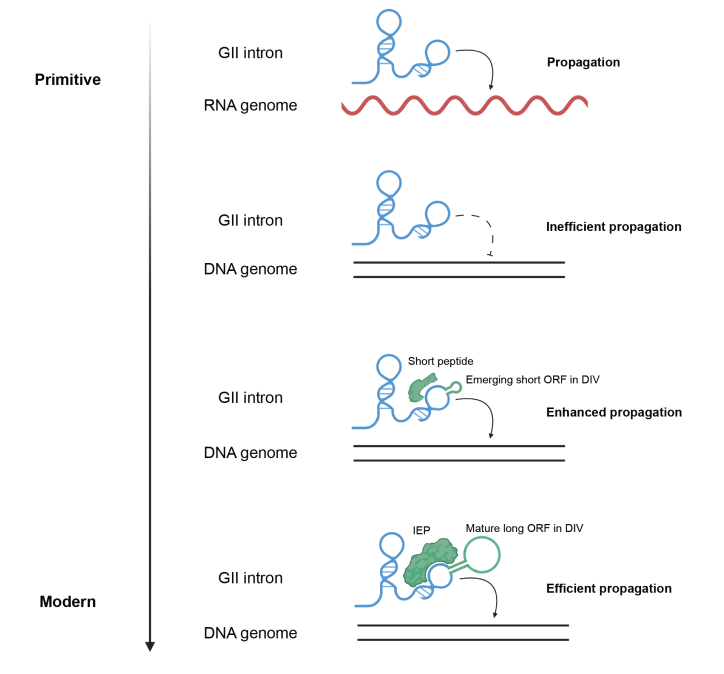
第二类内含子的进化假想
总的来说,该研究阐明了HYER的DNA靶向切割机制,其与众所周知的RNA引导的CRISPR-Cas核酸酶相似,使用可重编程的单链RNA进行DNA识别,并通过水解来切割DNA。相比广泛使用的CRISPR核酸酶的3-4kb的大小,HYER要紧凑得多,仅~0.6kb,因此更容易通过体外转录获取。
目前,天然HYER在体内编辑活性还很低,最有可能是因为其对双链DNA(dsDNA)的解旋能力有限。但正如我们在CRISPR-Cas9、CRISPR-Cas12f1等蛋白质核酸酶中所看到的那样,通过基因工程改造以提高体内基因编辑活性是可行的。
这项研究不仅拓展了科学界对RNA World假说和RNA催化功能的理解,也为创制具有中国完全自主知识产权的用于基因编辑和RNA编辑的新一代核酸操纵底盘工具奠定了基础。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


