Nature子刊:哺乳动物细胞高通量基因编辑平台构建及碱基编辑AI预测模型方面获进展
来源:天津工生所 2022-12-05 13:59
碱基编辑器(Base Editor,BEs)能够有效修改基因组的特定碱基,为纠正人类已知的致病性单核苷酸变异(Single nucleotide variations,SNVs)带来了希望。
碱基编辑器(Base Editor,BEs)能够有效修改基因组的特定碱基,为纠正人类已知的致病性单核苷酸变异(Single nucleotide variations,SNVs)带来了希望。基础科学探索和疾病治疗应用迫切需要构建大量具有致病性SNV的疾病细胞模型用于开展科学研究,而传统疾病细胞模型的构建主要依赖人工操作,不仅耗时,而且成本高昂、易出错。
中国科学院天津工业生物技术研究所研究员王猛带领的高通量编辑与筛选平台实验室、研究员毕昌昊带领的合成生物技术实验室、研究员张学礼带领的微生物代谢工程实验室合作,首次建立了自动化的哺乳动物细胞高通量基因操作平台,能够在一周内实现上千个动物细胞样本的自动化编辑,编辑效率与手动操作效率相当。基于高通量平台获得的293T细胞大量原位基因组编辑数据,研究人员开发了机器学习模型(CAELM)来预测胞嘧啶碱基编辑器(BE4max)的性能。与基于慢病毒整合靶点的传统机器学习建模相比,本研究创新性地在模型构建中考虑了目标序列的真实染色体环境,结合编辑靶点序列信息进行机器学习,首次提出染色质可及性对编辑结果的影响权重相对于靶点序列是1/6。为了扩展CAELM预测的兼容性,使CAELM模型适用于更多类型的细胞及不同的CBE碱基编辑器,科研人员在追加的相对较小的不同细胞类型(HepG2)及碱基编辑器(hyA3A-BE4max和Anc-BE4max)的编辑数据集上,令模型进行进一步训练学习,扩展了CAELM的预测范围。该模型可以比现有模型更准确的预测细胞原位靶点的碱基编辑结果,并为准确预测人类或其他物种细胞碱基编辑结果的机器学习模型的构建奠定了基础。这项工作有望加速基于BEs的基因疗法的开发和临床应用。
相关研究成果发表在《自然-通讯》(Nature Communications)上。研究工作得到国家自然科学基金、国家重点研发计划、天津市合成生物技术创新能力提升行动、天津市自然科学基金的支持。
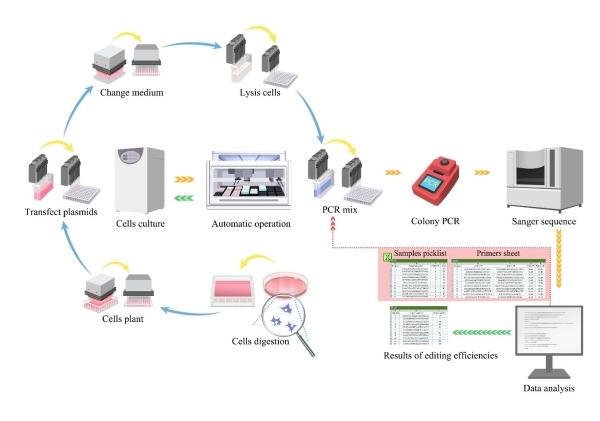
高通量自动化哺乳动物细胞编辑流程示意图
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


