低DNA用量、无扩增的PacBio建库技术开发成功
来源:网络 2024-07-25 10:24
LILAP作为三代低DNA输入量的建库方法,在不扩增的前提下将PacBio测序DNA投入量降低至100ng,适用于解析小型生物及其内共生体基因组。
基因组信息在生命科学研究中具有重要价值。基于三代长读长的测序组装越来越广泛地应用于基因组学研究。其中,PacBio高保真测序技术因高精度的测序质量被认为是三代测序的金标准。然而,PacBio标准建库方法需要大量DNA输入,限制了其在小型昆虫或珍稀样本中的应用。DNA扩增虽然可以降低样本输入量,但会引入扩增偏好和人工嵌合序列等错误,影响基因组的完整性以及变异检测的准确性。因此,亟待开发低DNA用量、无扩增且操作简单的PacBio建库方法。
7月5日,中国科学院动物研究所张勇研究组与中国农业科学院深圳农业基因组研究所阮珏研究组,在《自然-通讯》(Nature Communications)上在线发表了题为Low-input PacBio sequencing generates high-quality individual fly genomes and characterizes mutational processes的研究论文。该研究开发了基于Tn5转座酶的低DNA用量、无扩增、低成本的PacBio建库技术——LILAP。该团队利用这一技术实现了两个单只雄性黑腹果蝇的全基因组测序和高质量组装,获得了内共生菌Wolbachia完整基因组。基因组分析发现了两个突变特性即复杂转座事件更倾向于发生在non-B DNA序列富集的区域以及基因转换事件使转座子同质化。
LILAP利用Tn5转座酶和发卡结构的PacBio测序接头组成二聚体转座复合物,一步实现DNA片段化和测序接头连接。全部流程在单个试管内进行,最大限度减少了建库过程中的DNA损失,简化了建库流程。此外,测序接头内可添加条形码,可用于多样本混合建库测序。
进一步,该研究使用黑腹果蝇ISO1品系对该技术进行评估。结果显示,两个单只果蝇的读段测序质量中位数达到36即碱基错误率低于0.02%。从头组装的基因组N50数值超过6MB,质量分数超过60即碱基错误率低于10-6,保守单拷贝基因组装覆盖度大于98%,超过已发表工作中单只果蝇基因组的质量。
此外,科研人员在全基因组组装结果中发现了内共生微生物Wolbachia pipientis完整基因组。该团队检测了Wolbachia基因组内变异情况,发现了3个新突变,为后续研究共生菌和宿主的互作提供了新线索。
LILAP作为三代低DNA输入量的建库方法,在不扩增的前提下将PacBio测序DNA投入量降低至100ng,适用于解析小型生物及其内共生体基因组。未来,LILAP可应用于DNA用量受限的小型生物多样性研究及较大物种的体细胞变异分析、果蝇个体水平的全基因组关联分析以及宿主-共生体的协同进化研究。
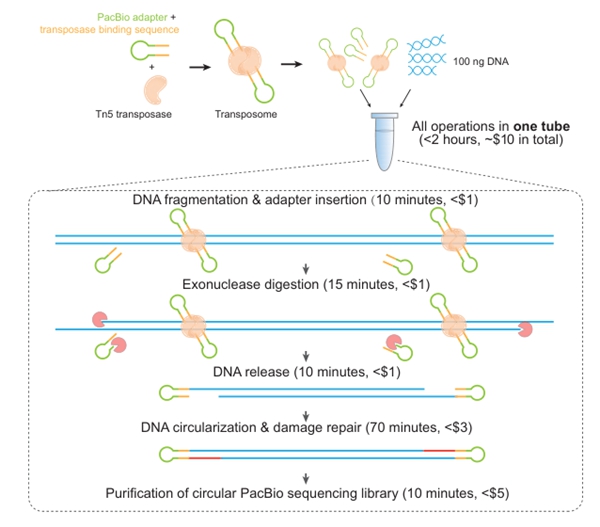
LILAP测序技术原理示意图
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


