Nature子刊:周英思/黄锦海/胥春龙/左二伟团队开发新型TadA碱基编辑器,实现DMD外显子55跳跃
来源:生物世界 2024-09-22 10:47
该研究通过简易的蛋白质改造策略,利用TadA同源物开发靶向范围更广的胞嘧啶碱基编辑器,并首次在DMD小鼠模型中实现了外显子55的跳跃。
碱基编辑(Base editing,BE)可以实现单个碱基的精准编辑,被认为是更安全的基因编辑方式,在基础研究、药物开发、基因治疗、动植物育种等方面具有巨大的潜力【1】。Broad研究所刘如谦团队将Cas9缺口酶(nickase)分别与APOBEC家族胞嘧啶脱氨酶或通过分子进化得到的大肠杆菌腺嘌呤脱氨酶TadA融合,开发出可实现C到T碱基转换的胞嘧啶碱基编辑器(Cytosine base editor,CBE)以及A到G碱基转换的腺嘌呤碱基编辑器(Adenine base editor,ABE)【2-3】。
2019年,中国科学院脑智卓越创新中心杨辉团队和中国科学院遗传所高彩霞团队分别在动物和植物上发现基于APOBEC的胞嘧啶碱基编辑器(CBE)存在基因组范围内的广泛脱靶,而基于TadA的腺嘌呤碱基编辑器(ABE)在基因组水平表现出了良好的特异性【4-5】。随后韩国汉阳大学Sangsu Bae团队发现基于TadA的碱基编辑器可以有效的编辑含有TC基序的位点【6】。华东师范大学李大力团队以及Broad研究所刘如谦团队分别基于不同的蛋白工程化方法,对大肠杆菌TadA进行突变,获得了基于TadA的胞嘧啶碱基编辑器【7-8】。复旦大学程田林团队通过对TadA同源物筛选获得了一系列具有不同功能的碱基编辑器【9】。
尽管多种基于TadA的胞嘧啶碱基编辑器被陆续开发出来,但不同的碱基编辑器仍然存在基序限制,腺苷脱氨酶活性残留,酶活效率不够高等问题。因此,开发基于TadA的胞嘧啶碱基编辑器对于基因编辑应用仍然具有重要意义。
2024年9月16日,辉大(上海)生物科技有限公司周英思团队,复旦大学附属耳鼻喉科医院黄锦海团队,临港实验室胥春龙团队和中国农业科学院农业基因组研究所左二伟团队合作,在 Nature Communications 期刊发表了题为:Engineering TadA ortholog-derived cytosine base editor without motif preference and adenosine activity limitation 的研究论文。
该研究基于辉大基因HGPRECISE®平台开发的TadA脱氨酶,成功升级开发了无基序限制的碱基编辑工具——aTdCBE,极大地拓宽了碱基编辑工具的靶向范围。通过AAV载体对aTdCBE进行体内递送,对DMD外显子55剪接位点进行编辑,成功地实现了DMD外显子55跳跃,并恢复了小鼠肌肉组织中dystrophin蛋白的表达,为杜氏肌营养不良(DMD)的治疗带来了新技术。

目前蛋白定向进化方法主要包括噬菌体辅助的持续演化技术(PACE),基于蛋白质三维结构的理性设计和基于人工智能的突变效应预测等方法,这些方法均已被成功应用于大肠杆菌TadA的工程化改造。在这项新研究中,研究团队试图根据氨基酸保守性信息开发一种简易的蛋白改造策略。
高度保守氨基酸位点(AHC)对蛋白功能的正常行使扮演了至关重要的作用,从而在漫长的进化过程中承受了强大的选择压力,无法容忍自然变异。而与高度保守氨基酸距离相近的非保守氨基酸可容忍自然变异的,那么对这些位点引入突变就可能有更高的概率对高度保守氨基酸的功能行驶产生上下扰动,从而获得酶活更强的突变体。基于这些假设,研究团队设计了一套TadA蛋白的筛选和优化策略(图1)。
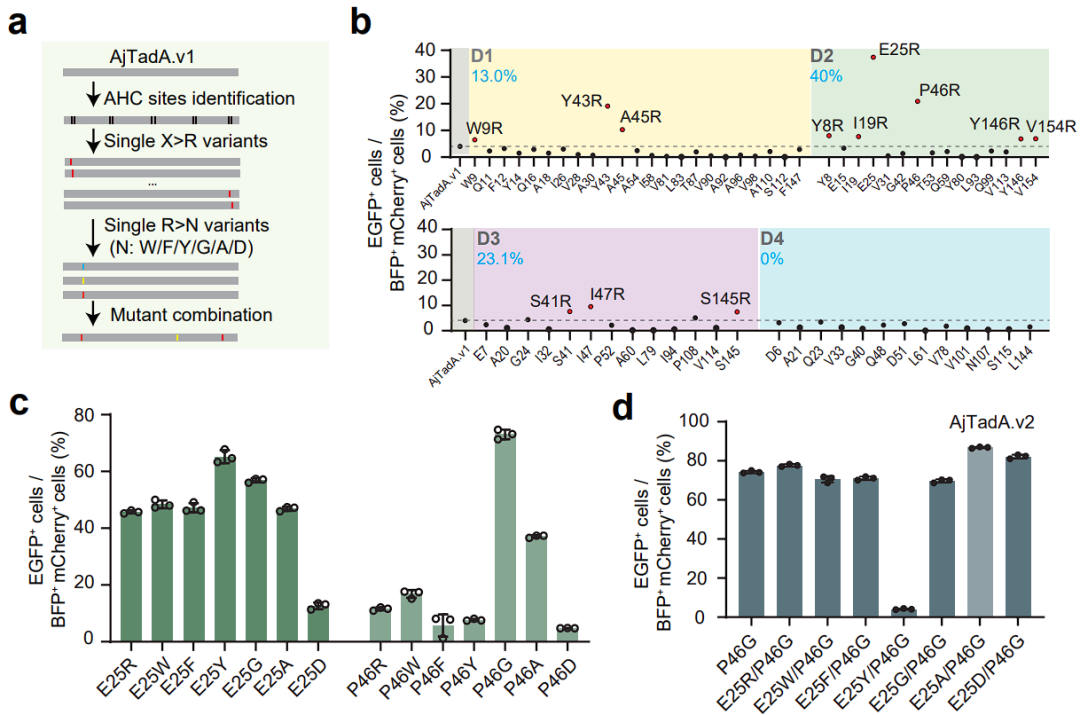
图1:TadA工程化策略、筛选及验证
首先,研究团队从NCBI数据库挑选的9个TadA同源蛋白,并根据最原始的ABE版本(ABE2.1)引入4个点突变。利用含有AC基序的C-to-G报告系统测试这些蛋白的活性,发现来源于Acinetobacter junii的TadA在报告系统上有高达4%的编辑活性。随后,研究团队对TadA家族的同源性进行了分析,鉴定了20个高度保守的位点,对高度保守氨基酸附近的非保守氨基酸进行了精氨酸突变。根据非保守氨基酸与高度保守氨基酸的距离不同,分成了D1、D2、D3和D4四组。与高度保守氨基酸间隔一位的非保守氨基酸被突变成精氨酸后(D2),有2/5的突变体的编辑活性是原来版本的1.5倍。
进一步,研究团队选择了其中最佳的两个突变体(E25R和P46R),并将其突变成其他五种不同类型氨基酸,发现P46G单突变体在荧光报告系统产生了高达63%的荧光。通过在P46G基础上进一步组合其他突变,获得了最终的aTdCBE碱基编辑器。通过与现有的胞嘧啶碱基编辑器进行比较,发现aTdCBE在20多个含有不同基序的内源位点具有较高的编辑效率,且不含有腺苷脱氨酶活性。
杜氏肌营养不良(DMD)是一种致死性X染色体隐性遗传神经肌肉疾病,其活产男婴发病率约为1:5000-1:3500。我国每年有400-500例DMD男婴出生,累计患者7-8万人,是世界上该病患者人数最多的国家之一。虽然有数千种记录在案的临床突变,但大多数引起DMD的突变发生在外显子45至55的“热点”区域,其中外显子55跃跳可以为大约2%的DMD患者提供治疗益处。设计具有广泛靶向范围和严格胞嘧啶脱氨酶活性的TadA可能是DMD基因编辑校正的潜在策略。
为了验证aTdCBE的应用潜力,研究团队首先用HEK293T细胞系测试,发现aTdCBE对DMD外显子55剪接受体位点(SAS)表现出高编辑活性。随后研究团队构建了DMD基因外显子54缺失的人源化DMD小鼠模型(DMD∆E54-mdx小鼠)。通过对3周龄DMD∆E54-mdx小鼠的胫骨前肌(TA)注射AAV9病毒,六周后收集TA肌肉样本进行后续分析。结果显示,aTdCBE在人源化DMD小鼠模型中实现了高达40%的DNA碱基编辑效率,同时dystrophin蛋白阳性纤维比达到99%,dystrophin蛋白表达水平达到60%。
这些结果表明,基于aTdCBE的胞嘧啶碱基编辑器作为一种具有广泛靶向范围的高效碱基编辑工具,为基础研究和治疗应用提供了一种有前景的方法。
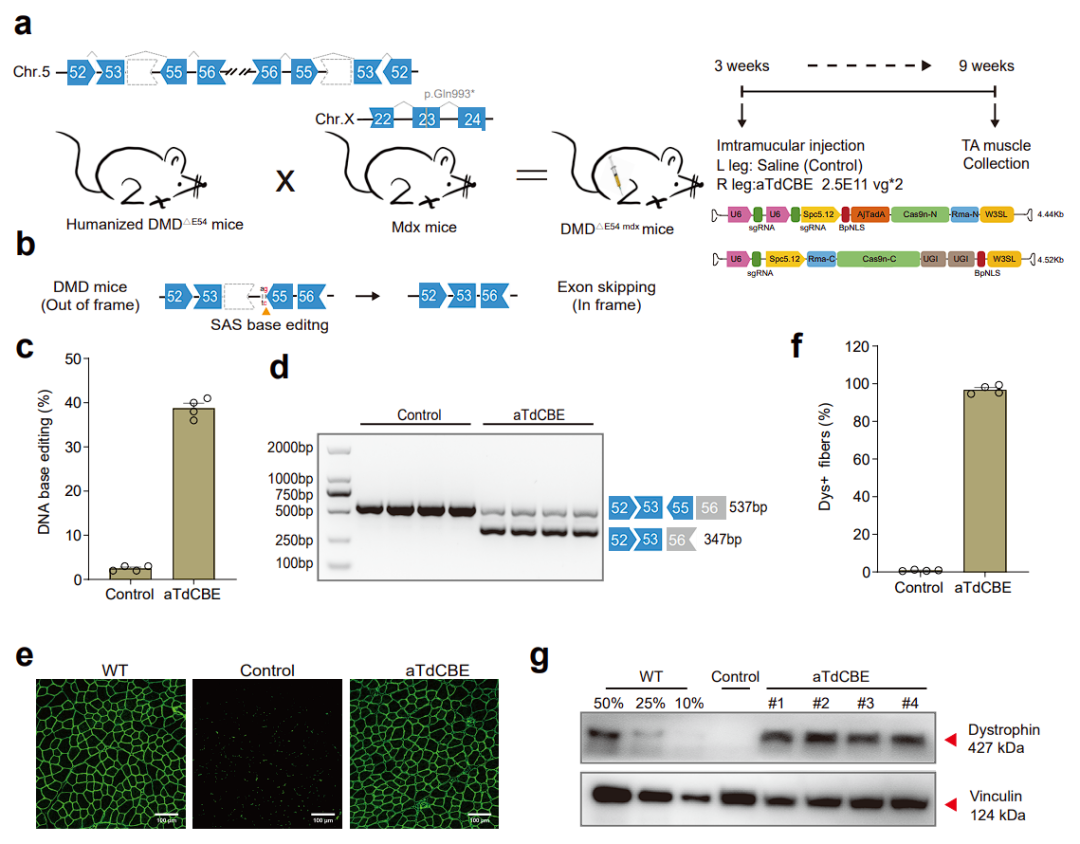
图2:通过外显子55跳跃治疗DMD疾病小鼠
总的来说,该研究通过简易的蛋白质改造策略,利用TadA同源物开发靶向范围更广的胞嘧啶碱基编辑器,并首次在DMD小鼠模型中实现了外显子55的跳跃。
辉大基因创新研究院李国玲博士、董雪博士、罗佳敏博士、李彤博士,深圳农业基因组研究所袁堂龙博士和复旦大学附属耳鼻喉科医院赵国丽博士为论文共同第一作者。辉大基因周英思博士(现中国科学院上海药物研究所研究员)、复旦大学附属耳鼻喉科医院黄锦海研究员、临港实验室胥春龙研究员和深圳基因组研究所左二伟研究员为论文共同通讯作者。
参考文献:
1.Porto, E.M., Komor, A.C., Slaymaker, I.M. & Yeo, G.W. Base editing: advances and therapeutic opportunities. Nat Rev Drug Discov 19, 839-859 (2020).
2.Komor, A.C., Kim, Y.B., Packer, M.S., Zuris, J.A. & Liu, D.R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature 533, 420-424 (2016).
3.Gaudelli, N.M. et al. Programmable base editing of A*T to G*C in genomic DNA without DNA cleavage. Nature 551, 464-471 (2017).
4.Zuo, E. et al. Cytosine base editor generates substantial off-target single-nucleotide variants in mouse embryos. Science 364(6437), 289–292. (2019).
5.Jin, S. et al. Cytosine, but not adenine, base editors induce genome-wide off-target mutations in rice. Science 364(6437), 292–295. (2019).
6.Kim, H. S. et al. Adenine base editors catalyze cytosine conversions in human cells. Nature biotechnology 37(10), 1145–1148. (2019).
7.Chen, L. et al. Re-engineering the adenine deaminase TadA-8e for efficient and specific CRISPR-based cytosine base editing. Nature biotechnology 41(5), 663–672. (2023).
8.Neugebauer, M. E. et al. Evolution of an adenine base editor into a small, efficient cytosine base editor with low off-target activity. Nature biotechnology 41(5), 673–685. (2023).
9.Zhang, S. et al. TadA orthologs enable both cytosine and adenine editing of base editors. Nature communications 14(1), 414. (2023).
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


