科研人员建立碱基编辑器消除脱靶的通用策略
来源:中山大学 2023-11-04 19:37
近日,中山大学生命科学学院李剑峰课题组在Nature Plants发表了题为“Split complementation of base editors to minimize off-target
CRISPR衍生的胞嘧啶/腺嘌呤碱基编辑器(CBE/ABE)由Cas9缺刻酶(nCas9)与胞苷或腺苷脱氨酶融合而成,能够高效实现基因组C>T或A>G的碱基突变,对动植物基因功能研究、作物分子育种和人类遗传病治疗具有重要意义,被喻为CRISPR皇冠上的明珠。和CRISPR技术一样,CBE/ABE也会因gRNA结合到基因组中与靶序列相似的序列而造成脱靶编辑(即gRNA依赖性脱靶)。近年来的研究进一步表明,CBE/ABE包含的胞苷或腺苷脱氨酶结构域会在动植物基因组和转录组中造成不可预测的DNA/RNA随机脱靶编辑(即gRNA非依赖性脱靶)。这种随机脱靶给CBE/ABE的广泛应用带来了负面影响。目前,解决gRNA非依赖性脱靶主要依赖点突变提高脱氨酶的高保真性或引入额外的脱氨酶活性调控元件。然而,提高脱氨酶特异性的点突变策略缺乏通用性,无法适用于不同序列/类型的脱氨酶,而且消除DNA和RNA脱靶的点突变叠加后可能会同时降低脱氨酶在靶位点的脱氨效率;引入脱氨酶调控元件则会增加系统的复杂性,也可能降低其通用性。
近日,中山大学生命科学学院李剑峰课题组在Nature Plants发表了题为“Split complementation of base editors to minimize off-target edits”的研究论文,报道了一种简单普适的CBE/ABE脱靶消除策略,即SAFE(split deaminase for safe editing)策略。该策略能够在水稻、拟南芥植株以及人、酵母细胞中实现高效、低脱靶的碱基编辑。Nature Plants同期以研究简报的形式对该研究进行了推荐。
作者首先提出关于SAFE策略的构想:从插入nCas9内部的脱氨酶结构域将碱基编辑器拆分为N端和C端两部分,使脱氨酶和nCas9同时失活,以防止脱氨酶的组成型活性造成gRNA非依赖性脱靶。在靶位点处,利用gRNA作为分子胶将拆分的两部分稳定重构为具备完全活性的碱基编辑器,从而实现中靶编辑。作者随后基于脱氨酶嵌入型CBE和ABE(即PIGS-AID10和PIGS-ABE8e),在胞苷脱氨酶AID10或腺苷脱氨酶TadA8e的无序区域内分别设计了7个候选拆分位点,利用植物原生质体瞬时表达系统和CBE/ABE的编辑活性报告系统对PIGS-AID10或PIGS-ABE8e的7种N端片段与7种C端片段之间形成的28种有效组合进行了重组编辑活性筛选。发现从AID10的Gly156-Arg157之间拆分PIGS-AID10或从TadA8e的Gly125-Met126之间拆分PIGS-ABE8e,可实现gRNA介导的中靶编辑。进一步,在水稻、拟南芥植株以及人HEK293T细胞、酵母中先后验证了前述策略介导的中靶编辑效率并全面评估了脱靶效应,发现该策略确实能够大幅抑制gRNA非依赖的基因组和转录组随机脱靶编辑。意外的是,该策略同时还能够降低gRNA依赖的DNA脱靶编辑,并且能够完全抑制CBE造成的缺失或插入(indel)突变。总之,SAFE策略不仅全面提升了动植物中碱基编辑的安全性和产物纯度,而且对不同脱氨酶变体组成的碱基编辑器通用,同时无需引入额外的脱氨酶活性调控元件。可预见的是,拆分后的碱基编辑器由于分子量变小,更利于病毒载体介导的碱基编辑器递送和在作物与人细胞中的表达。
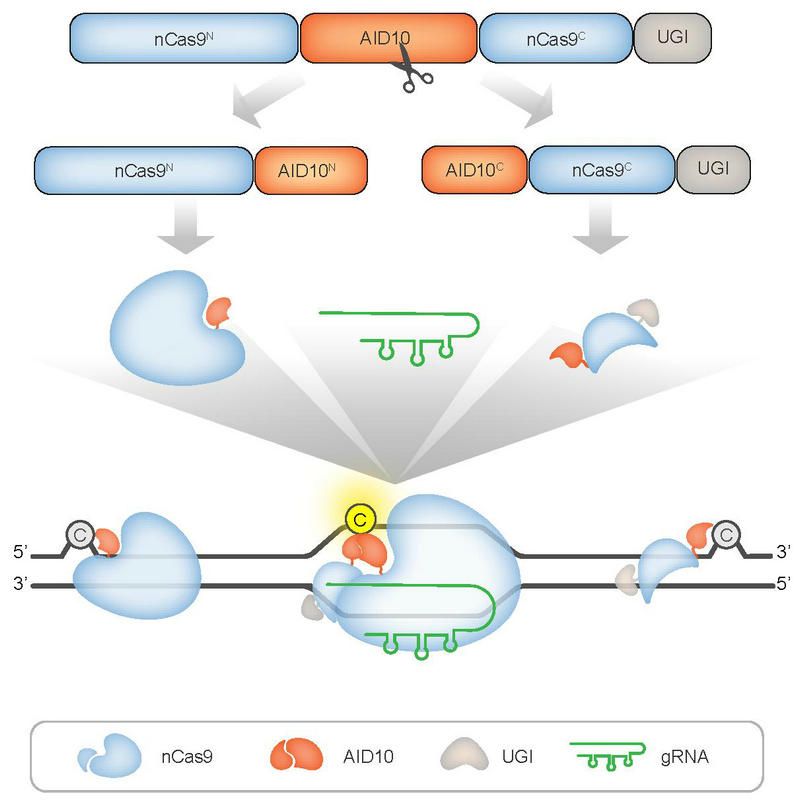
研究示意图
中山大学已毕业博士生熊翔宇和博士后刘科辉为该论文的共同第一作者,李剑峰教授和博士后刘科辉为共同通讯作者。中山大学贺雄雷教授、博士后黎镇祥、夏凡女和博士生阮雪铭参与了该研究。该研究得到国家重点研发计划合成生物学专项等经费资助。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


