Nat Biotechnol:新型计算方法CoNGA为预测T细胞行为奠定基础
来源:本站原创 2021-09-05 16:06
研究人员开发出一种称为CoNGA的新型计算方法,它可能有助于人们关注将T细胞受体(TCR)基因序列和T细胞功能相关联在一起的隐藏生物模式。此外,利用CoNGA,他们可以分析从单个T细胞中收集到的复杂数据,以揭示新的T细胞群体,并揭示塑造T细胞发育的TCR特征。
2021年9月5日讯/生物谷BIOON/---免疫细胞有许多工作要做:一些细胞识别受感染的细胞并将它们消除,其他细胞帮助控制炎症,以防止对健康组织的损害。许多免疫细胞是癌症治疗的关键组成部分。人们知道,一类称为T细胞的免疫细胞的特定受体有助于调节T细胞的活性和免疫作用。如今,在一项新的研究中,来自美国弗雷德-哈钦森癌症研究中心、圣犹大儿童研究医院、10X基因组学公司和南加州大学的研究人员开发出一种称为CoNGA(clonotype neighbor graph analysis, 克隆型邻接图分析)的新型计算方法,它可能有助于人们关注将T细胞受体(TCR)基因序列和T细胞功能相关联在一起的隐藏生物模式。此外,利用CoNGA,他们可以分析从单个T细胞中收集到的复杂数据,以揭示新的T细胞群体,并揭示塑造T细胞发育的TCR特征。相关研究结果于2021年8月23日在线发表在Nature Biotechnology期刊上,论文标题为“Integrating T cell receptor sequences and transcriptional profiles by clonotype neighbor graph analysis (CoNGA)”。

近期的技术进展使得科学家们能够窥视单个细胞内部并揭示哪些基因开启和关闭。论文共同通讯作者、弗雷德-哈钦森癌症研究中心的计算生物学家Phil Bradley博士说,然而,来自数百万个细胞的单细胞数据产生了巨大而复杂的数据集,超出了个人的解释能力。Bradley说,“CoNGA是一种算法,它的灵感来自于刚刚上线的这一类新的[单细胞]数据,为我们提供了一种可以分析它们的方法。”
Bradley和他的合作者设想其他研究人员使用CoNGA来更好地理解复杂的T细胞群体,包括那些对肿瘤作出反应的T细胞。CoNGA可能让致力于改善癌症免疫疗法---如CAR-T细胞免疫疗法或免疫检查点抑制剂---的科学家们更好地了解驱动患者对治疗成功作出反应的因素,这是设计这些免疫疗法的更好版本的第一步。
T细:多方面的免疫细胞
T细胞通常是所谓的适应性免疫的一部分----在你受到感染后,适应性免疫会发生变化,在长寿的免疫细胞中形成一种“记忆”,如果你再次遭受同样的感染,它们的反应会更加迅速和有效。这种能力的一部分在于位于T细胞表面的T细胞受体(TCR)。我们身体产生的每一个新的T细胞都有自己独特的TCR,它是从一系列选项中将TCR基因片段重排在一起而产生的。
Bradley说,“T细胞是非常有趣的免疫细胞--它们有这种受体,而这种受体的性质或它喜欢粘附的东西,在原则上决定了该细胞在感染或癌症时的命运。”
T细胞利用它们的TCR来调查我们体内的细胞,寻找被感染或患病的细胞。目标细胞在其表面显示供T细胞采样的分子“标记(tag)”。当T细胞的TCR与目标细胞表面上的分子标记结合时,就意味着目标细胞出现了问题。
不同的T细胞有许多不同的职责。“杀伤性”T细胞使用其TCR来确定应该被杀死的细胞。其他的T细胞为产生称为抗体的保护性蛋白质的免疫细胞提供帮助。还有一些T细胞抑制免疫反应以防止对健康组织的损害。在每个类别中都有许多不同的T细胞,帮助身体根据需要调整其免疫反应。
TCR和它“看到”的东西在决定T细胞是否会成为杀伤性T细胞、辅助性T细胞或免疫交通控制者方面起着作用。但这个过程的大部分工作原理仍然是神秘的。人们仍在寻求从TCR序列中预测TCR靶标的方法,此外还在了解TCR如何影响T细胞发育。能够将TCR序列与T细胞内部发生的事情相匹配---哪些基因开启或者说发生转录---使人们了解到该细胞正在发挥什么作用,以及它目前是在执行任务还是在等待被招募。
Bradley说,“我们真正感兴趣的是,如果你获得T细胞内的转录概况,这与表面的T细胞受体的序列有什么关系?最终的目标是,你可以利用T细胞受体序列并预测T细胞正在做什么。这是这个领域的最高目标,而这真的非常非常难。我们离这个目标还有一段距离。”
为了实现这个最高目标,科学家们需要来自大量单个T细胞的信息---他们越多地发现具有特定TCR特征的T细胞也开启了特定基因,这种联系就越有可能是真实的---但是分析这些复杂的数据集充满挑战性。近期的技术飞跃使得科学家们能够从数以百万个细胞中收集信息。这些数据集可以包括基因序列,哪些基因开启(以及在多大程度上开启),以及哪些蛋白质在细胞表面上表达。每一个新的数据层都使分析更具挑战性。
这不是一个人能自己分析的。我们需要数学。这就是为何Bradley、作为另一名论文共同通讯作者的圣犹大儿童研究医院免疫学家Paul Thomas博士、Thomas团队博士后研究员Stefan Schattgen博士和南加州大学的本科生Kate Guion开发出CoNGA。基本上,他们创建了能够比较两种图谱的算法。一种图谱对具有类似转录特征(它们开启的基因)的T细胞进行分组,另一种图谱根据TCR序列的相似性对T细胞进行分组。CoNGA算法定义了这两种图谱之间的关系,找到了TCR“邻域”(具有相似生化特征的TCR序列)和这些T细胞中开启的相同基因之间的关联。这些模式可能为TCR如何帮助决定T细胞行为提供线索。
新模式,新可能性
这些作者利用了主要由构建单细胞分析平台的生物技术公司10X Genomics产生的公开可用的T细胞数据集。这些单细胞数据包括有关TCR序列和每个T细胞中哪些基因开启的信息。这些数据的一个子集还包括关于TCR粘附在哪些分子靶标上的信息。
当将CoNGA应用于这些数据时,这些作者发现了TCR序列和T细胞中开启的关键基因之间的相关性。CoNGA分析还使他们能够在T细胞中找到一些与这些细胞的活性和信号传递有关的新的、意想不到的标签(signature)。尽管这些特征的生物学意义仍不清楚,但其中的一种标签可能指向一种以前未知的T细胞类型,而另一种标签可能提供了某些TCR特征如何塑造T细胞发育的线索。
Bradley及其队友们的最初目标是证明CoNGA能够在复杂的数据中找到有意义的模式。如今,它已经通过了第一次测试,他们正在努力跟进它所揭示的新的关联性。
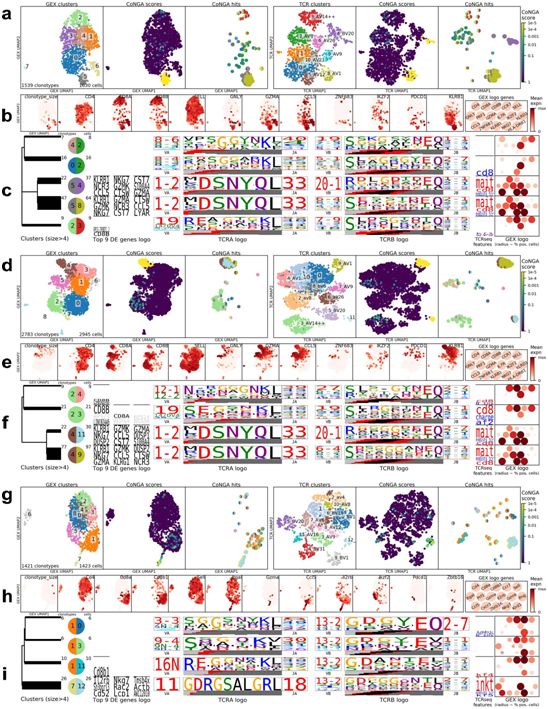
对人类和小鼠外周血T细胞进行CoNGA图谱分析,图片来自Nature Biotechnology, 2021, doi:10.1038/s41587-021-00989-2。
首先,Bradley团队希望确认CoNGA揭示的不寻常的T细胞群体对科学来说确实是新的。(布拉德利指出,当新工具推出时,重新发现以前描述的细胞的情况经常发生。)如果这些细胞真的是新的,他们将深入研究它们在免疫反应中发挥的作用。
Bradley和他的合作者们也在考虑更大的问题。他们希望将CoNGA应用于更大的数据集(更多的数据意味着对统计模式反映真实生物学的信心更强)。他们正在努力调整CoNGA,使之适用于B细胞,即一种制造抗体的免疫细胞。B细胞也携带称为B细胞受体(BCR)的特定受体,BCR可以调节它们的发育、行为和功能---而CoNGA可能帮助科学家们了解这是如何调节的。
Bradley团队还对CoNGA促进其他研究小组的分析的潜力感到兴奋。虽然该团队聚焦于TCR,但是CoNGA也可能应用于整合其他类型信息的单细胞数据集,如T细胞表面的特定分子。
该团队将向公众免费提供他们的CoNGA算法,供科学家们根据需要使用或修改。Bradley说,“我确实认为这将是一种对免疫学家有用的工具。”(生物谷 Bioon.com)
参考资料:
Stefan A. Schattgen et al. Integrating T cell receptor sequences and transcriptional profiles by clonotype neighbor graph analysis (CoNGA). Nature Biotechnology, 2021, doi:10.1038/s41587-021-00989-2.
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


