Cell Res:孙丽明团队发现功能性淀粉样蛋白纤维解聚酶抑制程序性细胞坏死
来源:生物世界 2023-08-18 10:44
综上所述,该项工作报道了哺乳动物中的第一个蛋白纤维解聚酶amyloidase—HSPA8。HSPA8通过逆转RHIM蛋白纤维化,成为细胞内抑制程序性细胞坏死发生的关键检验点。
中国科学院分子细胞科学卓越创新中心(生物化学与细胞生物学研究所)孙丽明研究组在 Cell Research 期刊发表了题为:HSPA8 acts as an amyloidase to suppress necroptosis by inhibiting and reversing functional amyloid formation 的研究论文。
该研究发现HSPA8特异性识别RHIM蛋白,抑制其形成蛋白纤维;此外,HSPA8还具备蛋白纤维解聚酶(amyloidase)活性,能够将已经形成的RHIM蛋白纤维解聚成非激活形式的单体形式。在细胞和小鼠体内HSPA8通过抑制RHIM蛋白聚合和解聚RHIM蛋白纤维而抑制程序性细胞坏死。

该研究中研究人员首先通过全基因组siRNA筛选,发现HSPA8 (也称为Hsc70)可以抑制细胞自发激活程序性细胞坏死。在细胞内敲低HSPA8促进了程序性细胞坏死的发生,敲除或抑制RIP3的激酶活性挽救了HSPA8敲低引起的细胞坏死增加;小分子化合物抑制HSPA8活性也促进了程序性细胞坏死信号的传递;说明在细胞中HSPA8抑制程序性细胞坏死。
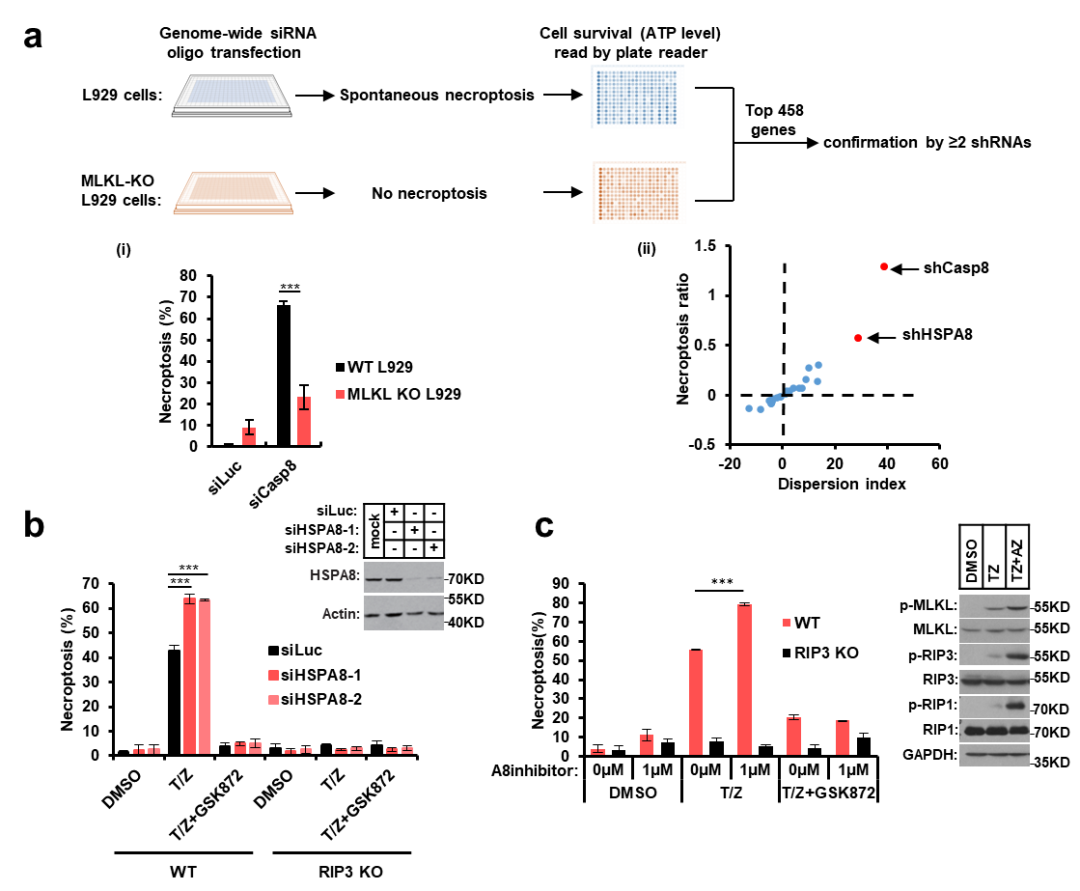
HSPA8抑制程序性细胞坏死
进一步研究发现在小鼠体内抑制HSPA8的活性,能够直接诱发程序性细胞坏死介导的体温失衡和肠道损伤,且能加重小鼠的全身性炎症反应综合征(SIRS)反应。敲除Rip3或Mlkl能够挽救HSPA8功能缺失所造成的组织损伤,验证了HSPA8在体内靶向程序性细胞坏死的特异性。
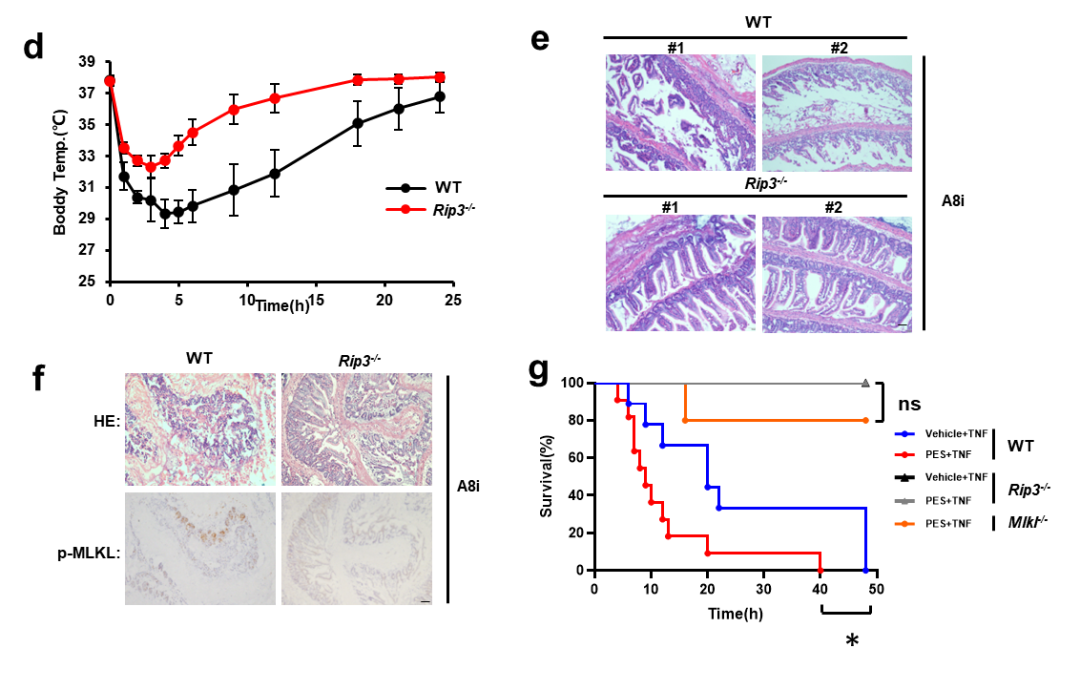
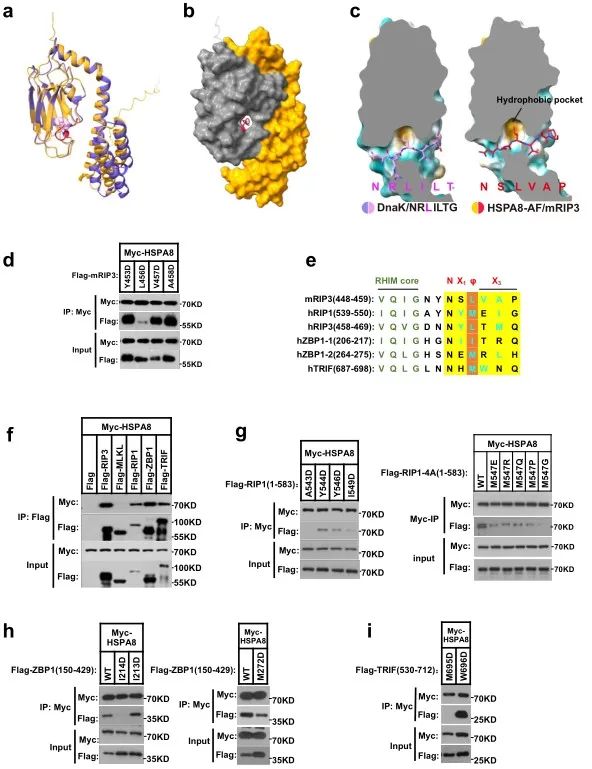
已被报道的HSPA8通过识别包含KFERQ基序的底物,引导底物进入溶酶体降解的途径参与分子伴侣介导的自噬(CMA)。但研究人员排除了HSPA8依赖的CMA的参与(Chaperone-Mediated Autophagy,分子伴侣介导的自噬),猜测HSPA8 通过独特的机制调控程序性细胞坏死。接着研究人员发现HSPA8的底物结合结构域SBD 可以与RIP3的RHIM结构域结合,抑制RIP3的寡聚化和纤维化。细胞内缺失HSPA8后,RIP3会自发地发生寡聚化和纤维化。
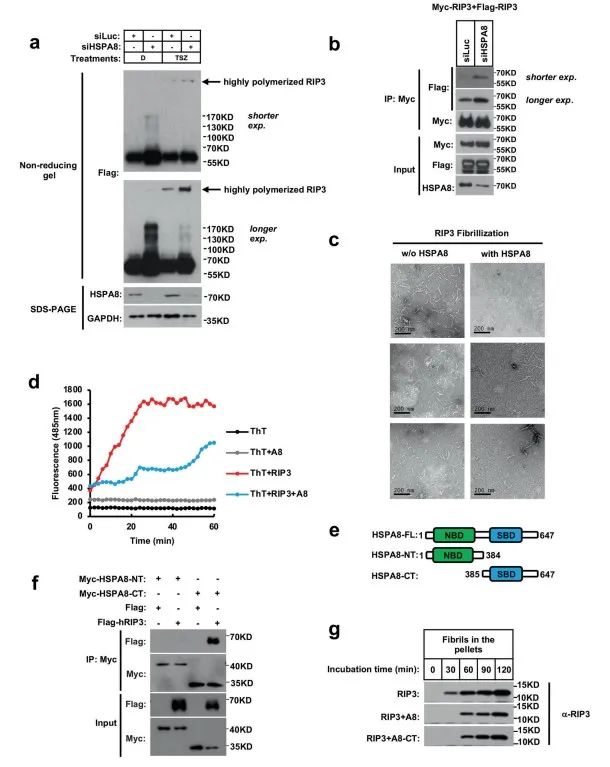
更进一步,研究人员发现HSPA8可以不依靠其他的co-chaperone,单独解聚已经纤维化的RIP3蛋白纤维,且这个过程依靠水解ATP 提供能量。
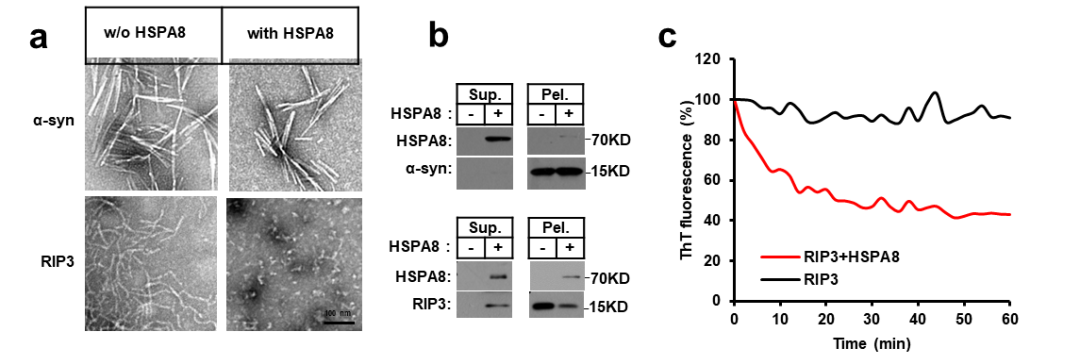
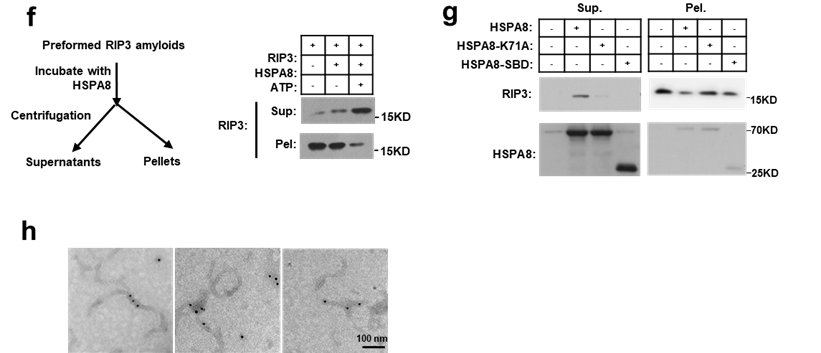
除了RIP3之外,研究人员还发现HSPA8具有广泛的能力来识别RIP1、ZBP1和TRIF。通过蛋白结构模拟和突变验证,研究人员总结出HSPA8通过识别RHIM结构域中的疏水性六肽基序—N(X1)φ(X3)与RHIM蛋白之间的相互作用。值得注意的是,六肽基序中的第三个氨基酸在介导RHIM蛋白与HSPA8之间的相互作用中起着至关重要的作用,其突变显著干扰了蛋白之间的互动。与对RIP3蛋白纤维的调控类似,HSPA8还能够抑制并逆转这些启动型RHIM蛋白(即RIP1、ZBP1和TRIF)的纤维化,从而抑制程序性细胞坏死通路的激活以及下游MLKL的磷酸化和寡聚化。
综上所述,该项工作报道了哺乳动物中的第一个蛋白纤维解聚酶amyloidase—HSPA8。HSPA8通过逆转RHIM蛋白纤维化,成为细胞内抑制程序性细胞坏死发生的关键检验点。HSPA8在体内不依赖于co-chaperone而独立解聚RHIM功能性蛋白纤维,从根本上支持了功能性蛋白纤维的可逆性特性,为区分疾病相关淀粉样蛋白纤维提供了直接依据。研究人员推测,细胞中多种功能性蛋白纤维可能对应多种潜在的蛋白纤维解聚酶,寻找新的纤维解聚酶对于人们深入了解细胞稳态维持和信号传递的监控十分重要。该项研究也拓展了对非蛋白降解型负调控信号转导的认识,为开发针对程序性细胞坏死相关疾病的治疗新靶点提供了新的视角。
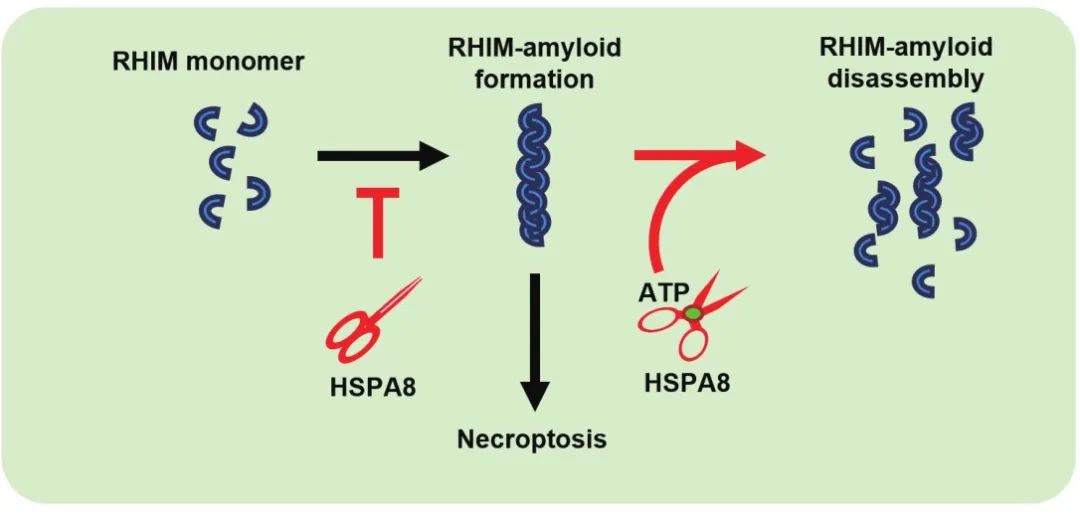
HSPA8抑制程序性细胞坏死的机制概要图
中国科学院分子细胞卓越中心孙丽明研究员为本文通讯作者,博士研究生武二鹏、何文艳为共同第一作者。该研究得到分子细胞卓越中心汪胜研究员、上海科技大学陆珺霞研究员和王华翌研究员、复旦大学李继喜研究员等单位专家的大力协助。该研究获中国科学院、国家自然科学基金委等经费支持,获分子细胞卓越中心动物实验技术平台、细胞分析技术平台、化学生物学技术平台技术支持。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


