研究发布新的非编码RNA整合资源NONCODEV6数据库
来源:生物物理所 2021-01-09 12:49
长非编码RNA(long non-coding RNAs)是一类长度大于200 nt的非编码RNA。已有研究表明,长非编码RNA具有重要的调控功能,在植物和动物的生物学过程中发挥重要作用。此外,lncRNA还与人类一些疾病的发生发展密切相关。因此,对lncRNA的注释、探索lncRNA的功能具有重要意义。近年来,一些研究集中于lncRNA功能的探
长非编码RNA(long non-coding RNAs)是一类长度大于200 nt的非编码RNA。已有研究表明,长非编码RNA具有重要的调控功能,在植物和动物的生物学过程中发挥重要作用。此外,lncRNA还与人类一些疾病的发生发展密切相关。因此,对lncRNA的注释、探索lncRNA的功能具有重要意义。近年来,一些研究集中于lncRNA功能的探索,但综合全面的lncRNA的注释仍需不断更新维护。
NONCODE数据库于2005年创建,受到Science专文推荐,由中国科学院计算技术研究所、生物物理研究所团队长期维护;2013年,受邀以专家数据库加入国际RNA联盟RNAcentral,数据库首次提出了非编码基因的分类体系,建立了多项非编码领域标准,推动了长非编码RNA的研究发展。
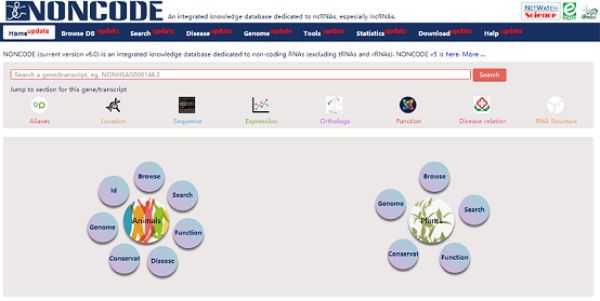
近期,中科院院士、生物物理所研究员陈润生课题组,生物物理所健康大数据研究中心研究员何顺民课题组和计算所研究员赵屹课题组合作,在Nucleic Acids Research上,在线发表题为NONCODEV6: an updated database dedicated to long non-coding RNA annotation in both animals and plants的研究论文(图1)。该工作NONCODEV6(http://www.noncode.org/)(图2)是关于动植物多个物种的长非编码RNA综合注释数据库的升级,旨在提供关于动植物各个物种中lncRNA的注释和分析。
在前期积累的NONCODE版本的基础上,研究人员通过关键词搜索近期发表文章并手工提取lncRNA及其在组织中的表达数据等信息;对其相应的植物物种、动物物种、组织表达数据进行了收集、整理;进入NONCODE数据库更新的流程,进行数据的过滤、处理、注释。NONCODEV6中总共记录了644,510个lncRNA。此次新加入的植物物种数目达23种,包括常见植物拟南芥、水稻、小麦、玉米等。植物物种的lncRNA注释包括基本位置信息、序列信息、长度、外显子个数、组织的表达量、功能注释等。同时,研究人员对人和小鼠的数据进行了更新,加入了lncRNA和肿瘤等疾病的关系注释,整合了多个lncRNA和肿瘤的数据源,提供了一个关于肿瘤等疾病和NONCODE数据库中lncRNA的综合注释。
此外,NONCODEV6数据库提供了一个用户友好的界面,可用于浏览各个物种的长非编码RNA的具体情况,包括长度、序列、位置、功能注释、保守性注释等;其还提供了blast功能、功能注释查询、保守性查询。总体来说,NONCODEV6是一个业内较认可的综合性lncRNA注释的知识库,为研究基于lncRNA的在动物和植物的相关注释提供了重要支撑。(生物谷Bioon.com)
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


