科研人员开发估算祖先细胞群体大小的算法TarCA用于揭示早期细胞命运决定
来源:中山大学 2024-03-18 09:06
该研究开发的TarCA算法可对任意细胞群体估计祖先细胞数目,而无需事先获得祖细胞的先验信息,也能推算少数细胞中的驱动基因,以此来预测早期细胞命运的分化倾向。
中山大学生命科学学院贺雄雷课题组在Nature Methods上发表了一篇题为A statistical method for quantifying progenitor cells reveals incipient cell fate commitments的研究文章。该研究基于近年来逐渐成熟的细胞谱系追踪技术构建的发育细胞谱系树 (developmental cell phylogeny),结合群体遗传学的经典溯祖理论 (coalescent theory) 思想,建立了一种估计祖先细胞群体大小的统计方法TarCA (targeting coalescent analysis),以此来研究胚胎发育过程中细胞群体的动态变化。
给定一颗包含多种细胞类型的细胞谱系树,它的末端节点代表了被采样的细胞,而中间节点则表示祖先细胞。如果一个中间节点的所有子代细胞均为相同的细胞类型,研究人员将其定义为细胞类型特异的单系分支 (monophyletic clade)。随后,研究人员根据单系分支的大小分布计算出随机两个细胞均来自同一单系分支的概率,而该类型细胞的祖先细胞群体数量即为该概率的倒数(图1)。

图1 估计祖先细胞数目的传统方法与TarCA
通过计算机模拟数据,该研究发现,与对单系分支进行直接计数的方法相比,TarCA对采样率要求更低,并且在实验噪音存在时仍能够快速收敛。这是因为在TarCA算法中,子代细胞数量较少的祖先细胞权重较低,从而TarCA能稳定地估计出对子代细胞群体做出主要贡献的祖先细胞数量。随后,研究人员在线虫、果蝇与小鼠的细胞谱系数据中,验证了TarCA在处理真实生物学数据中的可靠性。以小鼠为例,研究人员发现不同组织的祖先细胞群体数量在个体间高度一致。另外,除采样获得的终末细胞类型外,TarCA还能对发育初期的胚胎结构(如三胚层及胚外组织等)的祖先细胞群体数量进行估计,进一步证明了TarCA的可靠性与泛用性。
最后,研究人员还将TarCA拓展至早期细胞命运决定过程的研究中。在细胞命运决定的初期,群体内部仅有小部分驱动基因的表达发生了变化,在全转录组水平依然保持稳定;随着细胞命运决定的进行,细胞群体内逐渐产生全转录组水平的表达差异。如何在基因水平,而非全转录组水平,对基因表达在细胞群体中的异质性进行检测,是研究早期细胞命运决定过程的关键难题(图2)。为此,该研究使用TarCA算法对基因表达水平在细胞谱系树上的异质性进行了评估,识别出了表达水平与细胞分裂历史事件偶联的基因(lineage-specific upregulated genes, LUGs)。以小鼠早期胚胎的肠管细胞为例,研究人员筛选出了155个LUGs,其中包含众多标记前、中、后肠的明星基因。根据LUGs在不同细胞中的表达情况,研究人员进一步构建出小鼠肠管向下游器官分化过程的层级模型。该模型指出,胰腺、小肠与结肠间存在共享的祖先状态,与体外定向分化实验结果一致。
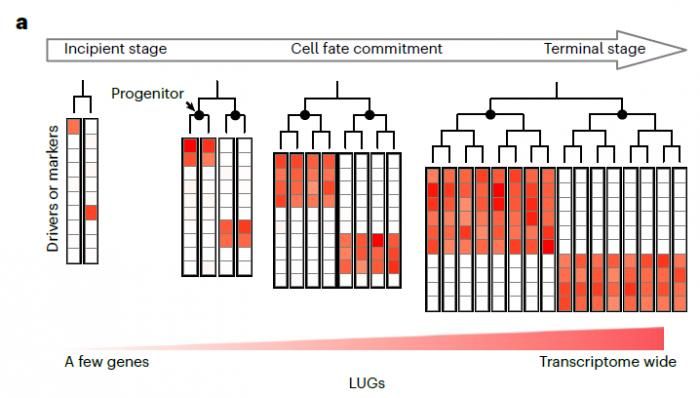
图2 定义细胞命运分配过程中的LUGs
总的来说,该研究开发的TarCA算法可对任意细胞群体估计祖先细胞数目,而无需事先获得祖细胞的先验信息,也能推算少数细胞中的驱动基因,以此来预测早期细胞命运的分化倾向。这些发现说明TarCA能促进包括人类在内的所有复杂生物组织的谱系多样性的量化,对其建立定量模型至关重要,可以帮助人们解决个体发育可塑性和衰老相关的组织功能退化的问题。这项工作是经典进化生物学与现代细胞发育生物学融合的一个典范。
中山大学生命科学学院贺雄雷教授为该论文的通讯作者,课题组博士后邓善俊与博士毕业生龚晗为共同第一作者。该研究得到了国家重点研发计划与国家自然科学基金的资助。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


