Nat Method:单细胞分辨率下对细胞信号交流进行分析的多实例学习模型-Spacia
来源:生物探索 2024-09-06 09:37
Spacia模型能够帮助研究者从各种生物学样品的单细胞空间转录组数据中获得新的知识。
来自德克萨斯大学西南医学中心的王涛博士团队在Nature Methods上发表了标题为Mapping Cellular Interactions from Spatially Resolved Transcriptomics Data的文章。作者开发了名为Spacia的全新的多实例学习(Multi-instance learning) 模型,实现了在单细胞分辨率下对细胞信号交流的分析与推断。不同于其它方法,Spacia 的模型利用单细胞空间转录组 (single cell spatially resolved transcriptomics) 数据中的空间和基因表达信息将细胞信号传输问题转换成为多实例学习问题,再利用贝叶斯统计方法来实现模型的参数推断。
Spacia不依赖于已知的配体-受体对,可以用于探索未知的CCC通道。同时,此模型可以预测单细胞之间的CCC,预测CCC中的正相关以及负相关基因表达关系,并且推测不同CCC的空间特征。

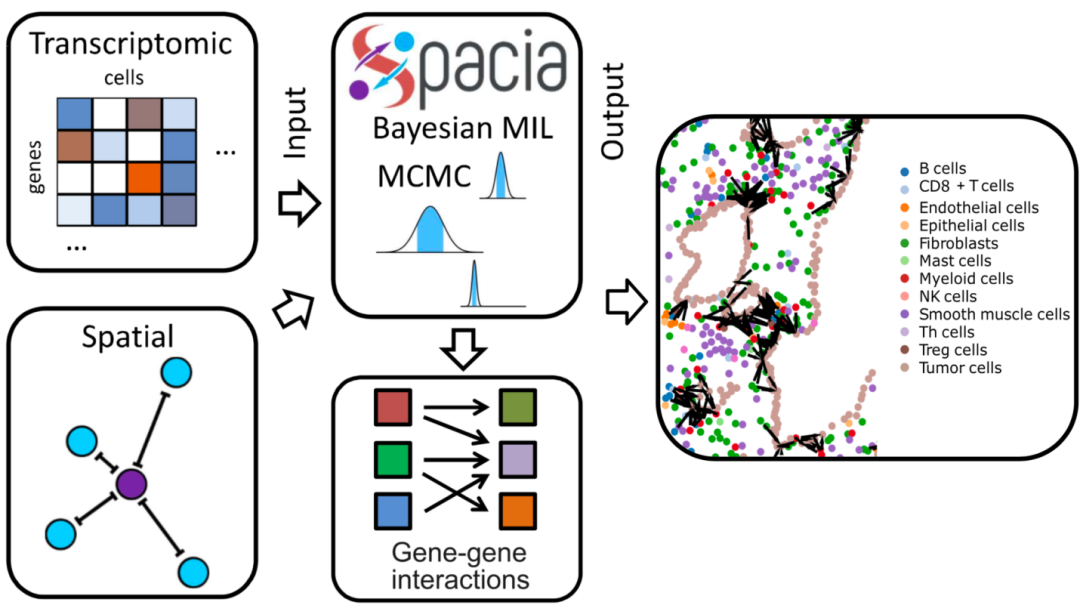
图1:Spacia模型简图。左:Spacia模型的输入输出数据;右:Spacia模型预测的CCC在组织体内的空间分布(Credit: Nature Methods)
Spacia模型能够帮助研究者从各种生物学样品的单细胞空间转录组数据中获得新的知识。作者首先用Spacia模型分析了前列腺癌的肿瘤微环境。他们通过分析发现,癌细胞的上皮-间质转化过程(epithelial-mesenchymal transition,EMT)不仅被纤维化细胞诱导,还可以被周边免疫细胞激发。由于上皮间质转化过程通常和谱系可塑性变化(lineage plasticity)相关。前列腺癌细胞的谱系可塑性现在被认为是激素疗法抗药性的一个重要机理。作者们继续分析了来自这些间质细胞的CCC和不同癌细胞谱系的关系。
研究发现,不同间质细胞可能会选择性诱导几种谱系,并且其诱导强度与细胞的空间分布具有强相关性。接着,作者利用Spacia对由PD-L1介导的肿瘤与CD8+ T细胞之间的信号交流进行了分析,对多种癌症建立了其独特的CD8-PDL1基因表达特征(CD8-PDL1 signature)。他们的后续分析中显示,此特征与病人的生存以及对免疫疗法的响应表现出高度一致性。此结果表明,Spacia可以通过对病人的肿瘤微环境的分析以帮助研究者探寻疾病的生物学机制,寻找新的疾病治疗的靶点,和预测各种治疗方案的效果。
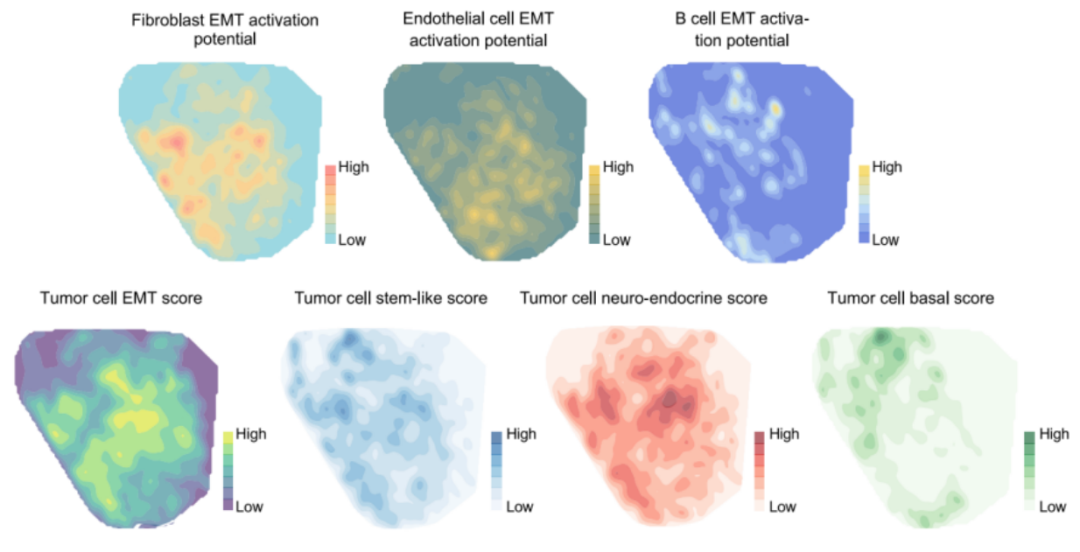
图2:Spacia模型对前列腺癌的肿瘤微环境分析结果。上:Spacia模型所预测的fibroblasts,endothelial cells,和B cells对肿瘤细胞EMT的激活作用强度的空间分布;左下:肿瘤细胞的EMT强度;右下:肿瘤细胞的谱系可塑性强度变化(Credit: Nature Methods)
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


