郭国骥/韩晓平团队发表基于人工智能神经网络的基因组解读系统——女娲
来源:生物世界 2022-10-17 10:48
基于DNA序列编码基因表达模式的假设,该研究提出了深度学习模型Nvwa(女娲),首次实现了完全基于基因组序列预测单细胞水平的基因表达,且预测准确度与实验测量精度相当。
浙江大学基础医学院/浙江省良渚实验室郭国骥/韩晓平团队在 Nature Genetics 期刊发表了题为:Deep learning of cross-species single cell landscapes identifies conserved regulatory programs underlying cell types 的研究论文。
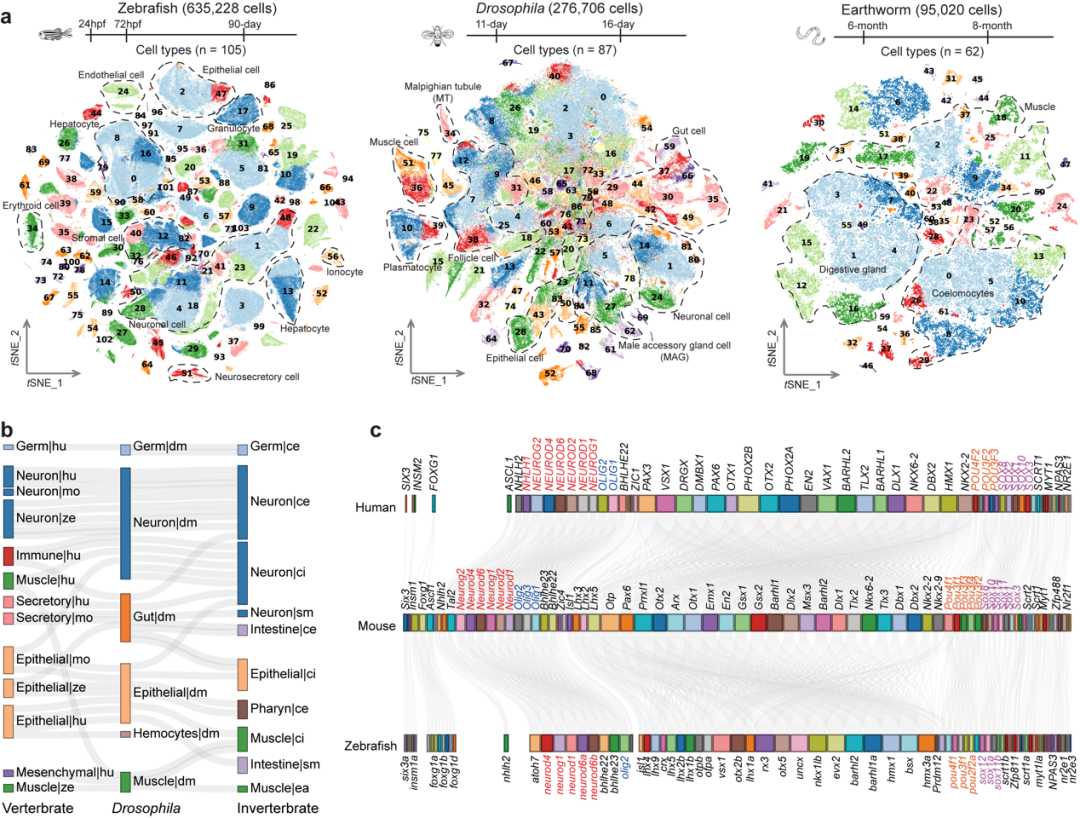
该研究利用自主构建的高通量单细胞测序平台Microwell-seq绘制了斑马鱼、果蝇和蚯蚓的全身单细胞转录组图谱,并探究了八种代表性后生动物细胞类型的跨物种可比性,揭示了脊椎动物细胞类型保守的调控程序。
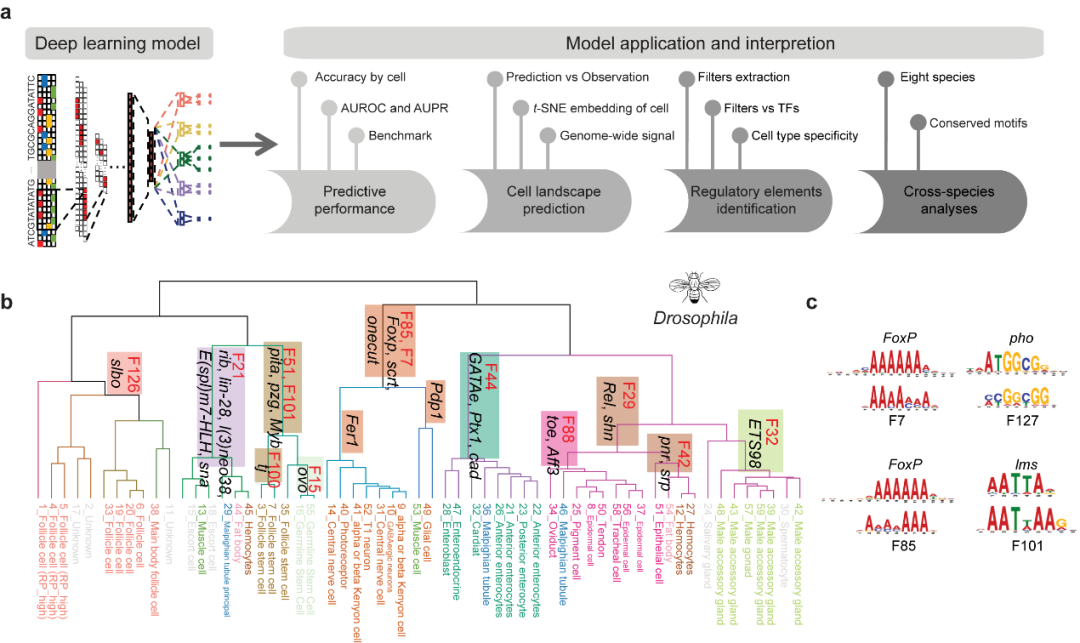
此外,该研究提出了深度学习模型——Nvwa(女娲),首次实现了完全基于基因组序列预测单细胞分辨率下的基因表达。该研究基于Nvwa模型学习衍生的谱系特异性基序,表征了跨物种细胞类型特异性的调节程序。

预测基因表达和解析基因调控机制一直是基因组学的重要目标。尽管研究人员已经努力使用细胞系或组织中的各种实验特征来预测调节信号和基因表达,但在单细胞分辨率下进行生物体规模的表达预测仍然具有挑战性。如今单细胞图谱能够以统一的标准呈现物种细胞的表型,因而人类有机会使用跨物种的单细胞数据来探索进化过程中不同细胞类型的表达和调控程序。
研究团队假设可以直接从基因组序列预测生物体规模的单细胞基因表达,并试图在具有巨大细胞类型多样性的后生动物中检验这一假设。
该研究中,研究人员首先使用其团队自主研发的高通量单细胞测序平台Microwell-seq绘制了斑马鱼、果蝇和蚯蚓的全身单细胞转录组图。其中,斑马鱼图谱收集了635,228个单细胞数据,果蝇图谱涵盖了276,706个单细胞数据,蚯蚓图谱包含了95,020个单细胞数据。该研究利用这三种模式动物的单细胞图谱,并结合其他五种代表性动物的单细胞图谱(人类、小鼠、海鞘、线虫和涡虫),挖掘了跨物种细胞谱系特异性的转录因子,探究了八种代表性后生动物细胞类型的跨物种可比性,揭示了脊椎动物细胞类型,特别是免疫细胞、基质细胞、神经元、上皮细胞、内皮细胞和生殖细胞的保守调节程序。
基于DNA序列编码基因表达模式的假设,该研究提出了深度学习模型Nvwa(女娲),首次实现了完全基于基因组序列预测单细胞水平的基因表达,且预测准确度与实验测量精度相当。
值得注意的是,Nvwa模型可以高度准确地预测几乎所有测试物种的基因表达。此外,通过检查模型第一层的卷积的基序特征Filter,团队揭示了细胞类型特异的基序。这些基序与在特异细胞类型中作用机制明确的转录因子基序相一致。基于模Nvwa模型Filter的跨物种比较,该研究还发现同源Filter倾向于保持跨物种的细胞类型特异性。该工作首次建立了物种层面基因组编码细胞图谱的整合模型,并为解码多物种基因调控程序提供了宝贵资源。
浙江大学基础医学院2019级直博生李佳琦、良渚实验室特聘研究员王晶晶、浙江大学基础医学院博士后张霈婧和汪仁英为本文共同第一作者,浙江大学基础医学院郭国骥教授、韩晓平教授和良渚实验室王晶晶研究员为共同通讯作者。研究获得了国家重点研发计划和国家自然科学基金的支持。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


