Cancer Cell:西湖大学郭天南团队等绘制泛癌种蛋白质组图谱,覆盖22种癌症类型
来源:生物世界 2025-06-02 16:03
该研究利用数据非依赖性采集质谱(DIA-MS)构建了一个大规模泛癌种蛋白质组图谱,覆盖了 22 种癌症类型的 999 例原发肿瘤样本,定量了 9670 种蛋白质。
癌症是一组异质性疾病,其特征是恶性细胞不受控制地生长和扩散,逃脱了正常的检查和平衡机制。肿瘤的分子特征可用于临床干预,以实现个性化治疗。
癌症基因组图谱(TCGA)和国际癌症基因组联盟(ICGC)的大规模基因组学研究旨在编录主要的致癌基因组改变,并提供一份全面的癌症基因组特征“图谱”。对 30 多种人类癌症的大规模队列进行广泛的基因组测序,并结合对单个癌症类型的综合分析以及全面的泛癌分析,揭示了细胞异常的非常复杂的图景,拓展了当前对肿瘤发生机制的认识,并创新了分子诊断方法。尽管取得了这些进展,但要预测肿瘤中的基因组/表观基因组和转录组改变如何影响下游蛋白质的表达和活性,仍颇具挑战性。
蛋白质是细胞中的功能单位,也是大多数疗法的作用靶点。因此,仅依据基因组/表观基因组和转录组特征来选择靶向治疗,仍有改进空间。蛋白质组不仅能够拓展我们对细胞动态分子行为的理解,帮助揭示癌症表型的形成机制,还可能改善诊断和治疗选择。然而,迄今为止,大多数癌症蛋白质组学研究都集中在单一癌症类型上。
2025 年 5 月 29 日,阿姆斯特丹自由大学 Connie Jimenez 团队联合西湖大学医学院郭天南团队,在 Cancer Cell 期刊发表了题为:The pan-cancer proteome atlas, a mass spectrometry-based landscape for discovering tumor biology, biomarkers, and therapeutic targets 的研究论文。
该研究利用数据非依赖性采集质谱(DIA-MS)构建了一个大规模泛癌种蛋白质组图谱(Pan-Cancer Proteome Atlas,TPCPA),覆盖了 22 种癌症类型(18 种实体瘤和 4 种非实体瘤)的 999 例原发肿瘤样本,定量了 9670 种蛋白质,系统分析了癌种间和同种癌症内的蛋白表达特征,识别出多个泛癌与特异性蛋白标志物、潜在靶点以及癌症亚型分类器。此外,研究团队还开源了全部数据资源。

该研究基于数据非依赖性采集质谱(DIA-MS)技术构建了泛癌种蛋白质组图谱(Pan-Cancer Proteome Atlas,TPCPA),以更好地理解癌症生物学并识别治疗靶点和生物标志物。
泛癌种蛋白质组图谱(TPCPA)包含了来自 999 个原发性肿瘤的 9670 种蛋白质,这些肿瘤代表了 22 种癌症类型。
研究团队进一步描述了在泛癌种及特定癌症类型中富集的蛋白质,并进行了广泛的外部注释,优先确定候选药物靶点和生物标志物。
对于蛋白靶向降解嵌合体(PROTAC)而言,该研究确定了在特定肿瘤类型中高表达的 E3 泛素连接酶,包括 HERC5(在食管癌中高表达)和 RNF5(在肝癌中高表达)。
该研究还共表达分析揭示了 13 个模块,其中包括一些意想不到的枢纽蛋白,它们可能是潜在的药物靶点,例如 GFPT1、LRPPRC、PINK1、DOCK2 和 PTPN6。
通过对 195 例结直肠癌的分析,该研究确定了基于 RNA 的共识分子亚型(CMS)和两种具有预后价值的免疫亚型。
此外,该研究还构建了一个基于 75 个蛋白的癌症亚型分类器,在内部验证及 4 个独立队列(包括转移癌)中均表现优异(AUC大于0.98),可用于识别原发灶不明的癌症。
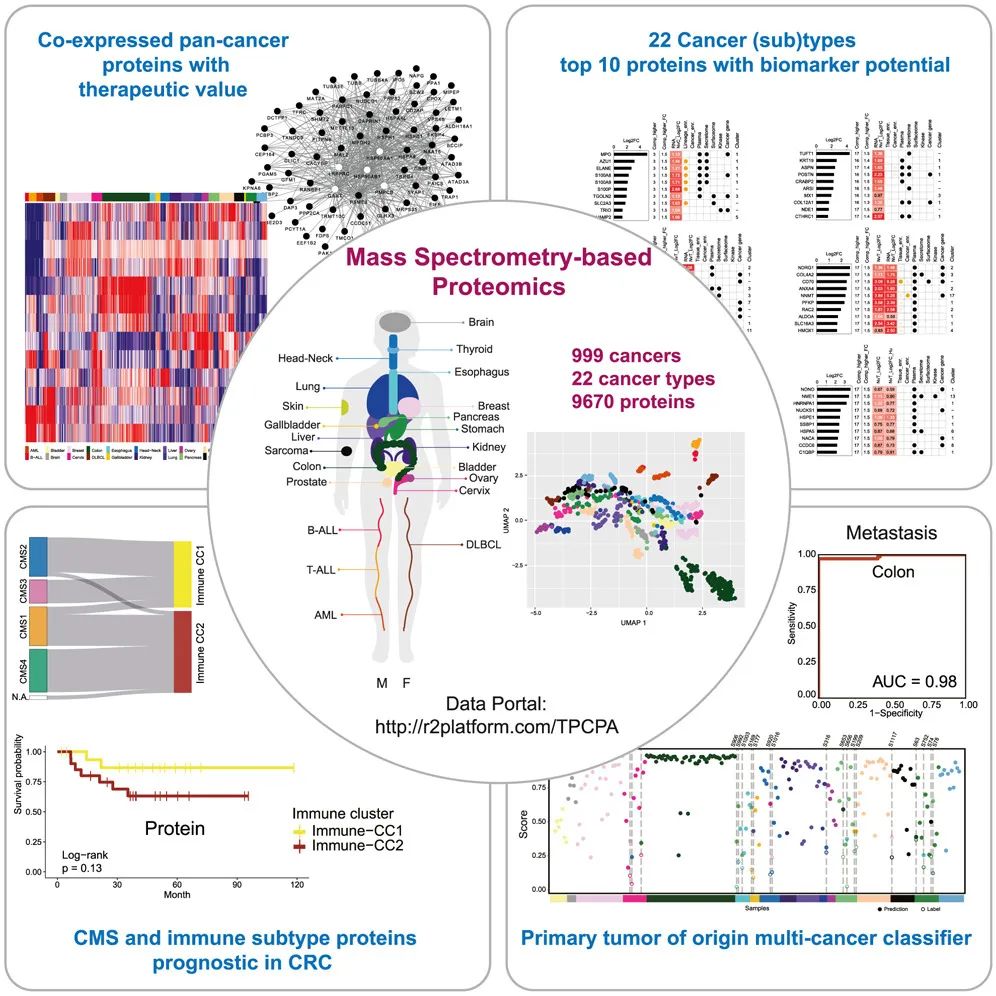
最后,研究团队开源了所有的 TPCPA 数据,这些数据均可在门户网站 http://r2platform.com/TPCPA/ 中免费查询和使用。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


