遗传与发育生物学研究所研究发布拟南芥高质量参考基因组
来源:遗传与发育生物学研究所 2022-06-06 14:31
Col-PEK组装具有较高的序列完整度,对Col-PEK的注释揭示了重复序列的分布规律,特别是着丝粒区域的CENH3结合区域分布规律和CEN180重复序列分布特征。
拟南芥(Arabidopsis thaliana)作为被广泛应用的模式植物,其基因组序列加快了植物分子生物学研究。在首个基因组发布二十余年后,仍存在大量未填补的缺口区域。在常用的TAIR10/Araport11版本的基因组序列中,存在165个缺口。这些缺失区域可能由高度重复的序列组成,包括端粒、着丝粒、5S rDNA簇和含有45S rDNA的核仁组织区(NORs)。近年来,ONT和PacBio等长读段测序技术的发展为组装高复杂度序列提供了有力工具。近期发表的两个高完整度基因组Col-CEN和Col-XJTU填补了着丝粒等缺失区域,但这两个新的组装仍不完整且有相互矛盾之处。
为提供更好的参考基因组,中国科学院遗传与发育生物学研究所焦雨铃研究组与中国科学院大学汪颖研究组合作,结合长读ONT、高保真的长读PacBio HiFi和短读Illumina NovaSeq测序数据获得了接近完整的拟南芥Col-0生态型的参考基因组Col-PEK。Col-PEK组装填补了包括五个着丝粒在内各区域中的绝大多数缺口。例如,Col-CEN中5号染色体中的缺口均已在Col-PEK中补齐。Col-PEK为目前最为完整的基因组组装,完成了1、3、5号染色体从端粒到端粒的完整组装,仅2号和4号染色体的多拷贝NORs区域尚不完全。Col-PEK组装总长度133.92 Mb,比TAIR10组装长14.77 Mb,即增加了12.4%的序列。在填补缺口之外,Col-PEK还修订了Col-CEN等组装中的拼接错误。
Col-PEK组装具有较高的序列完整度,对Col-PEK的注释揭示了重复序列的分布规律,特别是着丝粒区域的CENH3结合区域分布规律和CEN180重复序列分布特征。对编码基因的注释还发现了145个新的“隐藏基因重复”,其与已知基因序列高度相似,可能是由新近的串联重复等基因组扩增机制所产生。
Col-PEK组装补全了所有着丝粒序列及绝大部分其它缺口,纠正了之前的错误组装。该研究的初步分析展示了重复序列的分布规律,并揭示了一批新基因。Col-PEK参考基因组为植物学科研工作者提供了新的参照序列和重要数据资源。
相关研究成果以A near-complete assembly of an Arabidopsis thaliana genome为题,于6月1日在线发表在Molecular Plant上(DOI:10.1016/j.molp.2022.05.014)。研究工作得到国家重点研发计划的资助。
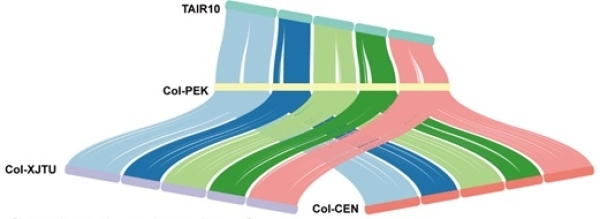
图1.不同参考基因组组装的完整度比较
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


