科研人员绘制图谱解析风险SNP驱动的促肿瘤机制
来源:中山大学第一医院 2025-10-06 16:29
吕万革教授团队联合张弩教授团队系统性绘制胶质瘤功能性增强子互作图谱,解析风险SNP驱动的促肿瘤机制。本研究提供了胶质瘤增强子连接组资源,阐明了非编码变异如何重塑远程调控并推动肿瘤进展。
附属第一医院吕万革教授团队联合张弩教授团队系统性绘制胶质瘤功能性增强子互作图谱,解析风险SNP驱动的促肿瘤机制,相关研究成果发表于《Nature Cell Biology》(自然-细胞生物学)。
神经胶质瘤(Glioma)是侵袭性强的中枢神经系统肿瘤,五年生存率不足10%。尽管全基因组关联研究(GWAS)已发现多个与风险相关的非编码区域的单核苷酸多态性(SNP),其致病机理仍不清楚。
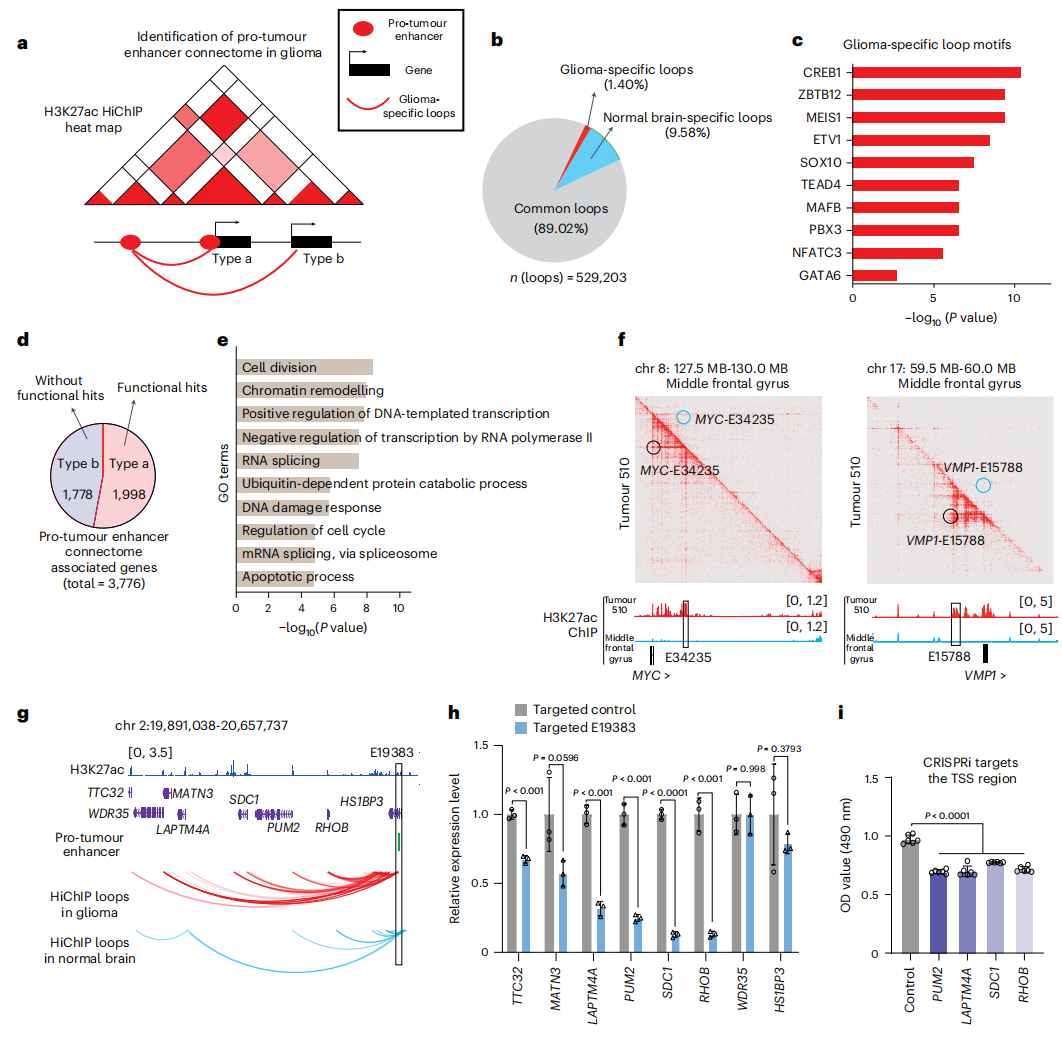
该研究通过结合高通量CRISPRi与H3K27ac HiChIP技术,系统绘制胶质瘤增强子互作网络,精准配对促肿瘤增强子与远端靶基因。团队发现多数已报道风险SNP位于增强子。以rs2297440为例,该位点所在增强子虽位于RTEL1内含子区域,却通过三维染色质与SOX18相互作用驱动其表达;敲除增强子或将风险等位基因单碱基编辑为非风险形式,均降低增强子互作与肿瘤细胞增殖。机制上,转录因子MEIS1偏好结合风险等位基因并上调SOX18,形成“rs2297440–MEIS1–SOX18”促肿瘤机制。
本研究提供了胶质瘤增强子连接组资源,阐明非编码变异如何重塑远程调控并推动肿瘤进展,为精准治疗提供潜在靶点。
附属第一医院吕万革教授、张弩教授和苏光松副教授为该论文的通讯作者,附属第一医院博士后毕进方、莫伟鹏、南开大学已毕业博士生刘满为该论文共同第一作者。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


