Nature:ecDNA“抱团行动”促使癌基因表达扩增
来源:生物探索 2021-12-08 16:32
一直以来大家都默认基因都是在染色体上,染色体外DNA(ecDNA)这一概念的出现可以说是颠覆了大家的传统认知,原来研究了半天,竟是搞错了方向,癌基因其实并不在我们关注的染色体上。由此,癌症研究打开了一扇新大门。ecDNA是一种存在于染色体外,高度开放的DNA 颗粒,普遍存在于人类癌细胞中。因其能通过基因扩增和改变基因调控来介导癌基因的
一直以来大家都默认基因都是在染色体上,染色体外DNA(ecDNA)这一概念的出现可以说是颠覆了大家的传统认知,原来研究了半天,竟是搞错了方向,癌基因其实并不在我们关注的染色体上。由此,癌症研究打开了一扇新大门。
ecDNA是一种存在于染色体外,高度开放的DNA 颗粒,普遍存在于人类癌细胞中。因其能通过基因扩增和改变基因调控来介导癌基因的高表达,被认为与肿瘤的异质性和耐药性相关。
编码致癌基因的ecDNA是癌症基因组的普遍特征,也是癌症进展的一个强有力的驱动因素。ecDNA是共价闭合双链,大小从100千碱基到几兆碱基不等。因缺乏着丝粒,ecDNA在细胞分裂过程中会随机分布于子细胞,这就方便了其快速积累,而且可以重新整合到染色体中,因此也可作为某些染色体扩增的前体。ecDNA具有更高的染色质可及性,但却缺乏更高的染色质致密性,且包含内源性致癌基因增强子元件,这表明癌基因扩增子可能是通过调控依赖性来扩增转录的。因其本身存在于染色体外,目前对其细胞核中的空间还不清楚。ecDNA在细胞分裂期间或DNA损伤后会相互聚集,但其聚集后生物学后果目前还不甚了解。
斯坦福大学HowardChang教授团队在《Nature》发表了题为“ecDNAhubs drive cooperative intermolecular oncogene expression” 的论文,他们研究了致癌ecDNA的细胞核空间、表观遗传因子和转录调控,发现聚集在间期细胞核中的ecDNA可以“抱团行动”,驱动分子间增强子信号来促使癌基因表达扩增。
ecDNA促进癌症基因表达
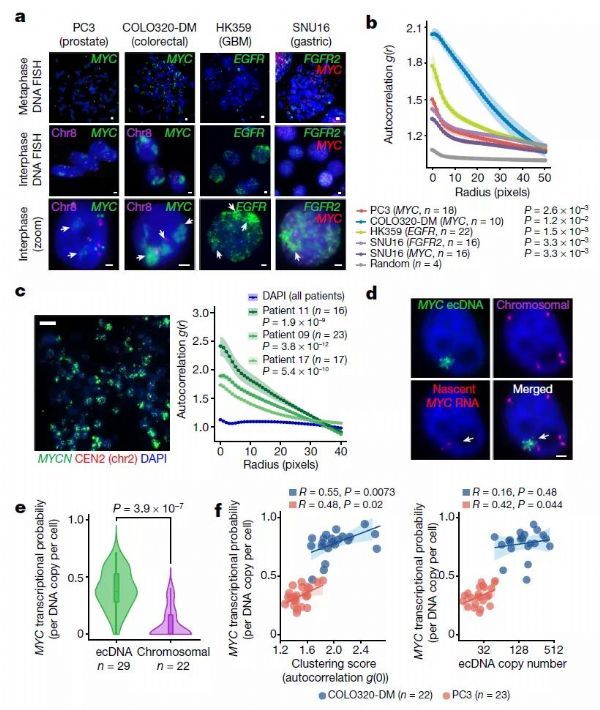
研究团队通过DNA荧光原位杂交(FISH)来观察间期细胞核中的ecDNA的聚集点,包括前列腺癌细胞系PC3(MYC扩增)、结直肠癌细胞系COLO320-DM(MYC扩增)、多形性成胶质细胞瘤细胞系HK359(EGFR扩增)和胃癌细胞系SNU16(MYC和FGFR2扩增)。结果显示,所有ecDNA阳性癌细胞中,除去有数十到数百个单独的ecDNA分子,ecDNAFISH基本都集中在间期细胞核中,这表明ecDNA彼此之间有强烈的聚集,研究团队将其称为ecDNA中心。进一步实验后发现,在具有不同癌基因扩增的各种癌症类型和原发性肿瘤中均存在ecDNA聚集。
研究人员利用DNA和新生RNAFISH,在PC3和COLO320-DM细胞中观察到活跃转录的MYC等位基因,并计算出每个ecDNA分子的MYC转录概率。大多数新生MYCmRNA转录本来自ecDNA中心,而不是来自染色体。此外,与单子ecDNA相比,ecDNA中心的转录活性更高。因此,ecDNAs中心聚集时,每个ecDNA分子更有可能转录致癌基因。
BRD4连接ecDNA中心和转录
癌基因MYC包含MYC和浆细胞瘤转化迁移基因1(PVT1,拷贝数扩增相关的lncRNA,已知的癌症相关区域),两侧是由组蛋白H3乙酰化赖氨酸27(H3K27ac)和BET蛋白(如BRD4)为标记的超级增强因子。为了检查活细胞中的MYCecDNA,研究人员在COLO320-DM细胞的MYCecDNA中插入了Tet-operator(TetO)阵列,并用Tet-eGFP标记了ecDNA,以尽量减少GFP二聚化。活细胞成像显示,有多个动态核病灶对应ecDNA聚集。内源性BRD4的表位标记显示,BRD4在TetO标记的ecDNA中心高度富集。通过对H3K27ac和BRD4进行测序和染色质免疫沉淀来分析染色质,结果显示标记活性H3K27ac的ecDNA也被BRD4占据。
为了确定BET蛋白在ecDNA相关转录中的作用,研究人员重点研究了同源的结直肠癌细胞系COLO320-DM(MYCecDNA)和COLO320-HSR(来源于同一患者肿瘤)。TetO-GFPCOLO320-DM细胞中进行的活细胞成像显示,ecDNA中心在有丝分裂过程中被分解成更小的颗粒。分解后,ecDNAs又再次聚集成大中心。值得注意的是,有丝分裂后,ecDNA中心重新聚集被JQ1(BRD4抑制剂)阻断。结果表明,在COLO320-DM细胞中,ecDNA中心的形成、维持和肿瘤基因转录对BET蛋白H3K27ac相互作用有独特的依赖性。
ecDNA中心反式激活
为了将ecDNA结构与MYC转录调控联系起来,研究团队使用五种正交方法重建了COLO320-DM的ecDNA,报告了目前已知的组装的最大的ecDNA结构,包含PVT1-MYC融合、标准MYC序列和来自多个染色体起源的序列(6、8、13和16号染色体)的多个拷贝,并且利用DNA FISH验证了PLUT、PCAT1和MYC基因在重建预测的ecDNA上的共定位。
研究团队在COLO320-DM细胞中观察到了PVT1启动子处的强BRD4结合,但在COLO320-DMecDNA细胞中未观察到。因PVT1启动子能被MYC激活,研究团队又假设PVT1-MYC融合可实现MYC表达的正反馈,避免PVT1和MYC启动子之间的过度表达,但在几种人类癌症中观察到了PVT1重排和基因融合并驱动基因过度表达。
接下来,研究人员确定了与癌基因高表达相关的ecDNA调控元件。72049个来自COLO320-DM和COLO320-HSR细胞的配对单细胞ATAC–seq和RNA-seq,经校正MYC拷贝数后确定了47个与高MYC表达相关的ecDNA调控元件,而目前驱动ecDNA上MYC癌基因表达的PVT1启动子(PVT1p)在ecDNA中心内接受了广泛的组合增强子输入。进一步实验表明,分子间增强子-启动子在ecDNA中心激活,同时研究人员证实PVT1p作为一种DNA元件,能够反式激活ecDNA中心。
ecDNA间的分子调控
研究人员进一步研究了分子间增强子-基因相互作用是如何进行精确定位和干扰。以人胃癌细胞系SNU16为研究对象,其包含8号和11号染色体的MYC扩增子和10号染色体的成纤维细胞生长因子受体2(FGFR2)扩增子。H3K27ac的染色质免疫沉淀和测序显示FGFR2和MYCecDNA之间存在分子间接触,并富集了焦点相互作用。CRISPR干扰(CRISPRi)靶向候选调控元件确定了与MYC或FGFR2在顺式和反式表达相关的功能元件。
实验结果表明,FGFR2和MYCecDNA已被共同选择,因此这两个扩增子上的增强子可协同激活MYC表达。MYC蛋白继而激活FGFR2表达。顺式和反式调控元件之间几乎没有重叠,表明分子间增强子元件是直接修饰反式基因表达,而非通过下游效应。癌症类型中的分子间ecDNA的相互作用评估显示,ecDNA中心的分子间增强子-基因激活发生在不同的肿瘤基因位点和多种癌症类型。
ecDNA中心介导分子间增强子-基因的相互作用
综上所述,上述研究显示ecDNA“抱团行动”促进了新的分子间增强子-基因相互作用和癌基因过表达,且可以发生在不同的肿瘤基因位点和多种癌症类型中。与传统的顺式染色体转录不同,ecDNA中心反式调控能够实现分子间转录调控,这对如何实现更好的癌基因转录具有重要的启示作用。未来的研究可能会发现在各种癌症类型中介导ecDNA转录活性的蛋白质,并可将其作为潜在的治疗靶点,癌症研究也将进一步进阶到新领域。(生物谷Bioon.com)
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。