Horticulture Research:植物基因组LTR类转座子分类研究方面取得进展
来源:昆明植物所 2022-02-22 08:02
转座子是构成基因组重复序列的主要成分。越来越多的研究表明,转座子在决定基因组大小、基因组结构变异、序列突变、基因丢失、基因融合和新编码基因的起源方面,都具有重要的生物学意义。LTR类转座子是植物基因组中占比最高的重复序列类型,它是逆转录转座子的一种。然而目前大部分软件对LTR类转座子仅停留在超科水平(superfamily level),没有提供
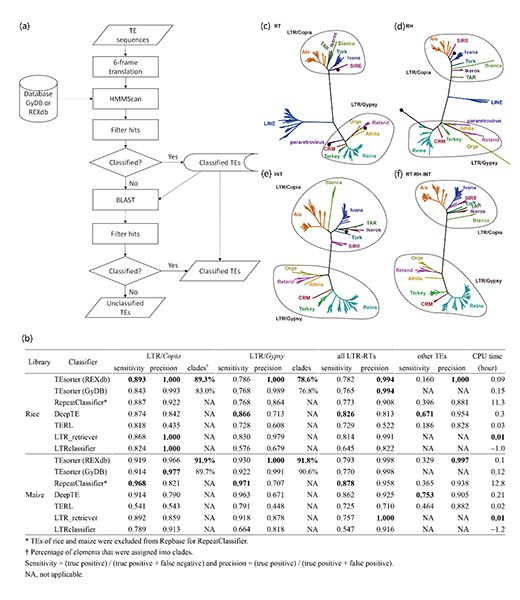
转座子是构成基因组重复序列的主要成分。越来越多的研究表明,转座子在决定基因组大小、基因组结构变异、序列突变、基因丢失、基因融合和新编码基因的起源方面,都具有重要的生物学意义。LTR类转座子是植物基因组中占比最高的重复序列类型,它是逆转录转座子的一种。然而目前大部分软件对LTR类转座子仅停留在超科水平(superfamily level),没有提供更细致的分类,无法反映LTR类转座子的多样性和进化关系。
基于此,中国科学院昆明植物研究所等研究团队开发出LTR类转座子更加准确、快速分类的新方法,命名为TEsorter。该方法是以已经分类的保守蛋白结构域(数据库来源REXdb或GyDB)为数据基础,采用hidden Markov models (HMMs)方法,对玉米和水稻基因组中转座子,尤其是LTR类转座子进行准确分类。大部分LTR类转座子可达到分支水平(clade level),分支水平的分类结果和基于系统发育树的聚类高度一致。同时,通过对比现在常用的5个转座子分类软件(RepeatModeler、DeepTE、TERL、LTR_retriever和LTRclassifier),发现TEsorter不论在准确率还是运算速度方面,都具有明显优势。研究成果以TEsorter: an accurate and fast method to classify LTR-retrotransposons in plant genomes为题在线发表于Horticulture Research。(生物谷Bioon.com)
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


