研究揭示拟南芥RNA聚合酶V的转录延伸机制
来源:分子植物卓越中心 2023-06-06 16:41
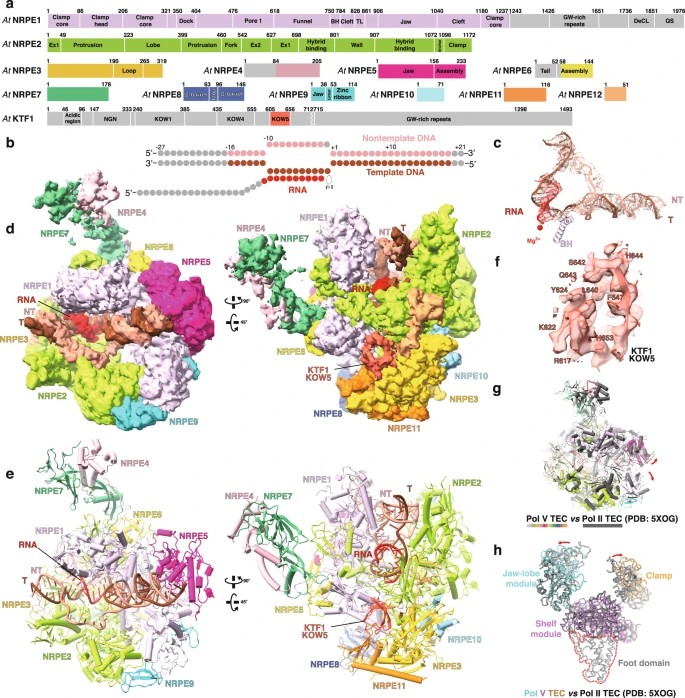
Pol V-KTF1转录延伸复合物的冷冻电镜结构显示,Pol V具有和Pol II类似的活性中心,但Pol V活性中心的结构单元和Pol II具有构象差异。
《自然-通讯》(Nature Communications)在线发表了中国科学院分子植物科学卓越创新中心/中科院合成生物学重点实验室张余研究组撰写的题为A cryo-EM structure of KTF1-bound polymerase V transcription elongation complex的研究论文。该研究报道了包含KTF1的RNA聚合酶V(RNA polymerase V; Pol V)转录延伸复合物的高分辨率冷冻电镜结构,为剖析Pol V转录延伸奠定了结构基础。
陆地植物通过RNA介导的DNA甲基化通路(RNA-directed DNA methylation pathway,RdDM)建立和部分维持基因组DNA甲基化。该通路中,基因组DNA甲基化与Pol V的转录延伸过程紧密偶联,Pol V转录延伸过程通过两个层次的相互作用招募Argonaute4/6-siRNA复合物。第一个层次是Pol V-KTF1和Argonaute 4/6蛋白的直接相互作用。该相互作用涉及Pol V和KTF1中包含GW基序的结构域。第二个层次是Pol V转录产生的long non-coding RNA与Argonaute4/6携带的siRNA序列匹配。进而,上述复合物进一步招募DNA甲基化转移酶DRM2在Pol V的转录区域进行DNA甲基化修饰(图1)。Pol V由Pol II进化而来,但其转录活性低于Pol II,其转录的基因组区域以及相互作用的转录调控蛋白和Pol II有显著区别。
Pol V-KTF1转录延伸复合物的冷冻电镜结构显示,Pol V具有和Pol II类似的活性中心,但Pol V活性中心的结构单元和Pol II具有构象差异。研究推测这一差异与Pol V的较弱转录活性相关。该研究进一步表明Pol V具有独特的结构特征,而该特征阻止其与Pol II的通用转录因子(TFIIB、TFIID、TFIIE、TFIIH、TFIIS)以及Pol IV的转录偶联因子(RDR2)相互作用,解释了Pol V相较Pol II以及Pol IV功能的独特性。此外,该研究揭示了KTF1的KOW5结构域和Pol V之间的相互作用,KTF1的KOW5结构域结合在Pol V的RNA退出通道附近。这一结合方式使KTF1 的C端GW结构域指向Pol V转录RNA的5’末端,从而便于其招募携带siRNA的AGO4/6。该成果为Pol V的转录特征提供了结构解释,为进一步研究Pol V转录延伸偶联的DNA甲基化过程奠定了基础。
研究工作得到国家重点研发计划和上海市基础研究特区计划项目的支持。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


