Cell:开发出更好的方法从海量序列中寻找RNA病毒
来源:生物谷原创 2022-10-12 16:54
在一项新的研究中,研究人员描述了一个可以专门扫描RNA病毒序列的计算管道。利用这一工作流程,他们梳理了来自世界各地不同环境样本的5000多个RNA序列数据集(宏转录组),使RNA病毒的多样性增加了五倍
一家动物园曾经提供过一本彩色画册,画的是北极熊在冬天的场景,配有各种深浅不一的白色蜡笔。对于在大型数据集中寻找RNA病毒序列的科学家们来说,他们的工作可能类似于在那本书的彩色页面上寻找一片雪花。

在一项新的研究中,来自以色列特拉维夫大学、美国国家生物技术信息中心(NCBI)和美国能源部联合基因组研究所(JGI)的研究人员描述了一个可以专门扫描RNA病毒序列的计算管道。利用这一工作流程,他们梳理了来自世界各地不同环境样本的5000多个RNA序列数据集(宏转录组),使RNA病毒的多样性增加了五倍。相关研究结果于2022年9月28日在线发表在Cell期刊上,论文标题为“Expansion of the global RNA virome reveals diverse clades of bacteriophages”。
在谈及发现的病毒多样性时,论文共同通讯作者、NCBI高级研究员Eugene Koonin说,“我们周围的病毒世界是巨大的,我们现在有了探索它的手段。尽管这种规模的数据分析面临的技术挑战是非常艰巨的。”
用于过滤序列的计算筛子
地球上的微生物比一把泥土中的颗粒还要多,而病毒的数量远远超过微生物。测序技术和计算工具的进步已发现了病毒的多样性,这些病毒不仅感染作物、动物和人类,而且还感染微生物,它们的存在或不存在会影响地球的营养循环。
虽然大多数有机体的遗传信息是在DNA中编码的,RNA将DNA内的指令传递给细胞,但RNA病毒将它们的遗传信息储存在RNA中,而不是储存在DNA中。论文共同作者、JGI 科学家Simon Roux说,“我认为RNA病毒在全球范围内甚至比DNA病毒更不为人所知。但与DNA病毒一样,RNA病毒在全世界范围内感染微生物,并在感染期间导致细胞死亡和/或细胞生理学的深刻变化。”
虽然所有的RNA病毒都有一个编码称为RNA引导的RNA聚合酶(RDRP)的基因,这是进行RNA基因组复制所必需的,但检测它一直是一个挑战。在海量的基因组数据中找到RNA病毒,需要开发特殊的计算筛子(computational sieve)来过滤掉不太可能包含RdRP序列的序列。
论文第一作者兼论文共同通讯作者、特拉维夫大学的Uri Neri回忆说,这项新的研究是2019年开始的三方合作的结果。特拉维夫大学的研究团队和NCBI团队的成员已经在一起合作分析原核生物病毒(噬菌体),他们从JGI的Nikos Kyrpides那里得知,Kyrpides的微生物组数据科学小组也在致力于分析RNA病毒。在这三个团队的几次视频会议之后,很明显,与较小的个人努力相比,更大的合作努力在取得更高质量的结果方面要有效得多。
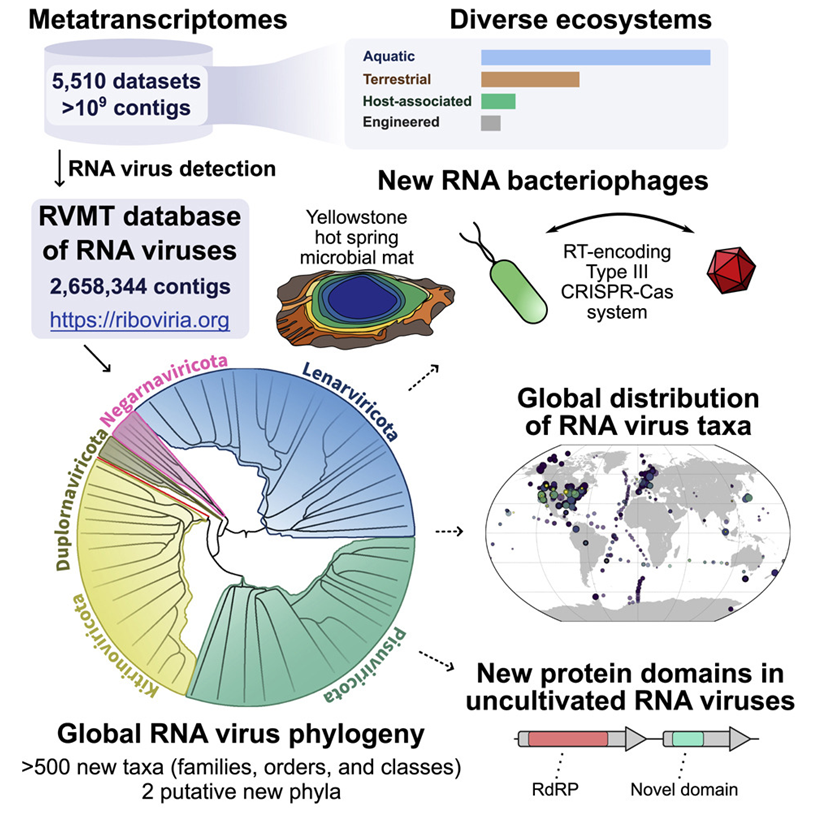
图片来自Cell, 2022, doi:10.1016/j.cell.2022.08.023。
这些作者使用了JGI的综合微生物基因组与微生物组(IMG/M)系统中所有公开的宏转录组数据集。Neri说,“我们随后研究了更多的样本并完善了我们的方法。我们的团队不断壮大,项目的范围也在不断扩大。”为此,Kyrpides强调,众多JGI科学用户在收集和提交他们的微生物组样本在JGI进行测序方面的贡献怎么强调都不过分。他说,他们的合作和支持,以及在一些情况下,他们允许使用尚未公布的序列数据,对于这项新研究的成功绝对是至关重要的,对他们贡献的承认也是如此。
Roux和Koonin都指出,所发现的大量RNA病毒序列“极大地改变了全球病毒多样性的观点”,尽管不是在更高层次的病毒群体(门)分类中。此外,RNA病毒似乎并不是均匀地分布在世界各地。
一个扩大的病毒群体是与细菌有关的病毒;直到现在,大多数已知的RNA病毒都与真核生物有关。Roux指出,伴随着与细菌相关的RNA病毒的扩大,发现“少数细菌使用CRISPR来防御RNA,尽管不清楚为何这种情况很少被检测到。”
开发协调“真实”大数据的方法
对于这些作者来说,导致发现丰富的RNA病毒的计算工作只是一个开始。Neri说,“我经常说,仅仅确定一个序列是病毒的,甚至还不是故事的一半。我们在发现后的分析中投入了大量的精力---我们尽可能地描述每一种病毒所携带的蛋白结构域,以及谁是它们可能的宿主。我们已经将所有这些信息完全免费并公开提供给更广泛的科学界。”
Koonin和特拉维夫大学的Uri Gophna都指出,其他平行的研究报告了全球RNA病毒组的类似“急剧扩张”。Koonin说,“我们如今需要比较和协调这些发现,提出一个单一的、非冗余的数据集。希望在相对较短的时间内,我们将能够估计出RNA病毒组(RNA virome)的实际规模。然而,这如今是真正的大数据,我们正在处理数十亿个序列,很快就会有数万亿个序列。开发高效、自动化的方法来分析和分类这种规模的序列数据是至关重要的。” (生物谷 Bioon.com)
参考资料:
1. Uri Neri et al. Expansion of the global RNA virome reveals diverse clades of bacteriophages. Cell, 2022, doi:10.1016/j.cell.2022.08.023.
2. A better way to find RNA virus needles in database haystacks
https://phys.org/news/2022-10-rna-virus-needles-database-haystacks.html
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


