阿里云发布LucaOne模型,首次统一DNA/RNA和蛋白质语言,能够理解中心法则
来源:生物世界 2025-06-21 10:08
LucaOne 的出现,标志着生物信息学正在进入一个由大型通用基础模型驱动的新时代。
生命的语言以 DNA、RNA 和蛋白质的形式编码,构成了生命的基石,但由于其复杂性,解读起来颇具挑战。传统的计算方法往往难以整合这些分子的信息,从而限制了对生物系统的全面理解。
自然语言处理(NLP)技术的进步,尤其是预训练模型的发展,为解读生命的语言带来了新的可能。想象一下,如果存在一种“翻译器”,能够像我们理解人类语言一样,读懂构成生命的核心“语言”——DNA、RNA 和蛋白质序列中蕴含的复杂信息,那将会怎样?
2025 年 6 月 18 日,阿里云智能飞天实验室李兆融、贺勇及中山大学施莽教授等,在 Nature 子刊 Nature Machine Intelligence 上发表了题为:Generalized biological foundation model with unified nucleic acid and protein language 的研究论文。
该研究开发了广义生物学基础模型——LucaOne,这是世界首个能够同时理解并统一处理核酸(DNA和RNA)和蛋白质序列的基础模型,堪称生命科学领域的“DeepSeek”。
LucaOne 在基于 169861 种物种的核酸和蛋白质序列进行了预训练,通过大规模数据整合和半监督学习,LucaOne 展现出了对诸如 DNA 翻译为蛋白质等关键生物学原理的理解。利用少样本学习,它能够有效地理解分子生物学的中心法则,并在涉及 DNA、RNA 或蛋白质输入的任务中表现出色。我们的研究结果突显了统一基础模型在解决复杂生物学问题方面的潜力,为生物信息学研究提供了一个灵活的框架,并有助于更好地解读生命的复杂性。

生命语言的复杂性:为何需要新工具?
从 DNA 的发现到对各种生物形式的测序,生物序列信息从 DNA 到 RNA 再到蛋白质的忠实且基于规则的流动一直是生命科学的核心原则——“中心法则”,即 DNA 携带遗传信息,转录成 RNA,再翻译成蛋白质执行功能。
这三种主要的信息承载生物大分子在细胞内承担了大部分工作,进而决定了各种生物体的结构、功能和调节机制。它们本质上都是线性排列的“字母”序列:DNA 和 RNA 均由 4 种核苷酸组成,前者是 A、T、C、G,后者是 A、U、C、G;蛋白质则由 20 种标准氨基酸以及少量非标准氨基酸组成。
正如达尔文在其著作《人类的由来》中所写:不同语言的形成以及不同物种的产生,以及两者都是通过一个渐进的过程发展起来的证据,竟如此惊人的相同。此后,各种研究都证实了这些相似之处,促进了对生命的语言的理解和破译。
就像人类的语言有其语法和语义一样,这些核酸(DNA、RNA)和蛋白质序列的排列组合、以及它们折叠形成的复杂结构,编码了生命的所有秘密(结构、功能、调控)。然而,传统计算方法往往只能孤立地分析其中一种分子(例如只分析蛋白质或只分析 DNA),难以整合三者之间的复杂关系(例如 RNA 如何精确翻译成特定蛋白质),限制了我们对生命系统的全面理解
LucaOne:生命语言的“通才”模型
LucaOne 正是为了解决这一挑战而诞生。它的核心思想借鉴了自然语言处理(NLP)领域的革命性突破——Transformer 架构(这也是 ChatGPT 的基础架构)和“基础模型”(Foundation Model)的概念。
海量数据训练: 研究团队构建了前所未有的庞大训练数据集,涵盖了 169861 个物种的核酸和蛋白质序列,数据来源包括权威数据库,例如 RefSeq(基因数据库)、UniProt(蛋白质数据库)、ColabFoldDB(蛋白质折叠数据库)等。
统一“词汇表”: LucaOne 拥有一个包含 39 个“字符”的词汇表,将核苷酸和氨基酸统一编码,从而能同时“阅读”核酸和蛋白质的“句子”。
半监督学习: 除了让模型像“完形填空”一样预测被掩盖的序列片段(自监督学习),研究团队还巧妙地融入了已知的生物学注释信息(例如基因组区域类型、蛋白质结构域、物种分类等)进行半监督学习。这相当于在让模型自学的同时,也给它一些“生物学教科书”作为参考,加速其理解。
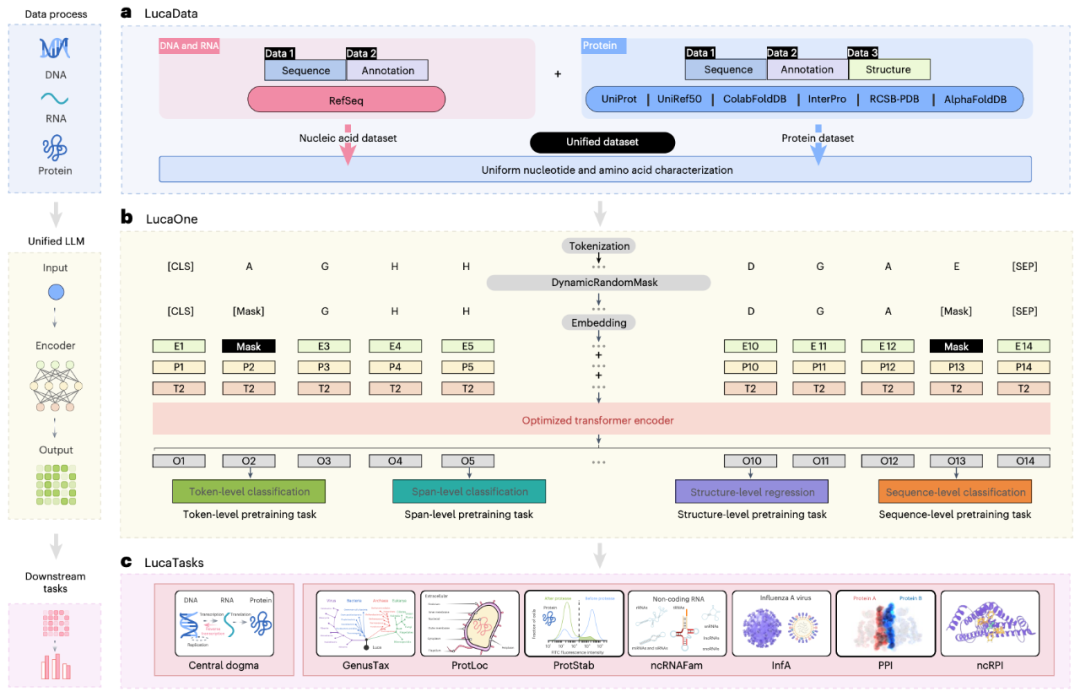 LucaOne 的工作流程
LucaOne 的工作流程
LucaOne 拥有 18 亿参数,是一个名副其实的“大模型”。经过相当于阅读了 369.5 亿个生物序列“单词” 的训练后,该模型学会了提取核酸和蛋白质序列中深层的、通用的模式和规律。
LucaOne的“超能力”展示
研究团队通过一系列精心设计的实验,验证了 LucaOne 的强大能力:
1、无师自通“中心法则”: 最令人惊讶的是,LucaOne 在没有被明确教导 DNA 和蛋白质对应关系的情况下,仅仅通过海量数据的训练,就自发地理解了 DNA 序列与其编码的蛋白质序列之间的内在联系!在判断一段 DNA 序列和一段蛋白质序列是否匹配的任务中,LucaOne 仅需极少量的样本进行微调(Few-shot Learning),其表现就远超其他专门为 DNA 或蛋白质设计的先进模型(例如 DNABert2、ESM2-3B),甚至超过了将这两个模型简单组合使用。这表明,统一训练让 LucaOne 真正“领悟”了生命信息传递的核心规则。
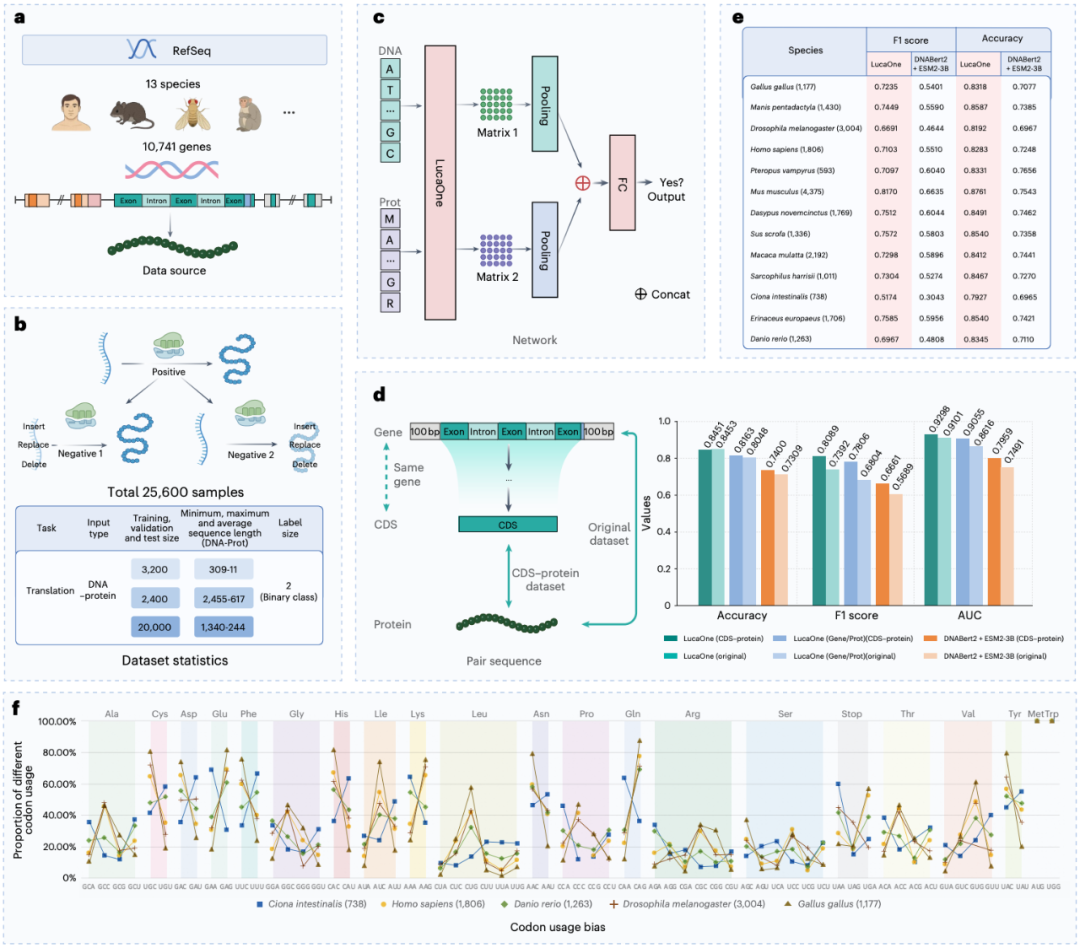 LucaOne 理解了中心法则
LucaOne 理解了中心法则
2、卓越的“嵌入”表示: LucaOne 能将任何输入的 DNA、RNA 或蛋白质序列,转换成一个高维的数学向量(称为“嵌入”或Embedding)。这个向量如同该序列的“数字指纹”,精准地捕捉了其生物学意义。实验证明,LucaOne 生成的嵌入在聚类相似序列(例如同物种 DNA、同功能蛋白质)方面,效果显著优于其他模型。
3、胜任各种生物计算任务的多面手: 利用 LucaOne 生成的嵌入作为起点,研究团队在 7 项具有挑战性的下游生物信息学任务中进行了测试,结果表现非常亮眼:
物种分类(GenusTax): 根据一段 DNA 片段预测它来自哪个物种(属或种级别),准确率大幅提升。
非编码 RNA 家族分类(ncRNAFam): 识别不同类型的非编码 RNA,准确率更高。
蛋白质亚细胞定位(ProtLoc): 预测蛋白质在细胞内的位置(细胞膜或细胞质),表现优异,与 ESM2-3B 模型相当,优于 SOTA 模型。
蛋白质热稳定性预测(ProtStab): 预测蛋白质结构是否稳定,相关性指标领先。
流感病毒抗原性预测(InfA): 基于病毒 RNA 序列对预测其抗原性是否相似,达到近乎完美(100%)的准确率。
蛋白质-蛋白质相互作用(PPI): 判断两个蛋白质是否会相互作用,效果拔尖。
非编码 RNA-蛋白质相互作用(ncRPI): 预测非编码 RNA 与蛋白质的相互作用,表现优于 DNABert2 + ESM2-3B 模型的组合。
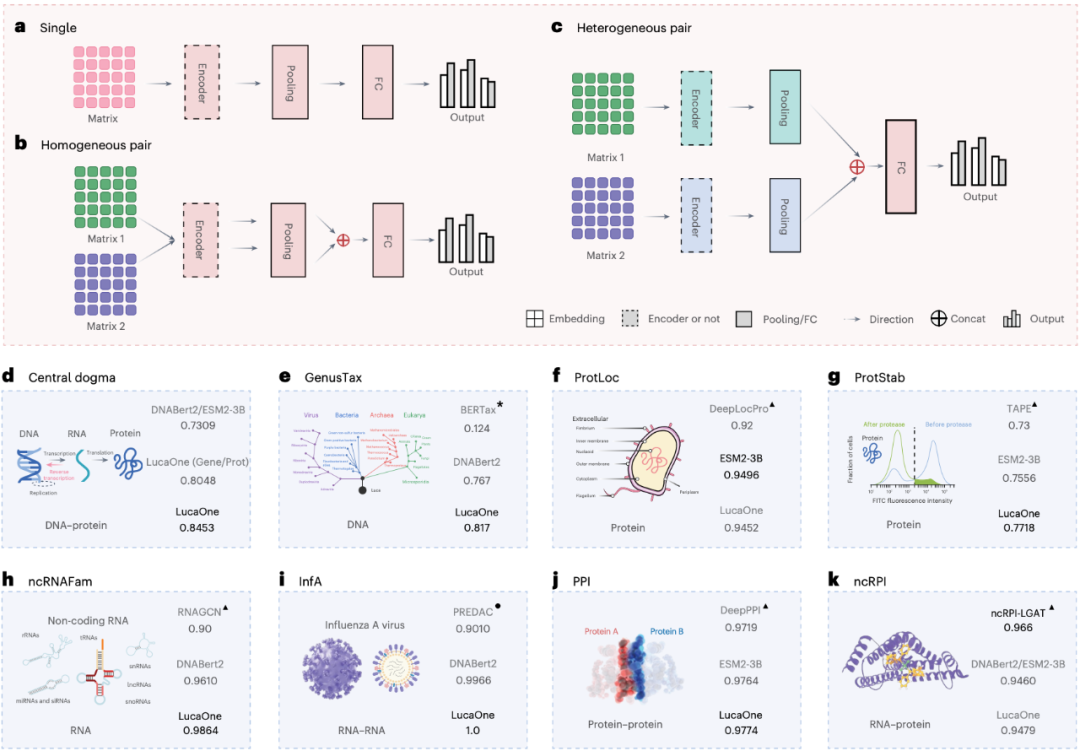 LucaOne 在 7 种任务中的表现
LucaOne 在 7 种任务中的表现
在这 7 个任务中,LucaOne 使用相对简单的下游网络就能取得媲美甚至超越专用复杂模型的效果,大大降低了后续任务开发的复杂度和计算成本。
意义与展望:打开生命密码的新大门
LucaOne 的诞生具有重大意义:
1、统一框架: 它首次为同时理解生命的两大核心分子载体(核酸和蛋白质)提供了一个强大的统一计算框架,打破了分子类型间的壁垒;
2、基础模型赋能: 它证明了“基础模型”范式在生物信息学中的巨大潜力。预训练好的 LucaOne 就像一个强大的“预训练大脑”,研究人员可以基于它提供的“嵌入”,用相对较少的数据快速开发各种特定的生物计算工具(例如疾病诊断、药物靶点发现、合成生物学设计),极大地提高效率;
3、解码生命复杂性: LucaOne 展现出的对“中心法则”等核心生物学原理的自发理解,为未来利用 AI 更深入地、更自动化地解析生命复杂系统(例如基因调控网络、疾病机制)铺平了道路。
当然,挑战依然存在: 作者也在论文也坦诚讨论了 LucaOne 在理解基因组中非编码区域、应对罕见密码子使用偏好物种、整合更多非序列信息(例如表型、环境)以及模型可解释性等方面仍需改进。此外,模型的训练也需要巨大的计算资源。
总的来说,LucaOne 的出现,标志着生物信息学正在进入一个由大型通用基础模型驱动的新时代。它不仅仅是一个强大的工具,更像是一把新打造的、能同时解读核酸和蛋白质这两种“生命语言”的钥匙,为我们开启“孟德尔图书馆”(The Library of Mendel)中更深奥的“书架”提供了可能。尽管前路漫漫,但 LucaOne 已经照亮了方向,让我们对利用 AI 彻底解码生命语言、加速生命科学研究的未来充满期待!
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


