戴俊彪/闫建斌团队通过纳米孔测序,破解模式生物小立碗藓的染色体数目之谜
来源:生物世界 2024-02-02 10:00
该研究首次构建了端粒到端粒无间断的小立碗藓参考基因组,证明了小立碗藓的染色体数目为26条,为基于苔藓模型的演化生物学,基因组学及其它基础研究提供了高质量参考基因组。
中国农业科学院基因组所戴俊彪团队和闫建斌团队合作,在 Nature Plants 期刊发表了题为:Near telomere-to-telomere genome of the model plant Physcomitrium patens 的研究论文。
该研究首次构建了端粒到端粒无间断的小立碗藓参考基因组,证明了小立碗藓的染色体数目为26条,为基于苔藓模型的演化生物学,基因组学及其它基础研究提供了高质量参考基因组。

在这项研究中,戴俊彪团队与闫建斌团队联合研究,借助纳米孔(Nanopore)单分子测序数据,使用NextDenovo组装、Hi-C文库辅助,经GBGF方法(Graph-Based Gap Filling)不断校正后,填补了scaffolds上存在的所有gap,并最终得到了以(GGGCCA)n或(TTTAGGG)n为端粒重复序列的26条染色体(V6)。
经过评估,该组装的单碱基正确率高达99.9989%。该研究还比较了V3和V6版本的同线性和结构变异,并对新版参考基因组进行了从头注释。注释结果表明,与V3版本相比,V6版本消除了许多转座子(TE)区存在的重复注释现象。研究团队已将V3版本注释与新版注释进行了整合,以使其它植物生物学研究者可以方便地将基于V3的研究移植到V6上。
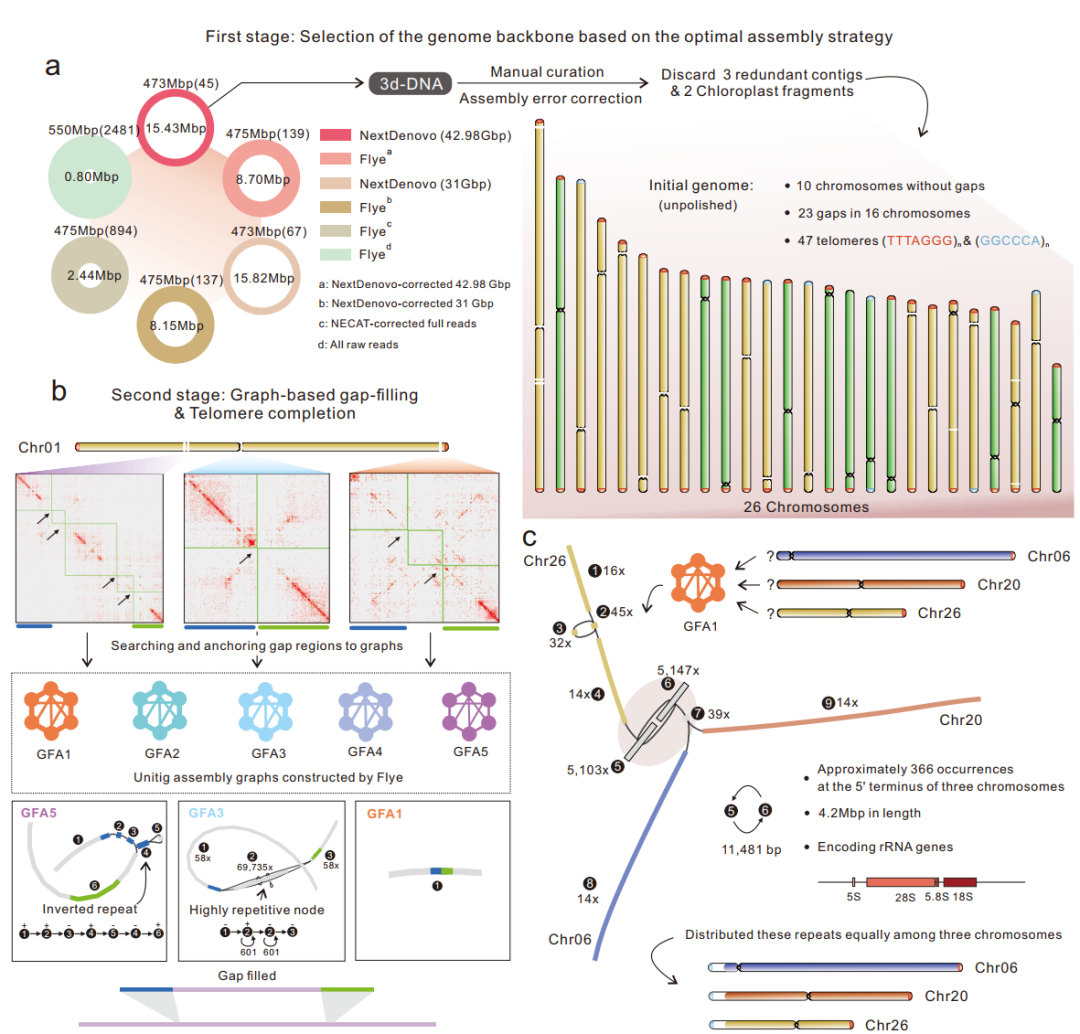
基于GBGF方法的小立碗藓基因组组装流程
该研究基于计算结果和免疫染色验证了V6版本的26条染色体结论,并进一步深入研究了基因组三维结构在发育过程及生命周期中的动态变化,为了解重要基因调控的分子机制提供了新的工具。该研究提供的参考基因组将极大有利于基于苔藓模型的演化生物学,基因组学及其它基础研究。
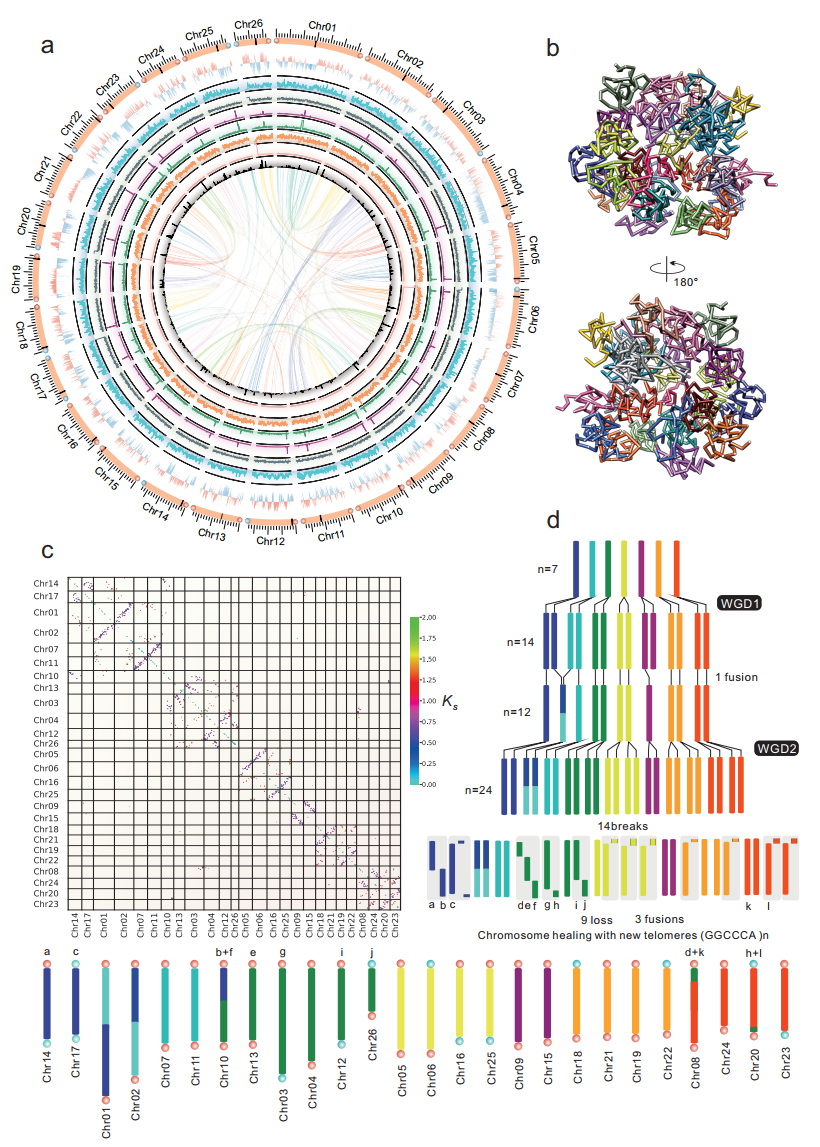
小立碗藓T2T基因组, 3D基因组结构与更正后的核型进化模型
闫建斌课题组博士后毕桂萁,戴俊彪课题组博士研究生赵世俊,闫建斌课题组博士研究生姚嘉伟、博士研究生汪欢,为该论文的共同第一作者。基因组所戴俊彪研究员和闫建斌研究员为该论文的共同通讯作者。该研究得到了国家重点研发计划、国家自然科学基金委员会、中国科学院、中国农业科学院和深圳市等多个项目的资助。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


