西湖大学曾坚阳团队开发自监督学习框架KPGT,为AI制药提供新工具
来源:生物世界 2023-11-24 17:14
总的来说,KPGT为有效的分子表示学习提供了一种强大的自监督学习框架,从而推进人工智能辅助药物发现(AIDD)领域的发展。
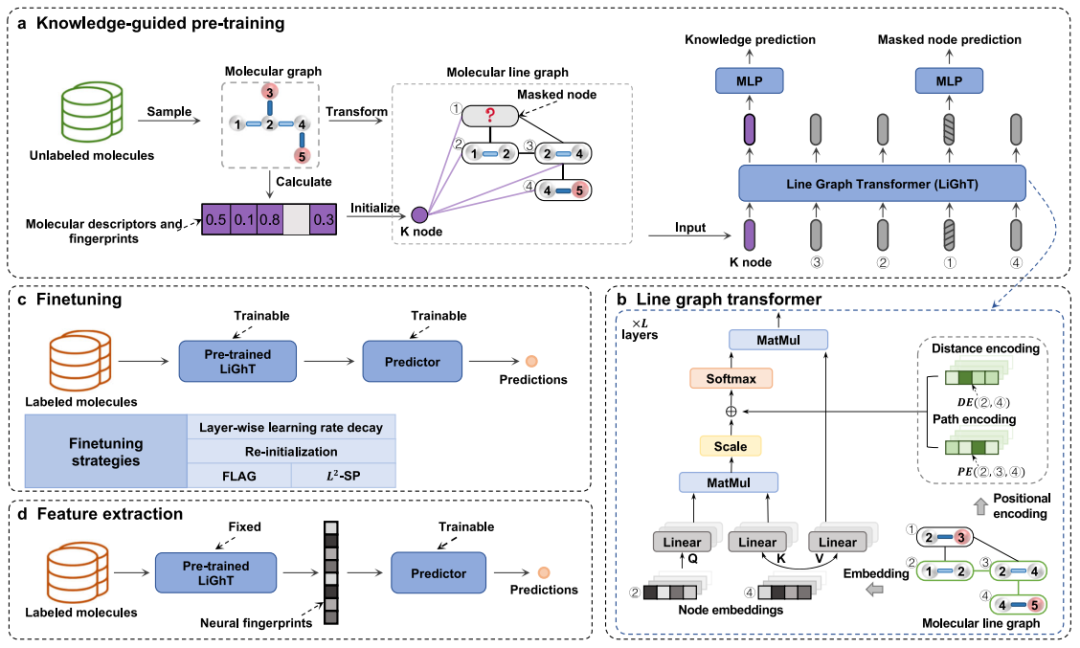
该研究介绍了一种增强分子表示学习的自监督学习框架——KPGT(Knowledge-guided Pre-training of Graph Transformer),在预测不同领域的分子特性方面表现出优异的性能。研究团队使用KPGT成功识别了两个抗肿瘤靶点HPK1和FGFR1的潜在抑制剂,验证了KPGT在药物发现中的实际应用。
这些结果表明了KPGT可以为推进人工智能辅助药物发现(AIDD)过程提供一个强大而有用的工具。

近年来,基于深度学习的方法作为预测分子特性的潜在有用工具出现,主要原因是它们具有从简单输入数据自动提取有效特征的卓越能力。例如包括循环神经网络(RNN)、卷积神经网络(CNN)和图神经网络(GNN)在内的各种神经网络架构。然而,标记分子的有限可用性和化学空间的无穷限制了它们的预测性能,特别是在处理离散数据样本时。随着自监督学习方法在自然语言处理和计算机视觉领域的显著成就,这些技术已被用于预训练GNN和提高分子表示学习,从而在下游分子特性预测任务中取得了实质性的改进。
现有的自监督学习方法通常依赖于GNN作为骨架模型。但GNN只能提供有限的模型容量,因为当增加层数(深度)时,它们会受到过平滑的影响。此外,GNN可能难以捕捉原子之间的远程交互。最新的Transformer模型,正在成为游戏规则的改变者,这些模型的特点是参数数量不断增加,并具有捕捉远程交互的能力,为全面模拟分子的结构特征提供了有希望的途径。
在这项研究中,研究团队介绍了一种增强分子表示学习的自监督学习框架——KPGT(Knowledge-guided Pre-training of Graph Transformer),以推进下游分子属性预测任务。
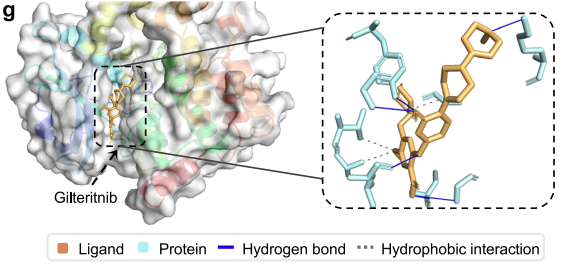
研究团队利用KPGT成功识别了两个抗肿瘤靶点——造血祖细胞激酶1(HPK1)和成纤维细胞生长因子受体(FGFR1)的潜在抑制剂,展示了KPGT在药物发现中实际应用。
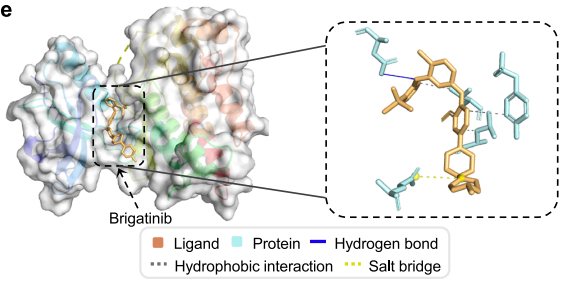
KPGT识别的FGFR1的抑制剂
总的来说,KPGT为有效的分子表示学习提供了一种强大的自监督学习框架,从而推进人工智能辅助药物发现(AIDD)领域的发展。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


