Nature子刊:清华大学蓝勋/李寅青/朱明团队开发通用型基因编辑脱靶检测技术
来源:生物世界 2024-07-05 09:13
相较于现有的GUIDE-seq和DISCOVER-seq等脱靶检测技术,Tracking-seq不仅展现了高召回率,而且具备很高的检测精确度。
清华大学医学院蓝勋团队与药学院李寅青团队合作,在 Nature Biotechnology 期刊上发表了题为:Tracking-seq reveals the heterogeneity of off-target effects in CRISPR/Cas9-mediated genome editing 的研究论文。
该研究报道了一项名为Tracking-seq的新型脱靶效应检测技术,并借助该技术揭示了不同细胞类型、不同编辑工具间存在的脱靶效应异质性。该研究将为基因编辑在基因治疗等领域的安全应用提供重要理论参考及技术支持。

目前的主流基因编辑器均依赖于DNA损伤后的修复过程实现基因编辑,复制蛋白A(replication protein A,RPA)广泛存在于基因组双链及单链损伤修复过程,Tracking-seq通过CUT&RUN技术特异性地识别并捕获与RPA结合的单链DNA(Single-stranded DNA,ssDNA),随后利用链特异性核酸酶消化双链DNA以提高信噪比,构建链特异性ssDNA文库进行高通量测序,并通过新开发的算法精确计算RPA信号强度,进而实现对多种编辑工具的脱靶检测。
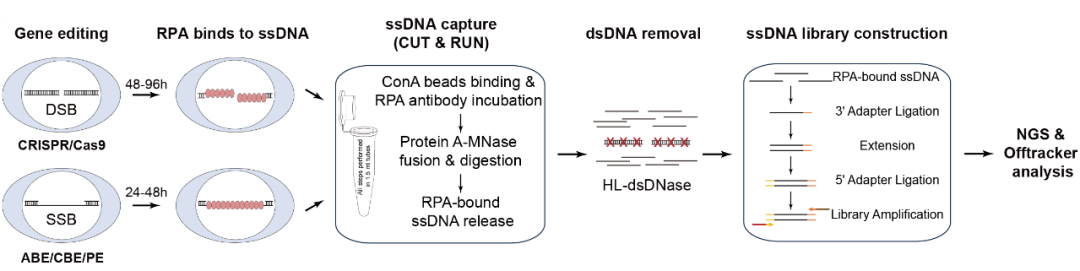
图1. Tracking-seq示意图
相较于现有的GUIDE-seq和DISCOVER-seq等脱靶检测技术,Tracking-seq不仅展现了高召回率,而且具备很高的检测精确度。重要的是,其具有高度的泛用性,使其能够适用于Cas9、CBE、ABE以及PE等各类主流基因编辑工具,并能适应不同的细胞类型及不同的编辑环境(包括体外、离体及体内编辑)。此外,Tracking-seq的高灵敏度使其能够在仅有数千个细胞的样品中,有效地进行脱靶检测,并保持优异的结果。
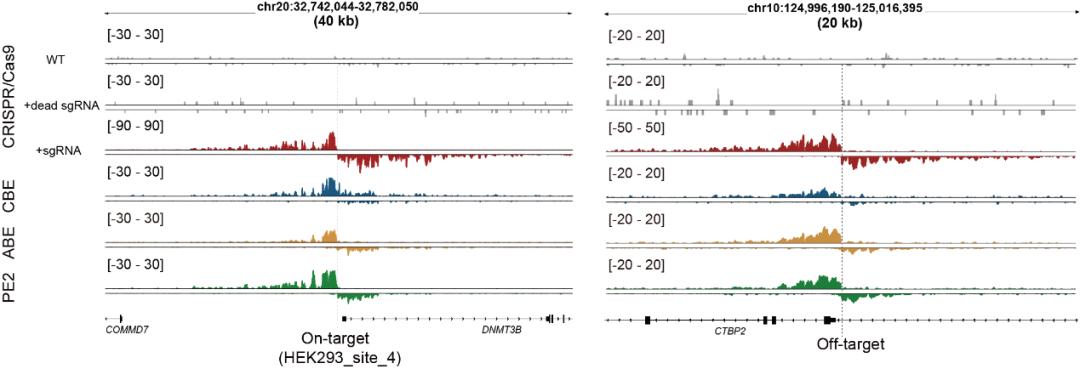
图2. Tracking-seq检测不同编辑工具的链特异性信号
得益于Tracking-seq的高泛用性,该研究发现当使用相同的向导RNA时,Cas9、CBE、ABE和PE的脱靶效应存在明显差异,其中Cas9和CBE之间观测到了许多近似于各自特异的脱靶位点。此外,不同类型的细胞中也观察到了脱靶效应的异质性,脱靶事件似乎更容易在染色质开放和活跃的区域发生。这些脱靶效应的异质性提示,使用替代性编辑系统或在不同细胞类型中进行的脱靶检测,可能无法准确反映目标细胞中实际的脱靶情况。相比之下,Tracking-seq技术能够直接对原始编辑系统进行脱靶检测,因此具有广阔的应用潜力和前景。
清华大学蓝勋副教授、李寅青副教授以及朱明助理研究员(博士后)为论文共同通讯作者。清华大学医学院朱明博士、博士生许润达、药学院博士生袁俊松和生命学院博士生王嘉程为共同第一作者。此外,该研究还得到了清华大学杜娟娟研究员团队、林欣教授团队、颉伟教授团队,烟台毓璜顶医院林春华教授团队,桂林医学院于鸿浩教授团队的重要帮助。同时感谢清华大学祁海教授团队及中国科学院动物研究所魏妥研究员团队的帮助。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


