生物学专属ChatGPT来了:对话式AI智能体——ChatNT,能够理解DNA、RNA和蛋白质语言
来源:生物世界 2025-06-30 11:32
名为 ChatNT的多模态对话智能体,能像生物学家一样,“读懂” DNA、RNA 和蛋白质的序列信息,并用自然语言(英语)与你对话,直接回答你关于生物分子的各种专业问题。
2022 年底,ChatGPT 横空出世,这个能够学习并理解人类自然语言的 AI 聊天机器人震惊了全世界,并掀起了大语言模型(LLM)浪潮。
而现在,人工智能公司 InstaDeep 将这种 AI 的这一强大能力带到了生命科学领域,打造了一款名为 ChatNT(Chat Nucleotide Transformer)的多模态对话智能体,能像生物学家一样,“读懂” DNA、RNA 和蛋白质的序列信息,并用自然语言(英语)与你对话,直接回答你关于这些生物分子的各种专业问题。
该研究以:A multimodal conversational agent for DNA, RNA and protein tasks 为题,于 2025 年 6 月 6 日 发表在了 Nature 子刊 Nature Machine Intelligence 上,该论文的作者还包括来自 mRNA 疫苗巨头 BioNTech 的研究人员。
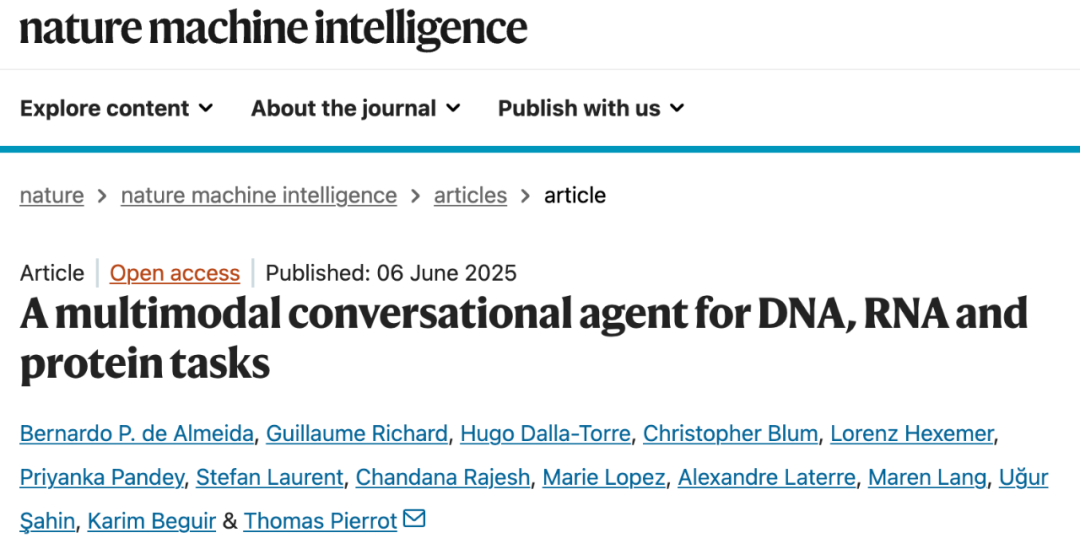
生物学研究的痛点:模型太多、门槛太高
在基因组学、转录组学和蛋白质组学研究中,科学家们常常需要预测特定 DNA 序列的功能(比如是不是启动子、有没有增强子活性)、RNA 的稳定性,或者蛋白质的特性。传统的方法是针对每一个具体任务,训练一个专门的深度神经网络模型。
痛点1、“模型海”:任务成千上万,每个任务都要单独训练和维护一个模型?这不仅效率低下,也阻碍了不同任务间知识的共享和迁移。
痛点2、“编程墙”:这些专业模型通常需要编程技能(例如 Python)才能使用和分析结果。对于广大没有计算机背景的生物学家来说,这无疑是一道高墙,限制了先进技术的普及和应用。
ChatNT:一个模型,自然对话,通吃多任务
ChatNT 的诞生,就是为了解决这两个核心痛点。研究团队创造性地将两类强大的 AI 模型“合体”:
1、DNA 编码器(DNA Encoder):核心是 Nucleotide Transformer v2 模型(一个拥有 5 亿参数、在 850 个物种基因组上预训练过的 DNA 大语言模型)。它能深度理解 DNA 序列中蕴含的复杂模式和特征。
2、英语解码器(English Decoder):使用的是经过指令微调的 Vicuna-7B 模型(一个基于 LLaMA 的70亿参数大语言模型)。它擅长理解人类自然语言指令并生成自然流畅的回复。
在二者之间,研究团队实现了关键的一步——投影层(Projection Layer),通过设计的英语感知投影(English-aware Projection),将 DNA 编码器理解的序列信息转化为英语解码器能够处理的格式,更重要的是,它能根据使用英语提出的问题,动态地从 DNA 信息中筛选和提炼出最相关的部分,这就像给信息流加了一个“智能滤网”,极大地提升了信息传递的效率和针对性。
ChatNT的工作原理简单来说就是:
1、用户输入问题(使用英语),并在问题中用特殊标记指代提供的 DNA/RNA/蛋白质序列文件。
2、DNA 编码器分析序列,生成深度特征。
3、英语感知投影接收这些特征和用户的问题,动态提取与问题最相关的信息,转换成英语解码器能理解的格式。
4、英语解码器结合问题和转换后的序列信息,生成自然语言答案。
5、答案返回给用户,直接回答提出的问题。
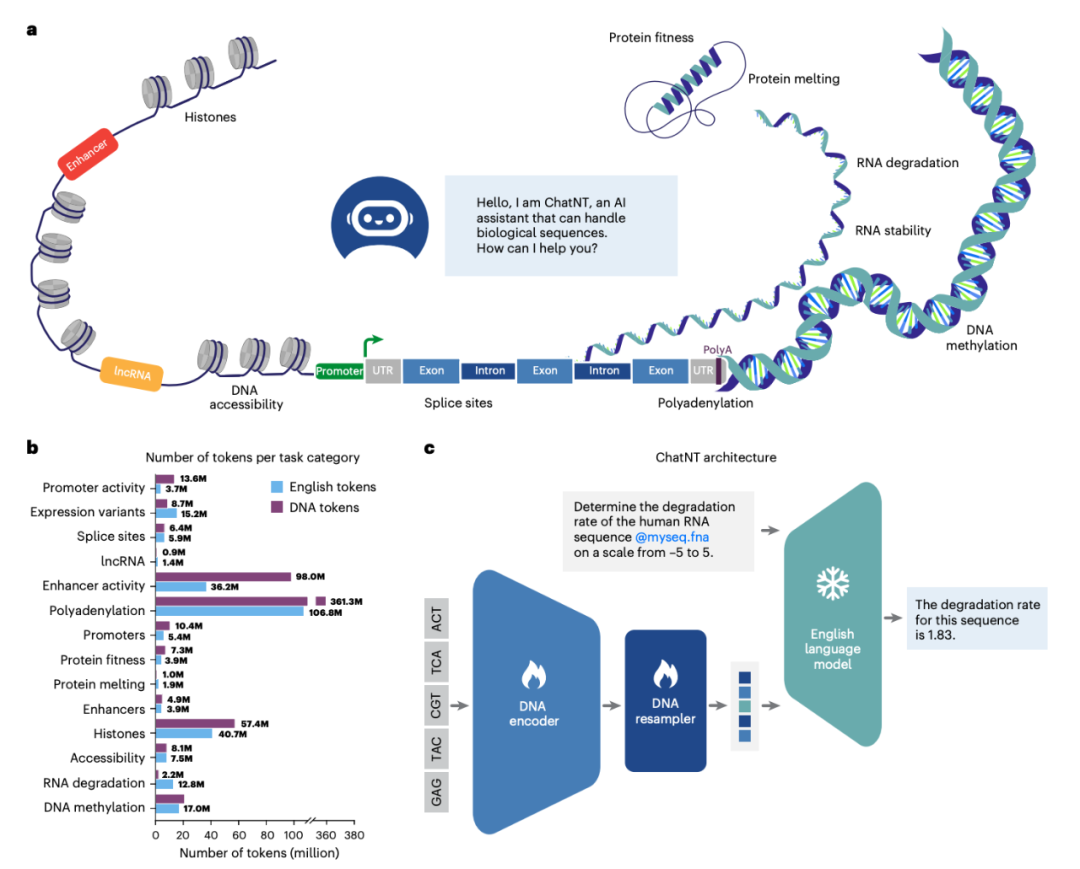
强大表现:媲美专用模型,还能聊天
ChatNT 可不是花架子,它在多项严格的生物信息学基准测试中展现了惊人的实力:
1、刷新纪录:在权威的“Nucleotide Transformer Benchmark”(包含 18 项基因组学任务)上,ChatNT 的平均马修斯相关系数(MCC)达到了 0.77,比之前最好的专用模型(Nucleotide Transformer v2)提高了 8 个百分点,创造了新的 State-of-the-Art(SOTA)。
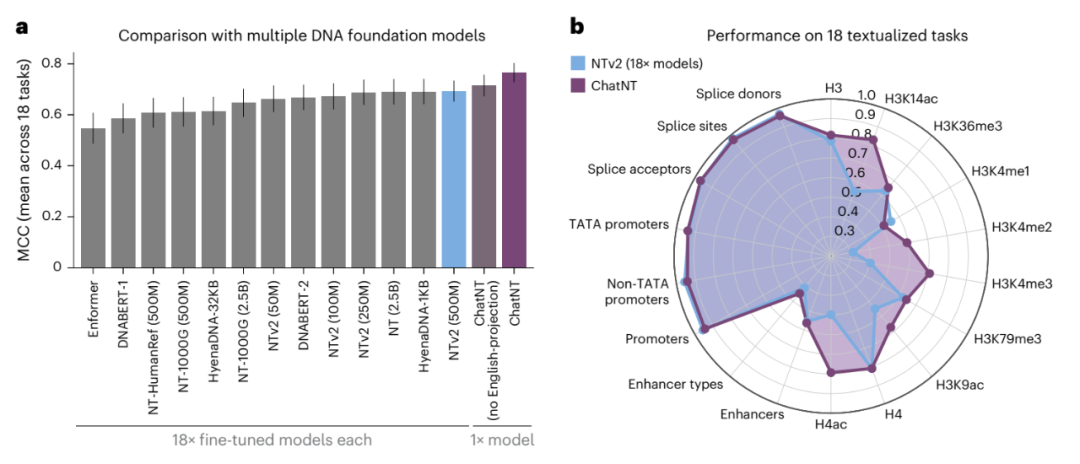
2、全能选手,一个顶N个:ChatNT 的突破性在于,它是一个统一的模型,能同时处理这 18 项完全不同的任务(例如预测启动子、识别甲基化位点、判断染色质开放性等)。用户只需去“问”它即可。这解决了传统方法需要“一任务一模型”的困境。
3、挑战复杂生物任务,不输专家:研究团队进一步构建了一个包含 27 项更具生物学意义任务的“基因组指令数据集”(涵盖DNA、RNA、蛋白质)。ChatNT 在其中大多数任务上表现优异,性能与当前领域内顶尖的专用模型不相上下。例如:
- 在人类启动子活性、增强子类型预测等 DNA 任务上表现出色;
- 在预测 RNA 多聚腺苷酸化位点比例的任务上,皮尔逊相关系数(PCC)达 0.91,略优于专用模型 APARENT2(0.90)。
- 在预测蛋白质熔点的任务上,PCC 达 0.89,优于蛋白质大模型 ESM2(0.85)。
- 虽然在 RNA 降解任务上略逊于专用模型 Saluki,但研究团队指出,未来整合 RNA 专用编码器将能弥补这一差距。
4、理解生物“语法”:利用模型解释技术,研究团队发现,ChatNT 在回答问题时,其决策依据与已知的关键生物学特征高度吻合。例如,在识别剪接供体位点时,它重点关注“GT”二核苷酸;在识别启动子时,它关注“TATA-box”基序。这表明它真正学习到了 DNA 序列中蕴含的生物学规则。
5、尝试“校准”信心:为了解决大模型生成答案时可能“信口开河”的问题,研究团队探索了一种基于“困惑度(Perplexity)”的方法来评估 ChatNT 在二元分类任务(“是/否”)上的回答置信度。初步结果显示该方法有效,未来可整合到工具中,帮助用户判断答案的可靠性。
意义与未来:生物学研究的“对话革命”
ChatNT 的出现,标志着生物学 AI 研究进入了一个新阶段:
- “对话式”生物信息学:最大的变革在于交互方式。生物学家无需编程,只需像提问同事一样,用英语描述问题并提供序列数据,ChatNT 就能直接给出答案或分析结果。这大大降低了先进 AI 工具的使用门槛。
- 统一、通用的生物大模型雏形:ChatNT 证明了用一个统一模型处理多种不同类型生物序列(DNA/RNA/蛋白)任务的可行性,朝着构建真正的“通用型生物学 AI 模型”迈出了重要一步。
- 知识迁移与零样本潜力:通过自然语言统一任务框架,模型在不同任务间学习到的知识更容易相互迁移,也为未来实现“零样本”(Zero-shot,无需额外训练数据)解决新任务奠定了基础(例如,用户直接问一个训练数据中未明确包含的问题)。
- 无限扩展可能:其模块化架构允许轻松集成更强大的 DNA/RNA/蛋白质编码器(如处理超长序列的 Borzoi 模型,或更强大的RNA/蛋白大模型)以及更强大的对话模型(例如 Llama 2-Chat)。未来可扩展到整合更多模态,例如结构信息(蛋白质三维结构)、组学数据、甚至医学影像,构建更全面的生物医学智能体。
- 解读基因突变的新途径:未来用户可能只需提供“野生型”和“突变型”序列文件,然后问 ChatNT:“这个突变会导致疾病吗?”,它就能基于对序列的理解给出分析预测。
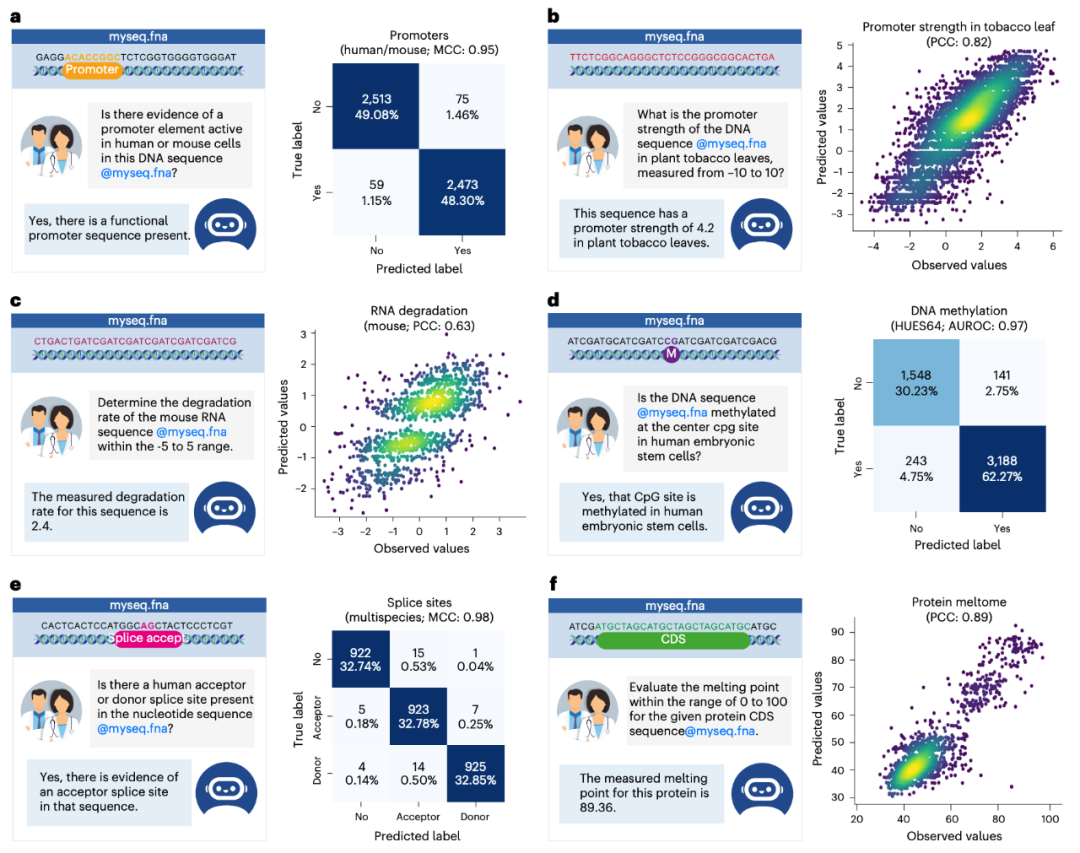
总的来说,ChatNT 并非只是一个“噱头”。它通过创新的多模态架构(结合 DNA 编码器、英语感知投影和英语解码器),首次实现了用自然语言对话的方式,让 AI 理解和分析复杂的 DNA、RNA 和蛋白质序列信息。它在多项基准测试中达到或逼近最先进水平,一个模型解决多个任务,并能揭示生物学相关的序列特征。
这项研究为生物学研究提供了一种革命性的交互范式。想象一下,不久的将来,只需对着 AI 助手描述:“帮我分析这段患者 DNA 序列,看看这个位置的突变会不会影响附近那个基因的表达,特别是在肝细胞里”,然后,ChatNT(或其进化版)就能给出专业的、可理解的回答。这无疑将极大加速生命科学的探索进程,让生物信息学分析变得更加直观、高效和普及。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


