Frontiers in Plant Science:大豆突变体基因定位测序方法改良研究获进展
来源:东北地理所 2021-06-07 20:24
随着新一代测序技术的发展和全基因组测序(Whole Genome Sequencing, WGS)成本的不断下降,基于WGS的分离群体分组分析(Bulk Segregation Analysis,BSA)已成为快速定位候选基因的常规工具,但已报道的方法仍有待完善。例如,依赖于自交系杂交的QTL-seq策略需要后期耗费大量精力进行精细定位;而基于突变体的Mut
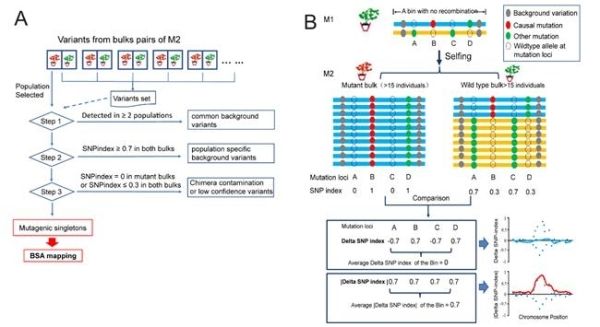
随着新一代测序技术的发展和全基因组测序(Whole Genome Sequencing, WGS)成本的不断下降,基于WGS的分离群体分组分析(Bulk Segregation Analysis,BSA)已成为快速定位候选基因的常规工具,但已报道的方法仍有待完善。例如,依赖于自交系杂交的QTL-seq策略需要后期耗费大量精力进行精细定位;而基于突变体的Mutmap策略,虽然不需要后期进行精细定位,但仍需耗费大量时间用于突变体的自交与回交。这些方法存在的不足限制了大规模开展功能基因定位的效率。
中国科学院东北地理与农业生态研究所大豆分子设计育种重点实验室研究员冯献忠团队围绕大豆突变体功能基因定位,报道了一种称为M2-seq的改良版WGS-BSA方法。它是一种快速有效的突变基因定位工具,仅基于M2代材料即可实现候选因果突变位点的快速定位,与此前报道的需要更高世代自交与回交的方法(如Mutmap)相比,在时间成本和测序费用成本上均更具有优势。利用M2-seq方法,在不知道突变植株的野生型变异信息情况下,通过M2群体之间的相互比较,背景变异即可被有效去除。此外,利用ΔSNP-index的绝对值可有效去除由相邻突变等位基因的排斥连锁引起的突变频率信号波动,从而有助于定位靶基因中的因果突变。该研究展示了M2-seq在10个独立的大豆M2突变体群体中成功定位因果突变。研究表明,基于M2世代的M2-seq方法可加速基因定位,尤其是在世代间隔较长的植物物种中。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
87%用户都在用生物谷APP 随时阅读、评论、分享交流 请扫描二维码下载->


