AI仍需努力!徐华强团队证实,AlphaFold3预测GPCR-配体复合物结构准确性不足
来源:生物世界 2024-12-13 14:47
该研究为在GPCR研究中使用AlphaFold3的研究人员提供了有价值的见解,强调了基于实验的结构确定的必要性,并为未来在计算模型中改进蛋白质-配体相互作用预测提供了方向。
2024年5月9日,谷歌DeepMind团队在 Nature 期刊发表论文【1】,推出了AlphaFold2的全面升级版——AlphaFold3。
相比AlphaFold2对单个蛋白质结构的精准预测,AlphaFold3能够高准确性预测蛋白质与各种生物分子相互作用的结构。这一最新模型能够预测含有蛋白质数据库内几乎所有分子类型的复合物的结构,包括配体(小分子)、蛋白质、核酸(DNA和RNA)如何聚集在一起并相互作用,以及预测翻译后修饰和离子对这些分子系统的结构影响,从而帮助我们在原子水平上精确地观察生物分子系统的结构。
2024年11月11日,DeepMind宣布将AlphaFold3正式开源,科学家们可以下载其底层代码,并将其应用于非商业领域。
中国科学院上海药物所徐华强团队(博士生何欣恒、助理研究员李俊睿、博士生沈诗怡为共同第一作者)在Acta Pharmacol Sin期刊发表了题为:AlphaFold3 versus experimental structures: assessment of the accuracy in ligand-bound G protein-coupled receptors 的研究论文【2】。
该研究全面评估了AlphaFold3预测配体结合G蛋白偶联受体(GPCR-配体复合物)的结构的准确性,包括整体结构的准确性、口袋预测能力、复合物预测能力和可变区预测效果,结果显示,AlphaFold3相比AlphaFold2有所提升,但预测结构与实验确定结构相比仍然存在很多差距。
这项研究强调了即使在用于了AI结构预测工具的情况下,通过实验确定结构仍是必不可少的,这项研究也为未来在计算模型中改进蛋白质-配体相互作用预测提供了方向。

G蛋白偶联受体(GPCR)是重要的药物靶点,参与许多生理过程,但由于其固有的灵活性以及与不同的配体相互作用,许多结构仍未解决。
在这项最新研究中,许华强团队系统地评估了AlphaFold3预测的GPCR结构与通过实验确定的结构相比的准确性,主要关注GPCR-配体结合状态。
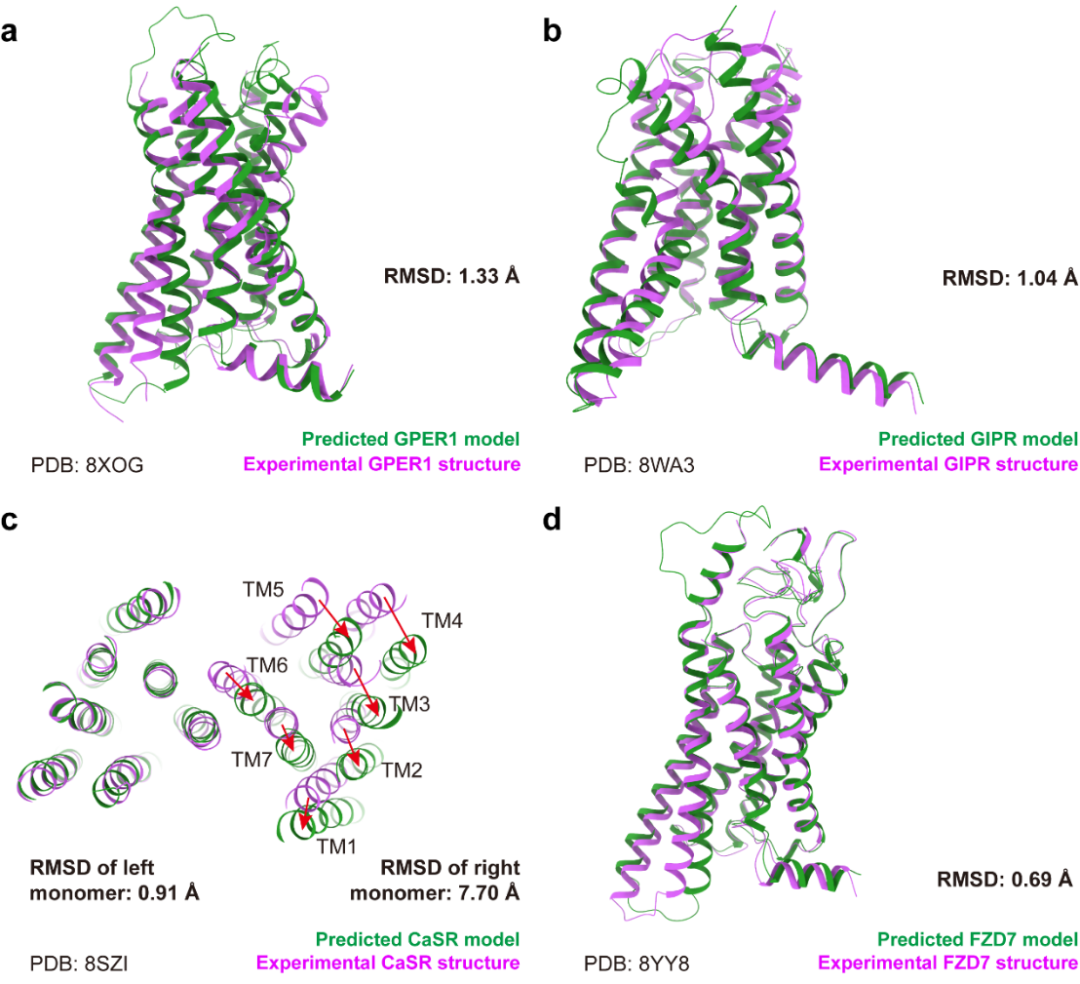
AlphaFold3预测结构和实验结构的整体对比
结果显示,虽然AlphaFold3在预测整体GPCR骨架结构方面比AlphaFold2表现出更好的性能,但在配体(尤其是离子、多肽和蛋白质)结合方面,预测结果与实验结果之间仍然存在显著差异。该研究还评估了预测结构在各种配体类型中的准确性,量化了结合口袋几何形状和配体方向的偏差。这些发现强调了在配体结合GPCR结构的计算预测方面存在的特殊挑战。
这些局限性限制了AlphaFold3在GPCR的功能研究和基于结构的药物设计中的应用,因为无论功能研究还是药物设计,GPCR-配体相互作用的高分辨率细节至关重要。
总的来说,该研究为在GPCR研究中使用AlphaFold3的研究人员提供了有价值的见解,强调了基于实验的结构确定的必要性,并为未来在计算模型中改进蛋白质-配体相互作用预测提供了方向。
论文链接:
1. https://www.nature.com/articles/s41586-024-07487-w
2. https://www.nature.com/articles/s41401-024-01429-y
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


