Nature Methods:开发胞嘧啶碱基编辑器脱靶效应检测的新工具
来源:北京大学生命科学学院 2021-06-20 13:12
北大-清华生命科学联合中心、北京大学生命科学学院伊成器教授课题组在Nature Methods杂志发表了题为“Detect-seq reveals out-of-protospacer editing and target-strand editing by cytosine base editors”的封面文章。研究人员在本研究工作的主要贡献如下:建立了一
北大-清华生命科学联合中心、北京大学生命科学学院伊成器教授课题组在Nature Methods杂志发表了题为“Detect-seq reveals out-of-protospacer editing and target-strand editing by cytosine base editors”的封面文章。研究人员在本研究工作的主要贡献如下:
建立了一种无偏向性的脱靶检测技术
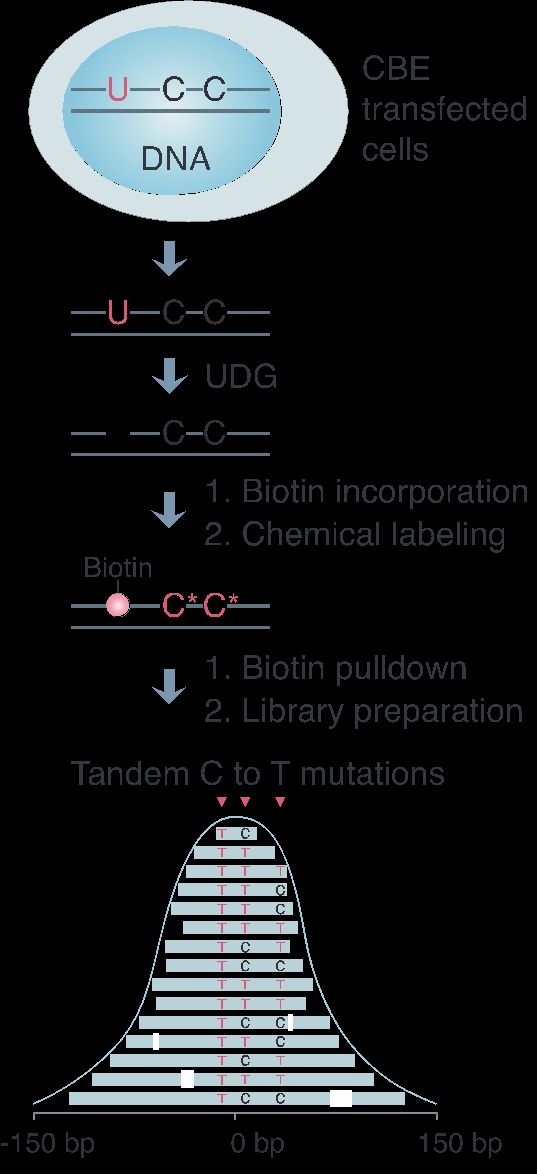
作者通过捕获胞嘧啶碱基编辑器CBE[1]产生的中间体(dU),并进行一系列标记和富集[2, 3],获得具有连续C-to-T突变的脱靶信号,并将其命名为Detect-seq(dU-detection enabled by C to T transition during sequencing)技术,该技术的主要技术路线如图1所示。
研究人员在人胚肾细胞HEK293T和人乳腺癌细胞MCF-7转染BE4max[4],使用多种不同的sgRNA对基因组靶向位点进行C-to-T的编辑;同时对这些样本中进行了Detect-seq检测。通过Detect-seq检测发现,除RNF2位点外,其它靶向位点均可以产生几十至数百个Cas9依赖型的脱靶位点(图2)。
发现了两种新型脱靶位点(out-of-protospacer edit和target-strand edit)
有趣的是,研究人员基于Detect-seq技术还发现了两种新型的Cas依赖型的脱靶编辑:sgRNA结合区域外编辑(out-of-protospacer edit)及靶向链编辑(target-strand edit)(图3)。sgRNA结合区域外编辑即C-to-T的编辑发生在sgRNA结合区域外;而靶向链编辑则是C-to-T编辑发生在sgRNA与DNA结合链。根据Detect-seq的结果,上述两种编辑普遍存在于Cas9依赖型的脱靶位点附近。
综上所述,Detect-seq技术为碱基编辑领域内提供了一种能够高灵敏、高特异、无偏好性地检测细胞内的CBE脱靶位点的方法。脱靶效应理解的加深,也将为阐明CBE的催化机制、优化改造更安全的CBE工具带来新的视角与可能。(生物谷Bioon.com)
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。
87%用户都在用生物谷APP 随时阅读、评论、分享交流 请扫描二维码下载->


