新型冠状病毒基因组的演化分析及谱系划分取得进展
来源:北京大学 2021-03-26 12:46
北京大学生命科学学院陆剑课题组与中国科学院昆明动物所吕雪梅、张亚平课题组,中国疾病预防控制中心谭文杰课题组合作在《科学通报》上发表了题为“Evolutionary analysis and lineage designation of SARS-CoV-2 genomes”的论文,解析了新型冠状病毒(以下简称新冠病毒)基因组的演化规律
北京大学生命科学学院陆剑课题组与中国科学院昆明动物所吕雪梅、张亚平课题组,中国疾病预防控制中心谭文杰课题组合作在《科学通报》上发表了题为“Evolutionary analysis and lineage designation of SARS-CoV-2 genomes”的论文,解析了新型冠状病毒(以下简称新冠病毒)基因组的演化规律,提出了具有层次结构的谱系/亚谱系划分系统。
病毒的演化极其迅速,随着感染人数爆发性增加,新冠病毒会快速发展出许多不同的株系。探明病毒的谱系发生历史,对不同的毒株进行分型,结合流行病学及临床数据揭示新冠病毒的变异规律,对疫情的防控尤为重要。在研究团队前期的工作中,已根据两个高度连锁的位点将新冠病毒划分为L和S两个主要谱系,并描述了这两个谱系在传播过程中不同的行为模式。国际上也已发表诸多研究为新冠病毒进行分型。然而,随着新冠病毒基因组序列的迅速积累,人们又检测到并发表了成千上万的基因组变异,出现了许多值得进一步标记的亚型,大量相似的病毒序列对SARS-CoV-2基因组系统发育的可靠推断提出了重大挑战,亟需研究对这些新出现的变异进行梳理与描述,更加精细化地展示新冠病毒在人群中的演变轨迹。
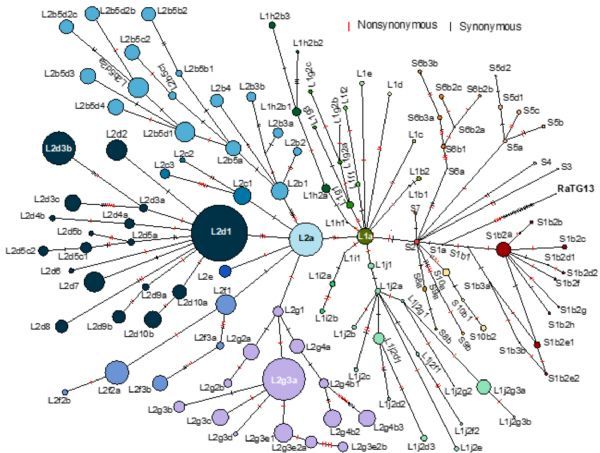
为解决这一问题,研究团队分析了121,618个高质量的病毒基因组中的单核苷酸变异(SNV),在L和S分型的基础上,根据3个主导性SNV将L谱系划分为L1和L2两个主要亚谱系,再根据位点的突变频率和连锁性筛选出201个代表性SNV,进一步逐级划分出130个亚谱系(S中37个,L1中35个,L2中58个),绘制成了完整的反映各个谱系之间亲缘关系的单倍型网络图(图1)。作者同时建立了实时更新的配套网站方便查看亚谱系信息,此外用户还可自由上传新冠病毒基因组序列进行谱系鉴定。
为阐明不同亚谱系的病毒的流行程度是否有所不同,作者依据上述谱系划分系统对有具体采样时间信息的119,168个高质量病毒基因组进行划分,并挖掘了这些谱系的时空分布演变规律。研究发现,各个谱系在不同地域上的传播表现十分不同。例如,在欧洲(n=71,120),大多数病毒属于L谱系(70,434,99.0%),其中L2d(26,206,36.8%)和L2b(19,416,27.3%)是两个最大的亚谱系,而在亚洲(n=8066),S谱系占到了7.1%(576),L谱系最初以L2d(2381,29.5%)为主,但随后L2g的频率显着增加。其余大洲各个谱系出现的频率也不尽相同。(图2)这表明,在地理条件、交通运输、文化风俗、防疫政策、超级传播者等各种因素共同作用下,病毒谱系在不同地域之间有各自独特的分布模式,这就为病例的分子溯源提供了理论依据。(生物谷Bioon.com)
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。