研究揭示活跃转座子识别及表观沉默机制
来源:分子植物卓越中心 2021-03-08 12:15
Nature Plants在线发表了中国科学院分子植物科学卓越创新中心、中科院-英国约翰英纳斯中心植物和微生物科学联合研究中心Jungnam Cho研究组题为Ribosome stalling and SGS3 phase separation prime the epigenetic silencing of trans
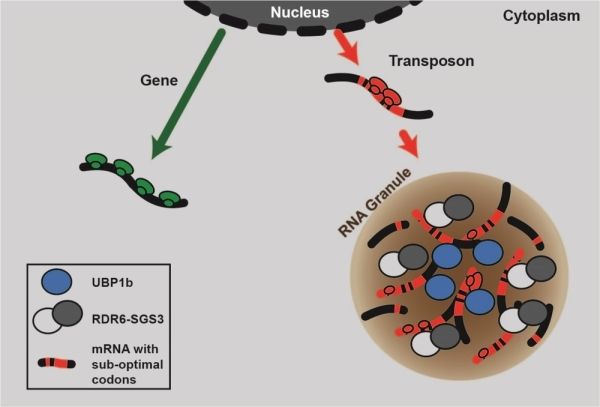
Nature Plants在线发表了中国科学院分子植物科学卓越创新中心、中科院-英国约翰英纳斯中心植物和微生物科学联合研究中心Jungnam Cho研究组题为Ribosome stalling and SGS3 phase separation prime the epigenetic silencing of transposons的研究论文,该研究从全新的角度阐述了宿主识别细胞中活跃转座子并诱导其沉默的机制。
转座子(TEs)是基因组中可移动的DNA元件,其移动可导致致命的突变,为了抑制其活性,宿主基因组进化出了利用干扰小RNA(siRNA)触发并维持转座子表观沉默的机制。由RNA聚合酶Pol IV和Pol V介导的DNA甲基化RdDM途径作用于已含有甲基化修饰的转座子,以增强TE的沉默状态。而激活的或未被甲基化修饰的转座子则通过RNA依赖的RNA聚合酶6(RDR6)介导产生21-22 nt的siRNA,触发RNAi和从头开始的DNA甲基化,这一过程称为RDR6-RdDM。RDR6-RdDM对于引发活跃转座子的沉默十分重要,但RDR6如何特异识别转座子RNA并选择性加工siRNA未知。此前研究表明,mRNA的初始切割是RDR6识别的关键先决条件,且siRNA的产生在细胞空间上局限于RDR6和SGS3共定位的siRNA小体中。研究表明,植物转座子RNA由于含有非最优密码子导致翻译过程中出现常见的核糖体停滞,核糖体停滞随后诱发RNA截短并定位到细胞质siRNA小体中。此外,SGS3通过其朊蛋白样结构域在体内外均表现出相分离现象,显示了液-液相分离在siRNA小体形成中的关键作用。(生物谷Bioon.com)
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


